平台介绍¶
校级高性能计算平台,由 arm 和 x86 两类芯片架构的CPU节点组成,作业调度系统采用的多瑙(Donau Scheduler)。
使用指南¶
-
在满足本实验室正常使用的情况下,面向全校师生开放使用,每学年4次集中考核、申请;收费标准
-
禁止在登录节点运行作业,登录节点运行的程序使用CPU或内存超标会被监控程序杀掉。所有作业应由多瑙作业调度系统调度至计算节点运行,多瑙使用文档见 多瑙使用。
-
多瑙作业资源申请规则及注意事项:
- 多瑙作业申请资源时需遵守资源申请的基本规则,即申请的CPU核心数和程序使用的线程数相等,避免集群资源的浪费或者节点负载过高,如果违反规则导致资源大规模浪费,账号将会被暂停提交作业一段时间或降低可使用的核心数;
-
本集群拥有用 ARM 和 X86 两类架构的 CPU,其上运行的软件有差异,即在 ARM 上编译的软件无法在 X86 节点运行、在 X86 上编译的软件无法在 ARM 节点运行。鉴于此,建议用户在 ARM 上运行作业时,尽可能使用集群上编译安装好的公共软件,见 公共软件。在 X86 节点上运行作业时可自行在 X86 登录节点编译安装应用软件。bioconda 中的软件绝大部分都是基于 X86 编译的,因此如果需要使用 bioconda 中的应用软件,建议在 X86 节点安装 conda(或 mamba),然后再安装所需的应用软件。
-
每个用户可使用的计算和存储资源有一定限制。数据较多、作业任务较多的用户建议时常使用
diskquota命令查看自己的存储使用量,以免因为存储空间达到配额限制导致程序挂掉。存储超配额后,无法写入数据,同时也无法登录集群,均会出现Disk quota exceeded的报错。 -
集群存储只存储用户目前使用的数据,结果数据应该及时下载到本地,计算中间数据及时删除。测序原始数据一定要在本地备一份。长时间不使用的数据及时压缩后备份到本地,避免额外的存储使用费用。删除数据前请仔细确认,集群上的数据删除后不可恢复。
-
NR、NT等生信数据库目录
/share/software/biodb/。 -
大批量的群体数据处理时,不要直接保留sam文件,见 短序列比对输出bam, 数据使用规范 。群体数据处理过程中会使用大量的存储空间,建议群体数据处理过程中时常使用
diskquota命令查看自己的存储使用量,以免存储使用达到配额限制导致程序挂掉或空跑。
平台使用 ↵
计算资源¶
华中农业大学超算平台集群计算资源包含2类资源,一类为HPC算力(x86),一类为HPC算力(ARM),其中x86资源包含:2台登录节点、58台计算节点、3台大内存节点;ARM 资源包含:1台运维节点、2台登录节点、2台管理节点、43台计算节点、1台NPU节点、3台大内存节点。
各节点详细硬件配置如下:
| 节点类型 | 算力类型 | 节点名称 | 节点数量 | CPU型号 | 核心数 | 内存 |
|---|---|---|---|---|---|---|
| 登录节点 | x86 | cli_X86_01 cli_X86_02 | 2 | Intel Xeon Sliver 4316, 2.3GHz, 20c | 20 | 512G |
| 计算节点 | x86 | agent_X86_01 ~ agent_X86_58 | 58 | Intel Xeon Platinum 8358P, 2.6GHz, 32c | 64 | 512G |
| 大内存节点 | x86 | fat_agent_X86_01 ~ fat_agent_X86_03 | 3 | Intel Xeon Platinum 8358P, 2.6 GHz, 32c | 64 | 2048G |
| 运维节点 | arm | eSightSever | 1 | 鲲鹏 920, 2.6GHz, 32c | 64 | 64G |
| 管理节点 | arm | master_01 master_02 | 2 | 鲲鹏 920, 2.6GHz, 64c | 128 | 512GB |
| 登录节点 | arm | cli_ARM_01 cli_ARM_02 | 2 | 鲲鹏 920, 2.6GHz, 64c | 128 | 512GB |
| 计算节点 | arm | agent_ARM_01 ~ agent_ARM_43 | 43 | 鲲鹏920, 2.6GHz, 64c | 128 | 512GB |
| NPU节点 | arm | AI-node | 1 | 鲲鹏 920, 2.6GHz, 48c ;8 路昇腾 NPU AI,单AI规格为313TFlops@FP16,64GB HBM | 128 | 1024G |
| 大内存节点 | arm | fat_agent_ARM_01 ~ fat_agent_ARM_03 | 3 | 鲲鹏920, 2.9GHz, 64c | 128 | 2048G |
存储资源¶
存储资源包含1框8节点华为全闪存储OceanStor Pacific 9950,4框8节点华为大容量存储OceanStor Pacific 9550,其中9550采用DPC+标准协议组网场景,2个25G端口上行连到业务面接入交换机,每节点2个100GE端口接入到计算存储接入交换机,1个GE端口连到带外管理接入交换机。9950采用DPC组网场景,每节点2个100GE端口接入到计算存储接入交换机,1个GE端口连到带外管理接入交换机。
全闪存储OceanStor Pacific 9950单节点数据盘10块7.68TB SSD,总体提供可用容量460TB。大容量存储OceanStor Pacific 9550 单节点数据盘60块16TB SATA,单节点缓存盘4块1.6TB NVME SSD,提供可用容量5.8PB。
网络资源¶
网络资源包含22台交换机、2台防火墙。集群网络分为四个网络平面,计算/存储网络、业务面网络、带外管理网络、外网接入区。
计算/存储网络采用RoCE网络技术,通过AI人工智能等RoCEv2分布式应用提供“无丢包、低时延、高吞吐”的网络环境,满足分布式应用的高性能需求;管理网络采用Spine-Leaf架构,用于管理节点上集群管理软件收集集群各个节点状态信息(如CPU状态、内存使用率、磁盘使用率、在线状态等),并实现管理功能(如时间同步、集群部署、用户管理、作业调度等);带外管理网络通过SNMP技术,带外管理网络和设备的各种状态,主要用于网络设备、计算节点、存储节点等带外监控。
集群整体架构¶
| 资源类型 | 单位 | 校内(元) | 校外(元) |
|---|---|---|---|
| ARM 节点 | 核时 | 0.04 | 0.08 |
| X86 节点 | 核时 | 0.05 | 0.1 |
| NPU 节点 | 卡时 | 4 | 8 |
| 存储 | GB/月 | 0.025 | 0.05 |
帐号申请¶
用户范围¶
本系统实行收费使用的管理办法,面向全校师生开放使用。
帐号申请时间¶
本集群每学期初和学期中(第12周左右)开放一次申请、考核,具体的通知会发到超算网站(hpc.hzau.edu.cn)、超算用户QQ群、公众号(作物遗传改良卓越工程师工作室)等处。由于各种原因错过每学期帐号申请的同学,需等到下次开放申请。
帐号申请流程¶
- 学期初或12周左右,管理员下发集群开放申请通知,并开放集群考核报名的在线文档,如果有需要可填写报名文档参加考核。时间约为一周,期间练习账号也会开放,可向管理员申请使用。
- 申请用户参加集群用户考核,考核形式为纸质试卷+上机测试,考核内容见 集群考试内容。考试为开卷考试,可以携带学习资料、使用机房电脑搜索查询等。
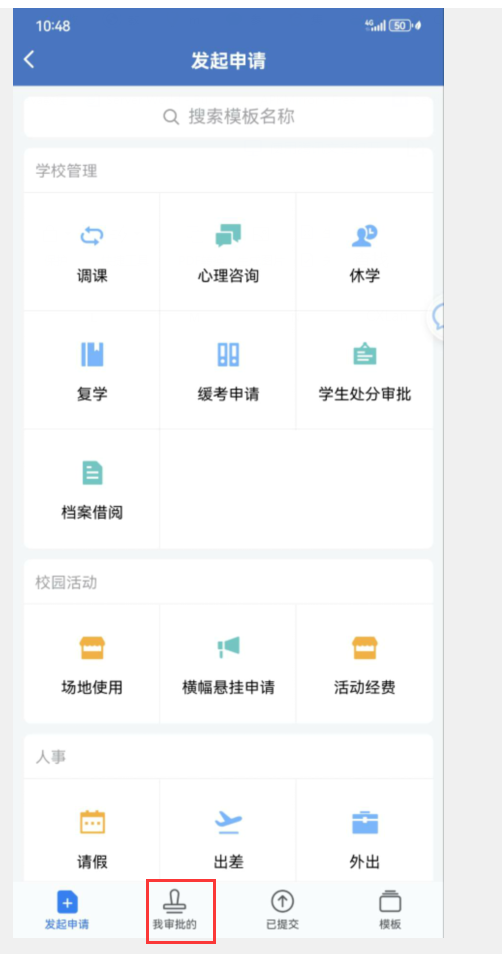
- 考核80分通过,通过的申请用户需在企业微信上提交
超算账号申请审批单,审批人为导师。 - 管理员开通集群帐号,并将相关信息发到申请用户邮箱。
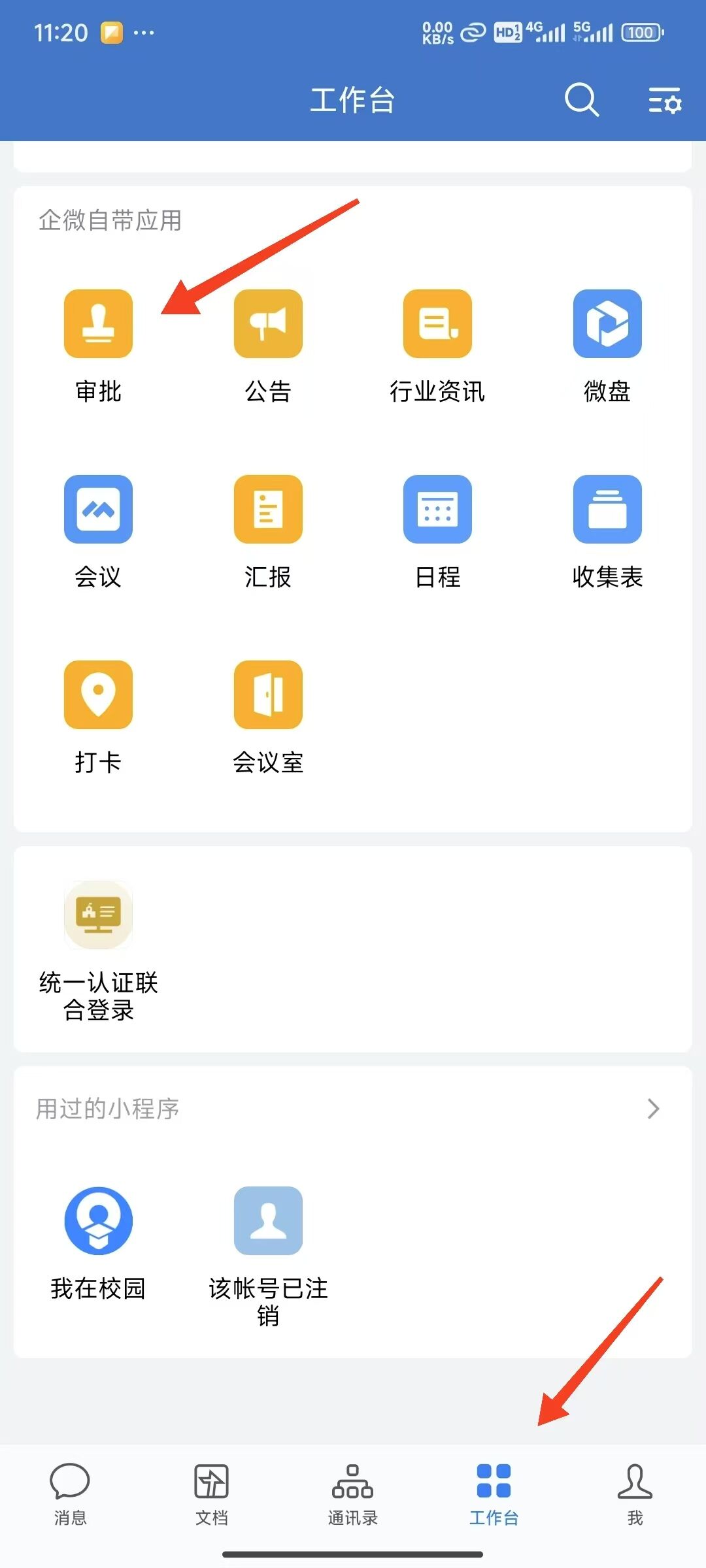
使用企业微信提请账号审批流程¶
如果没有加入“华中农大微校园”企业微信,可以按链接中的指南加入,企业微信加入指南
通过企微提请一个新集群账号申请的审批单,审批人为导师,
审批的位置:打开企微app->工作台->审批
导师审批的位置:工作台->审批->我的审批
计算资源限制¶
x86 计算资源(x86、fat_x86队列)每人可同时使用 1200 核,ARM 计算资源(arm、fat_arm 队列)每人可同时使用 2000 核。
存储资源¶
目前对每个新用户所能使用的存储空间限制为10TB,超过配额账号无法写入数据。当系统总的存储使用过高时会通知大家清理数据。
查看账号存储使用¶
用户可使用diskquota 命令查看当前存储使用量和配额,避免写满。特别是当有产生大量中间文件和结果文件的作业运行时,需时时关注,及时清理,以免超过配额。由于海量的小文件(小于128KB)会对存储系统性能有一定影响,因此大量小文件使用完之后需要即使删除或者打包压缩存放。
如下,当前的存储使用量为 5339125964800KB,存储配额为 10995116277760 KB;账号下文件数据量为 2097711,文件数配额为 1000000000;
$ diskquota
username space_used(KB) space_quota(KB) files_used files_quota
-------- -------------- --------------- ---------- -----------
user 5339125964800 10995116277760 2097711 1000000000
Disk quota exceeded。清理账号下的数据至配额之下方可正常写入数据。
$ diskquota
username space_used(KB) space_quota(KB) files_used files_quota
-------- -------------- --------------- ---------- -----------
user 15339125964800 10995116277760 2097711 1000000000
$ cp data/NT/taxdb.btd.gz .
cp: error writing ‘./taxdb.btd.gz’: Disk quota exceeded
cp: failed to extend ‘./taxdb.btd.gz’: Disk quota exceeded
命令行¶
参考
Warning
第一次使用多瑙调度器之前需要执行 dlogin ,输入用户密码后获取用户token。
作业提交¶
dsub
命令行提交作业¶
dsub -n jobname -q arm -aa -R 'cpu=4' -o jobname_%J.out sleep 100
脚本提交作业¶
#!/bin/bash
#DSUB -n jobname
#DSUB -R 'cpu=2'
#DSUB -o jobname_%J.out
#DSUB -aa
#DSUB -q arm
date
echo "this is script job"
sleep 10
date
提交
# 作业脚本需要有可执行权限
$ chmod +x submit.sh
$ dsub -s submit.sh
-
-n指定作业名称。 -
-R作业申请的资源,-R 'cpu=2申请 2 个 CPU 核。 -
-aa使用任意 CPU 架构的计算节点。 -
-q指定作业列,-q arm指定使用 arm 队列,作业队列的详细说明见下文的 作业队列。-q选项和-aa选项需一起使用。
-
-nl指定作业运行的节点,如#DSUB -nl 'agent-ARM-01';也可以指定作业运行的节点范围,#DSUB -nl 'agent-ARM-0[1-9]'。 -
-o指定作业日志。-o以追加的方式写入,-oo以覆盖的方式写入。日志文件名可以使用特殊变量:%U表示userName;%J表示JobID;%G表示TaskGroupName;%I表示Index;%A表示UserName_JobID_TaskGroupName_Index。
dsub -w 提交阻塞式作业,提交作业并等待作业结束,用于写分析流程,与 lsf -K 作用相同。
Warning
目前已弃用使用标签对资源进行分类使用的方式,请使用队列的方式将作业提交到 arm 或 x86 节点。作业队列
资源标签:集群有多种硬件资源,arm 节点、x86 节点、ai 节点以及胖节点,集群上使用不同的资源标签代表不同类型的节点,以方便作业提交到对应类型的节点运行,可以使用 dadmin label show 查看资源标签及对应的节点。
交互作业¶
使用 -I 选项可以提交交互作业,用于程序调试等。
$ dsub -q interactive -I bash
交互模式进入 AI 节点,AI 节点使用需要向管理员申请。
$ dsub -aa -q AI -I bash
MPI 并行作业¶
提交MPI跨节点并行作业时,需要使用 --mpi 选项指定支持的MPI类型,支持 openmpi,intelmpi,hmpi和mpich。
这里以 lammps 为例。
#DSUB -n lammps
#DSUB -N 256
#DSUB -nn 2
#DSUB --mpi hmpi
#DSUB -o lammps_arm_%J.out
#DSUB -aa
#DSUB -q arm
module load arm/lammps/29Aug2024_hmpi
# 以下三行打印hostfile文件,用于debug,正常运行可以不需要
echo $CCS_MPI_OPTIONS
hostfile=$(echo $CCS_MPI_OPTIONS| sed s'/-hostfile //g')
cat $hostfile
mpirun $CCS_MPI_OPTIONS -x OMP_NUM_THREADS=1 lmp_mpi -in in.lj
# 3d Lennard-Jones melt
variable x index 4
variable y index 4
variable z index 4
variable xx equal 20*$x
variable yy equal 20*$y
variable zz equal 20*$z
units lj
atom_style atomic
lattice fcc 0.8442
region box block 0 ${xx} 0 ${yy} 0 ${zz}
create_box 1 box
create_atoms 1 box
mass 1 1.0
velocity all create 1.44 87287 loop geom
pair_style lj/cut 2.5
pair_coeff 1 1 1.0 1.0 2.5
neighbor 0.3 bin
neigh_modify delay 0 every 20 check no
fix 1 all nve
run 10000
提交 NPU 作业¶
NPU 作业脚本,使用 -R 'npu=1' 选项申请 NPU 资源。
#!/bin/bash
#DSUB -n jobname
#DSUB -R 'npu=1'
#DSUB -o jobname_%J.out
#DSUB -aa
#DSUB -q AI
python npu.py
dnode --npu 命令可以查看集群 NPU 的使用情况。
$ dnode --npu
NAME NPU_ID DEVICE_ID NPU_NAME STATUS AICORE_UTIL DDR_USAGE HBM_USAGE
AI-01 0 0 Atlas A2 OK 0% 0/0 64433/655*
AI-01 1 0 Atlas A2 OK 0% 0/0 64890/655*
AI-01 2 0 Atlas A2 OK 0% 0/0 63353/655*
AI-01 3 0 Atlas A2 OK 0% 0/0 63353/655*
AI-01 4 0 Atlas A2 OK 0% 0/0 63359/655*
AI-01 5 0 Atlas A2 OK 0% 0/0 63353/655*
AI-01 6 0 Atlas A2 OK 0% 0/0 63353/655*
AI-01 7 0 Atlas A2 OK 0% 0/0 63355/655*
批量作业提交¶
一般用于使用同一个应用程序处理多个数据样本、或运行不同参数的场景,以提高作业提交效率、避免手工提交失误。
for sample in /share/home/username/work/lsf_bwait/raw_data/*_R1.fastq.gz;do
index=$(basename $sample |sed 's/_R1.fastq.gz//')
prefix=$(dirname $sample)
dsub -n "bwa_${prefix}" -aa -q arm -R 'cpu=40' -o bwa_${prefix}_%J.out "module load arm/bwa/0.7.18 arm/samtools/1.21;bwa mem -t 40 -R "@RG\tID:${prefix}\tPL:illumina\tLB:library\tSM:humen146" hg38.fa ${prefix}_R1.fastq.gz ${prefix}_R2.fastq.gz |samtools sort -@ 40 -o ${prefix}_srt.bam"
作业依赖¶
用于提交作业时,指定作业间的依赖关系,当被依赖的作业达成约定的状态后依赖的作业将会被调度。
dsub -D "key=value"
-
输入的参数值必须满足
key=value格式。key支持jobid和JobName,value值只能是STARTED、RUNNING、SUCCEEDED、FAILED、ENDED,如'2=ENDED'。STARTED表示RUNNING、STOPPED、SSTOPPED、SUCCEEDED和FAILED。ENDED表示SUCCEEDED和FAILED。
-
jobid约束如下:
- 必须满足jobid的限制条件,即1~12位正整数。
- 支持指定同一个jobid的多种状态,但指定同一作业的不同状态为与逻辑时,将导致依赖无法达成。
-
JobName约束如下:
- 若包含特殊字符
%=()|,需要使用\对特殊字符进行转义。 - 支持使用通配符
(*)模糊查询,可放置开头、中间或结尾,分别对应的匹配方式为后缀匹配、关键字匹配和前缀匹配。 - 只能依赖本用户提交的作业。
- 若包含特殊字符
-
多个匹配条件组合时,支持多匹配条件间的
“与(&&)/或(||)”逻辑,且支持以分号表示与逻辑,分号和逻辑符(&&、||)不支持混用。 -
不加单引号的纯数字按
jobid处理;加单引号的纯数字按JobName处理。
使用举例:
-
示例一:
按照jobid指定单个作业依赖关系
dsub -D "1=RUNNING" "echo 'hello world'"显示如下:
Submit job <2> successfully. -
示例二:
按照JobName指定依赖关系
dsub -D "job1=RUNNING" "echo 'hello world'"显示如下:
Submit job <3> successfully. -
示例三:
按照jobid指定多个作业依赖关系
dsub -D "(1=RUNNING||2=SUCCEEDED)&&3=ENDED" "echo 'hello world'"显示如下:
Submit job <4> successfully. -
示例四:
按照JobName指定依赖关系(支持通配符*)
dsub -D "job_*=SUCCEEDED" "echo 'hello world'"显示如下:
Submit job <5> successfully. -
示例五:
指定JobName包含特殊字符(特殊字符需要转义)
dsub -D "job\(202306\)=SUCCEEDED" "echo 'hello world'"显示如下:
Submit job <6> successfully. -
示例六:
指定纯数字的JobName
dsub -D "'12'=RUNNING" "echo 'hello world'"显示如下:
Submit job <7> successfully.
作业队列¶
Warning
目前已弃用使用标签对资源进行分类使用的方式,请使用队列的方式将作业提交到 arm 或 x86 节点。
为了对资源和作业进行管理,将节点分成了不同的队列,使用 dqueue 查看队列。
$ dqueue
NAME STATUS PRIORITY RUNNING_JOBS PENDING_JOBS SSTOPPED_JOBS
default OPEN,ACTIVE 1 266 2140 0
x86 OPEN,ACTIVE 1 146 143 0
AI OPEN,ACTIVE 1 0 0 0
interactive OPEN,ACTIVE 1 9 0 0
fat_x86 OPEN,ACTIVE 1 6 10 0
arm OPEN,ACTIVE 1 202 335 0
fat_arm OPEN,ACTIVE 1 30 523 0
| 队列 | 对应的节点 | 节点CPU核数 | 节点内存(GB) |
|---|---|---|---|
| arm | agent-ARM-{01-43} | 128 | 512 |
| x86 | agent-X86-{01-65} | 64 | 512 |
| AI | AI-01 | 192 | 1024 |
| fat_arm | fat-agent-ARM-{01-03} | 128 | 2048 |
| fat_x86 | fat-agent-X86-{01-03} | 64 | 2048 |
-q 选项和 -aa 选项需一起使用,否则作业无法调度至计算节点运行。
- 提交作业到普通 x86 节点,需要指定 x86 队列
#DSUB -n jobname # 指定作业名,可自定义
#DSUB -R 'cpu=2' # 每节点使用的资源,示例表示每节点使用2个cpu核心
#DSUB -o jobname_%J.out # 指定错误输出日志,%J为固定写法,表示作业ID
#DSUB -aa # 任意架构
#DSUB -q x86 # 指定 x86 队列
program # 用户自己的程序
- 提交作业到普通 arm 节点,需要指定 arm 队列
#DSUB -n jobname # 指定作业名,可自定义
#DSUB -R 'cpu=2' # 每节点使用的资源,示例表示每节点使用2个cpu核心
#DSUB -o jobname_%J.out # 指定错误输出日志,%J为固定写法,表示作业ID
#DSUB -aa # 任意架构
#DSUB -q arm # 指定 arm 队列
program # 用户自己的程序
- 提交作业到 x86 大内存节点,需要指定 fat_x86 队列
#DSUB -n jobname # 指定作业名,可自定义
#DSUB -R 'cpu=2' # 每节点使用的资源,示例表示每节点使用2个cpu核心
#DSUB -o jobname_%J.out # 指定错误输出日志,%J为固定写法,表示作业ID
#DSUB -aa # 任意架构
#DSUB -q fat_x86 # 指定 x86 队列
program # 用户自己的程序
- 提交作业到 arm 大内存节点,需要指定 fat_arm 队列
#DSUB -n jobname # 指定作业名,可自定义
#DSUB -R 'cpu=2' # 每节点使用的资源,示例表示每节点使用2个cpu核心
#DSUB -o jobname_%J.out # 指定错误输出日志,%J为固定写法,表示作业ID
#DSUB -aa # 任意架构
#DSUB -q fat_arm # 指定 arm_fat 队列
program # 用户自己的程序
内存限制¶
为了防止作业使用过多内存导致计算节点崩溃,调度系统对所有作业按核数强制限制了作业能使用内存的最大值,如 arm 队列的节点限制为每核 5GB,超过限制作业会被系统杀掉,同时日志文件内会有相关提示 Job was terminated due to reaching the mem limit, errCode: 143,如果程序需要使用 50GB 内存,则应该申请 10 个核, -R 'cpu=10' 。各节点对应的内存限制如下:
| 队列 | 节点 | 每核内存限制(GB) |
|---|---|---|
| arm | agent-ARM-{01-43} | 5 |
| x86 | agent-X86-{01-65} | 10 |
| AI | AI-01 | 10 |
| fat_x86 | fat-agent-X86-{01-03} | 32 |
| fat_arm | fat-agent-ARM-{01-03} | 16 |
dsub 其它选项¶
查看作业¶
基本使用¶
提交作业后,查看作业运行状态:djob 或 djob jobid。
$ djob
ID NAME STATE USER ACCOUNT QUEUE START_TIME END_TIME EXEC_NODES
9 gatk RUNNING usrename default default 2024/10/09 08:49:52 - agent-ARM-15
| 字段 | 含义 |
|---|---|
| ID | 作业ID |
| NAME | 作业名称,若未指定作业名称,则默认为default。 |
| STATE | 作业状态,包含WAITING、PENDING、RUNNING、STOPPED、SSTOPPED、FAILED、SUCCEEDED。 |
| USER | 提交该作业的用户名。 |
| ACCOUNT | 作业所在组织帐户。若未指定组织帐户提交或该用户未配置defaultAccount,则默认提交至default。 |
| QUEUE | 作业所在队列。若未指定队列提交或该用户未配置 defaultQueue,则默认提交至default。 |
| START_TIME | 作业开始时间。 |
| END_TIME | 作业结束时间。 |
| EXEC_NODES | 作业执行节点。 |
作业状态:
-
WAITING:调度器接收用户提交的作业,作业的初始状态是WAITING,等待调度器调度作业。 -
RUNNING:调度作业后,其状态产生两种变化:如果计算资源分配成功,作业标识为RUNNING,并分发到执行节点运行; -
PENDING:如果资源未分配成功,作业标识为PENDING,调度器将作业挂起,并发布具体原因。使用djob -l查看作业的调度详情,了解挂起原因。 -
SUCCEED:如果作业正确执行完成,标识为SUCCEED。通过查看作业输出数据了解业务计算结果。 FAILED:如果执行失败,标识为FAILED。使用djob -l查看作业的运行时详情,了解失败原因。
定制 djob 输出¶
为方便查看作业的CPU时间、内存消耗,可以改写 djob 的输出,将下面这行 alias 命令写入 ~/.bashrc 中。
alias dbs="djob --output 'jobId:8 name:8 user:8 state:8 queue:8 startTime:20 execNodes:15 totalMaxMem:15 totalUtime:15 totalStime:15'"
$ dbs
jobId name user state queue startTime execNodes totalMaxMem totalUtime totalStime
13 gatk liuhao RUNNING default 2024/10/09 11:28:57 agent-ARM-15 151168 156416 2560
作业实际 CPU 消耗¶
部分作业申请了使用多核,但由于程序本身无法充分利用多核,因此建议关注作业是否实际使用了申请的核,并以此为依据申请作业使用的核数,以免浪费核时,产生不必要的费用。
cpuUtil:值格式:avg/recent,表示作业的总CPU利用率,即从作业开始运行到最近一次采样。
-
avg:平均CPU使用率,从作业开始运行到最近一次采样的平均使用率;MPI作业为节点上该作业所有任务的平均值。 -
recent:最近一次上报的CPU使用率;
如下作业所示,当前使用了 98 核,作业从运行到目前平均使用了 9.7 核
$djob -ll jobid
...
Runtime Details:
...
nodeUsages
execNode resGroup resIndex utime stime wallclockDuration sstopedTime cpuUtil gpuUtil memResource
agent-ARM-42 0 0 200370 71784 27882 0 976/9820 - 329394/0/0/397740/397740/363698/329640
nodePids
execNode pids
agent-ARM-42 3107938
...
djob 其它选项¶
查看作业日志¶
dpeek
$ cat jobname_112345.out
TASK_INFO:
JOB_ID: 25866930
JOB_NAME: GB12
TASK_NAME: rg0.0
REPLICA: 1
TASKGROUP_NAME: rg0
SUBMIT_NODE: cli-X86-01
EXEC_NODE: agent-X86-26
EXIT_CODE: 0
EXIT_MESSAGE: Job execution succeeded
RESOURCE_USAGE_SUMMARY:
REQ_CPU: 20
REQ_MEM: 128
REQ_GPU: 0
REQ_NPU: 0
USER_CPU_TIME: 66703
SYSTEM_CPU_TIME: 550
MEMSW_MAX: 26080
SWAP_MAX: 0
MEM_MAX: 26080
MEM_AVG: 20302
MRUN_TIME: 3373
SSTOPPED_TIME: 0
CPU_UTIL_AVG(%): 1993
CPU_UTIL_RECENT(%): 1375
USER_CPU_TIME_SUM: 66703
SYSTEM_CPU_TIME_SUM: 550
MEMSW_MAX_SUM: 26080
SWAP_MAX_SUM: 0
MEM_MAX_SUM: 26080
MEM_AVG_SUM: 20302
CPU_UTIL_AVG_SUM(%): 1993
CPU_UTIL_RECENT_SUM(%): 1375
GPU_SM_UTIL_AVG(%): -
GPU_SM_UTIL_RECENT(%): -
NPU_SM_MEM_SUM(MB): -
NPU_SM_AICORE_UTIL(%): -
终止作业¶
dkill jobid 终止作业
dkill --force jobid 强制终止作业
作业控制¶
djctl stop JobID 挂起作业
djctl resume JobID 恢复作业
djctl requeue JobID 重启作业
djctl resubmit JobID 重新提交作业
修改作业资源¶
资源查看¶
节点信息¶
dnode 查看所有可用的节点及每个节点的状态、CPU、内存等资源;
| 状态 | 状态说明 |
|---|---|
| OK | 节点正常,允许关闭节点。 |
| CLOSE_ADMIN | 节点被管理员关闭,无法接收作业下发任务,允许开启节点。 |
| CLOSE_LOCK | 节点上作业批量失败,被隔离,需要管理员手动开启。 |
| CLOSE_BUSY | 节点繁忙,资源使用达到阈值,停止下发新作业,阈值下降后恢复为OK状态。 |
| CLOSE_FAULT | 节点局部故障,通信、网卡、磁盘等异常,停止下发新作业需要管理员检查处理,检测到故障解除后恢复为OK。 |
| UNAVAILABLE | 节点无法连接,或出现严重故障(共享存储故障/所有CX网卡掉线等),长期未收到节点心跳,无法接收作业下发任务,允许开启/关闭/删除节点,但是节点状态不发生变化。 |
| REGISTERING | 节点注册中,无法接收作业下发任务,不允许开启/关闭/删除节点。 |
| UNLICENSED | 在线下场景,表示系统中没有可用License文件、License文件中无计算节点资源项(CCSU-00-E01R/CCSU-00-E02R)或License文件中计算节点资源项(CCSU-00-E01R/CCSU-00-E02R)数量不足。 |
| SUSPENDED_ADMIN | 管理员手动休眠节点,无法接收作业下发任务,不允许开启/关闭/删除节点。 |
| SUSPENDED_AUTO | 节点自动进入休眠状态,可以接收作业下发任务,不允许开启/关闭/删除节点。 |
| CLOSE_EXIT | 节点即将删除状态。无法接收作业下发任务,但是正在运行的作业不受影响。该节点上的所有作业运行完成后,会自动被删除。该状态的节点仅支持强制删除操作,此时节点上的作业会被终止。 |
| CLOSE_FULL | CPU核数和GPU卡数全部分配完毕,停止下发新作业,资源释放后恢复为OK。 |
dqueue 查看集群可用的作业队列
其它¶
drun 使用不同的作业步骤(Job Step)提交不同的任务
dattach 用于支持用户直接连接作业执行节点或Docker容器
drespool 用于显示资源池信息
dacct 显示组织帐户作业统计和资源等信息
duser 用于显示用户作业统计等信息
dcluster 用于显示集群中节点、作业及队列等信息
流程软件¶
snakemake¶
v8 之前的版本
snakemake --cluster 'dsub -R "cpu={threads}"' -s Snakemake
常见故障¶
- 故障现象:作业提交后立即结束,没有日志输出
查看作业详细信息 djob -ll jobid,可以看到关键报错信息 Job execution environment build failed, err: container save failed failureHost: agent-ARM-03,大概率可能是节点的根目录写满了,请联系管理员及时处理。
查看作业详细日志
$ djob -ll 25670440
Basic Details:
jobId 25670440
name arm_STAR
state FAILED
user username
queue default
account default
cmd module load arm/stringtie/3.0.0;stringtie -p 12 -G /share/home/username/biobase/p_clarkii/pc_v2.0_ncbi_v20631_agat.gtf -o M_N_zx_1.gtf -i M_N_zx_1Aligned.sortedByCoord.q20.bam
submitNode cli-ARM-01
createTime 2025/09/03 17:31:37
startTime 2025/09/03 17:31:39
lastModifiedTime -
endTime 2025/09/03 17:31:39
currentOperation EMPTY
jobRequeueMaxCnt 0
jobRequeueCurrentCnt 0
mpiType DEFAULT
interactionMode DEFAULT
assistJob DEFAULT
x11ForwardEnabled false
description -
transferType LOCAL
forwardTime -
recallTime -
autoResize false
Scheduling Details:
exclusive false
priority 1
adminPriority -1
expectSchedTime -
startTimeEstimated -
reserveRes -
backfillRes -
backfilledRes -
Resource Details:
reqJobLicense -
allocJobLicense -
resGroup[0]:
minReplica 1
replica 1
labels arm aarch64
preferNodes -
reqNodeSelectPolicy -
effNodeSelectPolicy SEQUENCE
reqCPU 12
reqMem 128
reqGPU nvidia:0
reqNPU 0
allocCPU 12
allocMem 128
allocGPU nvidia:0
allocNPU 0
allocSdr -
reqTaskLicense -
allocTaskLicense -
limitMem 61440
allocAffinity
execNode resGroup resIndex hwThreads gpuIds npuIds
agent-ARM-03 0 0/1 - - -
Runtime Details:
execNodes agent-ARM-03
execNodeCnt 1
execPath /share/home/username/biodata/wgcna/bam_2pass
resizeCmd -
timeout 0
stderrRedirectPath /share/home/username/biodata/wgcna/bam_2pass/M_N_zx_1_star_arm_%J.err
stderrRedirectType append
stdoutRedirectPath /share/home/username/biodata/wgcna/bam_2pass/M_N_zx_1_star_arm_%J.out
stdoutRedirectType append
totalUtime 0
totalStime 0
totalMaxMemSwp 0
totalMaxMem 0
preHookRunTime 0
jobWallclockDuration 0
postHookRunTime 0
jobSstoppedTime 0
jobExitCode 0
systemExitCode 12008
exitMessage Job execution environment build failed, err: container save failed failureHost: agent-ARM-03
nodeUsages
execNode resGroup resIndex utime stime wallclockDuration sstoppedTime cpuUtil gpuUtil npuUtil memResource
agent-ARM-03 0 0/1 0 0 0 0 0/0 - - 0/0/0/0/0/0/0/0
nodePids
execNode pids
agent-ARM-03 -
traceMessages 2025/09/03 17:31:37 : [JOB_ADD] Job 25670440 is submitted by username, job state is PENDING.
2025/09/03 17:31:38 : [JOB_START] Job 25670440 start message has been sent, job state is PENDING.
2025/09/03 17:31:38 : [JOB_START_ACK] Job 25670440 start message has been received by agent agent-ARM-22.
2025/09/03 17:31:38 : [JOB_EXECUTION] Job 25670440 state is from PENDING to RUNNING.
2025/09/03 17:31:38 : [JOB_FINISH] Job 25670440 state is from RUNNING to FAILED, reason: job build failed.
2025/09/03 17:31:38 : [JOB_RETRY] Job 25670440 will retry for the 1st time, and max retry times is 1, and retry type is cluster requeue.
2025/09/03 17:31:38 : [JOB_STATE_CHANGE] Job 25670440 state is from FAILED to PENDING.
2025/09/03 17:31:39 : [JOB_START] Job 25670440 start message has been sent, job state is PENDING.
2025/09/03 17:31:39 : [JOB_START_ACK] Job 25670440 start message has been received by agent agent-ARM-03.
2025/09/03 17:31:39 : [JOB_EXECUTION] Job 25670440 state is from PENDING to RUNNING.
2025/09/03 17:31:39 : [JOB_FINISH] Job 25670440 state is from RUNNING to FAILED, reason: job build failed.
NPU 节点 (AI-01) 含 8 张 NPU 卡,卡型号为华为昇腾 910B(Ascend),单卡显存为 64GB HBM。
驱动¶
参考:
Note
集群NPU节点已安装驱动
安装固件¶
# 根据芯片型号选择相应的安装包安装
# 安装firmwire
chmod +x Ascend-hdk-*-npu-firmware_${version}.run
./Ascend-hdk-*-npu-firmware_${version}.run --full
安装驱动¶
# 根据CPU架构 以及npu型号 安装对应的 driver
chmod +x Ascend-hdk-*-npu-driver_${version}_*.run
./Ascend-hdk-*-npu-driver_${version}_*.run --full --install-for-all
npu-smi¶
npu-smi 是 npu 的系统管理工具,可以用于收集设备信息,查看设备健康状态,对设备进行配置以及执行固件升级、清除设备信息等功能。
驱动安装过程中会默认安装 npu-smi 工具。
安装完成后,npu-smi放置在 /usr/local/sbin/ 路径下。
# 查看卡基本信息及运行的进程
$ npu-smi info
# 监控卡使用信息,默认每秒刷新一次
$ npu-smi info watch
NpuID(Idx) ChipId(Idx) Pwr(W) Temp(C) AI Core(%) AI Cpu(%) Ctrl Cpu(%) Memory(%) Memory BW(%)
0 0 89.6 25 0 0 6 5 0
1 0 92.3 26 0 0 3 5 0
2 0 88.6 28 0 0 4 5 0
3 0 90.7 29 0 0 15 5 0
4 0 86.0 28 0 0 5 5 0
5 0 86.7 30 0 0 5 5 0
6 0 109.2 36 0 0 4 62 81
7 0 118.6 36 0 0 2 62 39
ascend-dmi¶
Ascend DMI(Ascend Device Management Interface)工具通过调用底层DCMI(设备控制管理接口)以及AscendCL(Ascend Computing Language,昇腾计算语言)相关接口完成相关检测功能,对于系统级别的信息查询通过调用系统提供的通用库来实现。
ascend-dmi工具主要为Atlas产品的标卡、板卡及模组类产品提供带宽测试、算力测试、功耗测试等功能。
安装
$ ./Ascend-mindx-toolbox_6.0.RC3_linux-aarch64.run --install --install-for-all --nox11
$ source /usr/local/Ascend/toolbox/set_env.sh
-
带宽测试,测试数据从Host侧传输到Device 0,迭代100次的带宽与总耗时。更多 带宽测试
# 定长模式 $ ascend-dmi --bw -t h2d -d 0 -s 8388608 --et 100 Host to Device Test Device 0: Ascend 910B3. ------------------------------------------------------------------- ID Size(Bytes) Execute Times Bandwidth(GB/s) Elapsed Time(us) ------------------------------------------------------------------- 0 8388608 100 23.566224 355.96 ------------------------------------------------------------------- -
算力测试,测试Device 2,指定算子运算类型默认为fp16,执行次数为6千万的算力。更多 算力测试
$ ascend-dmi -f -d 2 --et 60 ----------------------------------------------------------------------------------------- Device Execute Times Duration(ms) TFLOPS@FP16 Power(W) ----------------------------------------------------------------------------------------- 2 360,000,000 1988 313.531 223.300003 -----------------------------------------------------------------------------------------
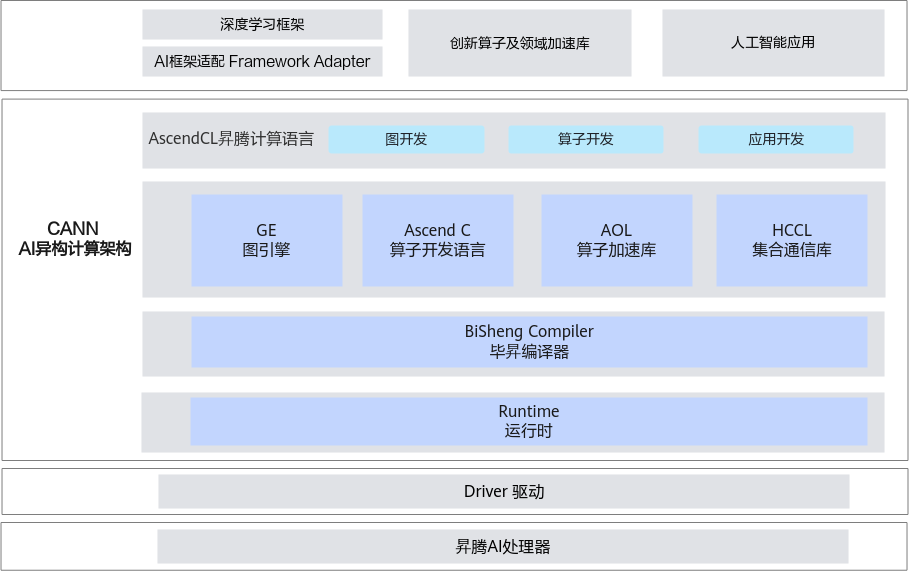
CANN¶
异构计算架构CANN(Compute Architecture for Neural Networks)是昇腾针对AI场景推出的异构计算架构,向上支持多种AI框架,包括MindSpore、PyTorch、TensorFlow等,向下服务AI处理器与编程,发挥承上启下的关键作用,是提升昇腾AI处理器计算效率的关键平台。同时针对多样化应用场景,提供多层次编程接口,支持用户快速构建基于昇腾平台的AI应用和业务。
参考
Note
集群已安装CANN
安装 toolkit¶
# 安装toolkit 以arm为例
chmod +x Ascend-cann-toolkit_${version}_linux-aarch64.run
./Ascend-cann-toolkit_${version}_linux-aarch64.run --install --install-for-all
source /usr/local/Ascend/ascend-toolkit/set_env.sh
安装 kernel¶
# 根据芯片型号选择相应的安装包安装
# 安装 kernel
chmod +x Ascend-cann-kernels-*_${version}_linux.run
./Ascend-cann-kernels-*_${version}_linux.run --install --install-for-all
pytorch for npu¶
预编译包¶
https://www.hiascend.com/document/detail/zh/Pytorch/60RC2/configandinstg/instg/insg_0001.html,这个链接中下载 torch 和 torch_npu 预编译包(whl)。
这里选择的pytorch 版本为 2.3.1,python 版本为 3.9(集群默认python版本),系统架构为 aarch64。可以根据自己的python版本、需要的pytorch版本选择对应版本的 python 包下载安装。
查看 CANN 版本 ls /usr/local/Ascend/ascend-toolkit/。
# 下载PyTorch安装包
$ wget https://download.pytorch.org/whl/cpu/torch-2.3.1-cp39-cp39-manylinux_2_17_aarch64.manylinux2014_aarch64.whl
# 下载torch_npu插件包
$ wget https://gitee.com/ascend/pytorch/releases/download/v6.0.rc2-pytorch2.3.1/torch_npu-2.3.1-cp39-cp39-manylinux_2_17_aarch64.manylinux2014_aarch64.whl
# 安装命令
$ pip3 install torch-2.3.1-cp39-cp39-manylinux_2_17_aarch64.manylinux2014_aarch64.whl
$ pip3 install torch_npu-2.3.1-cp39-cp39-manylinux_2_17_aarch64.manylinux2014_aarch64.whl
可安装的版本 https://pypi.org/project/torch-npu/#history
$ pip3 install torch_npu==2.3.1 "numpy<2.0"
import torch 出现报错 Failed to load PyTorch C extensions,可尝试设置环境变量
export LD_LIBRARY_PATH=$HOME/.local/lib/python3.9/site-packages/torch_npu/lib:$LD_LIBRARY_PATH
安装后测试 python3 -c "import torch;import torch_npu; a = torch.randn(3, 4).npu(); print(a + a);"。
自行编译 torch_npu¶
版本配套关系,这里以 v2.1.0-6.0.rc2 为例。
git clone -b v2.1.0-6.0.rc2 https://gitee.com/ascend/pytorch.git
cd pytorch
bash ci/build.sh --python=3.9
pip3 install --upgrade --upgrade dist/torch_npu-2.1.0.post7+gitunknown-cp39-cp39-linux_aarch64.whl
Warning
经测试,集群上使用预编译的 torch_npu 使用会报错,需自行编译。
使用集群预安装 torch_npu¶
$ module load arm/torch_npu/2.1.0.post7-py3.9
测试使用¶
# 交互模式进入NPU节点
$ dsub -q interactive --label AI -I bash
# 查看卡是否存在
$ lspci | grep d802
# 加载相关环境
$ source /etc/profile
$ source /usr/local/Ascend/ascend-toolkit/set_env.sh
# 查看卡信息
$ npu-smi info
# 测试 pytorch 安装是否正常
$ python3 -c "import torch;import torch_npu;print(torch_npu.npu.is_available())"
# 测试使用 torch 运算,如果出现下面的运算结果,表明 pytorch 框架与插件安装成功
$ python3 -c "import torch;import torch_npu; a = torch.randn(3, 4).npu(); print(a + a);"
tensor([[-0.6066, 6.3385, 0.0379, 3.3356],
[ 2.9243, 3.3134, -1.5465, 0.1916],
[-2.1807, 0.2008, -1.1431, 2.1523]], device='npu:0')
参考
代码迁移¶
已有的基于英伟达 cuda 的 pytorch 代码可以通过更改少量的代码迁移到 npu 上运行,相关方法见:
考试内容¶
集群用户考核内容给分为三块:
- Linux基本使用
- 多瑙调度器使用
- 规章制度和注意事项
Linux基本使用¶
- 文件目录操作,要求掌握文件所属的用户和用户组、文件权限的概念(可读、可写、可执行),理解不同的文件类型,如普通文件、目录、块设备、链接文件等,要求掌握的命令
ls、pwd、cd、mv、rm、mkdir、ln、cp、chmod、alias; - 文本操作,需要掌握至少一种文本编辑器(推荐vim),理解管道、重定向,要求掌握基本的文本处理命令
cat、more、less、head、tail、wc、nl、cut、sort、uniq、grep、sed、awk、file; - 查看系统信息,能使用Linux系统提供的工具和命令查看磁盘使用、系统负载、内存使用、系统版本等信息,需要掌握的命令
df、du、free、uptime; - 进程管理,了解进程和多线程的基本概念,需要掌握的命令
top、ps、bg、fg、jobs、kill、pgrep、nohup、screen、Ctrl+z、Ctrl+c;
- 简单的Linux shellscprit编程知识,掌握基本的语法;
- 其他,文件查找
which、locate、find、where,文件压缩tar、gzip、pigz,查看帮助文档man,远程文件传输scp rsync,不同节点间跳转ssh,查看已使用的命令history,查看当前登陆用户who,文件下载wget,windows文件转Unix文件dos2unix。
linux学习可以参考:Linux 基础
多瑙调度器使用¶
规章制度和注意事项¶
Ended: 平台使用
应用软件 ↵
module¶
MODULEPATH 配置,在个人账号 ~/.bashrc 文件中,写入下面两行代码,执行 source ~/.bashrc 生效。
export MODULEPATH="/share/software/modulefiles/:/share/software/HPCKit/latest/modulefiles:$MODULEPATH"
export appdir="/share/software/app/"
$ module av
---------------------------------------------------------- /share/software/modulefiles ----------------------------------------------------------
arm/automake/1.13 arm/cmake/3.30.5 arm/hisat2/2.1.0 arm/minimap2/2.28 arm/star/2.7.11b x86/hifiasm/0.19.9
arm/automake/1.14 arm/curl/8.10.1 arm/htslib/1.21 arm/ncbi-vdb/3.1.1 arm/tassel/5 x86/hisat2/2.1.0
arm/bcftools/1.21 arm/fastp/0.23.4 arm/isal/2.30.0 arm/nextdenovo/2.5.2 arm/vg/1.60.0 x86/htslib/1.21
arm/bedtools/2.31.1 arm/fastqc/0.12.1 arm/java/17.0.11 arm/openblas/0.3.28 arm/zlib/1.3 x86/java/21.0.2
arm/blast/2.16.0 arm/fasttree/2.1.11 arm/jellyfish/2.3.1 arm/openssl/3.3.2 arm/zstd/1.5.6 x86/megahit/1.2.9
arm/boost/1.86.0 arm/gatk/4.6.0.0 arm/libdeflate/1.8 arm/pangenie/3.1.0 x86/bwa/0.7.18 x86/minimap2/2.28
arm/bowtie2/2.5.4 arm/gcc/12.2.0 arm/libpsl/0.21.5 arm/r/4.4.1 x86/bzip2/1.0.6 x86/samtools/1.21
arm/bwa/0.7.18 arm/gsl/2.8 arm/maven/3.9.9 arm/samtools/1.21 x86/cellranger/7.0.0 x86/star/2.7.11b
arm/clapack/3.2.1 arm/hifiasm/0.19.9 arm/megahit/1.2.9 arm/sratools/3.1.1 x86/gatk/4.6.0.0 x86/vg/1.60.0
-------------------------------------------------------- /usr/share/Modules/modulefiles ---------------------------------------------------------
dot module-git module-info modules null use.own
Key:
modulepath
module 更详细的用法见 module 使用。
$ module load arm/bwa/0.7.18
$ bwa
Program: bwa (alignment via Burrows-Wheeler transformation)
Version: 0.7.18-r1243-dirty
Contact: Heng Li <hli@ds.dfci.harvard.edu>
Usage: bwa <command> [options]
Command: index index sequences in the FASTA format
mem BWA-MEM algorithm
fastmap identify super-maximal exact matches
pemerge merge overlapping paired ends (EXPERIMENTAL)
aln gapped/ungapped alignment
samse generate alignment (single ended)
sampe generate alignment (paired ended)
bwasw BWA-SW for long queries (DEPRECATED)
shm manage indices in shared memory
fa2pac convert FASTA to PAC format
pac2bwt generate BWT from PAC
pac2bwtgen alternative algorithm for generating BWT
bwtupdate update .bwt to the new format
bwt2sa generate SA from BWT and Occ
Note: To use BWA, you need to first index the genome with `bwa index'.
There are three alignment algorithms in BWA: `mem', `bwasw', and
`aln/samse/sampe'. If you are not sure which to use, try `bwa mem'
first. Please `man ./bwa.1' for the manual.
Singularity 镜像¶
集群公共目录 /share/software/image/arm/ 目录下放了一些在 arm 上打包好的 Singularity 镜像,可以直接调用。注意,arm 镜像不可在 x86 节点上使用。
Singularity 更详细的用法见 Singularity使用。
$ module load arm/singularity/4.1.5
$ singularity exec /share/software/image/arm/bwa-0.7.18.sif bwa
Program: bwa (alignment via Burrows-Wheeler transformation)
Version: 0.7.18-r1243-dirty
Contact: Heng Li <hli@ds.dfci.harvard.edu>
Usage: bwa <command> [options]
Command: index index sequences in the FASTA format
mem BWA-MEM algorithm
fastmap identify super-maximal exact matches
pemerge merge overlapping paired ends (EXPERIMENTAL)
aln gapped/ungapped alignment
samse generate alignment (single ended)
sampe generate alignment (paired ended)
bwasw BWA-SW for long queries (DEPRECATED)
shm manage indices in shared memory
fa2pac convert FASTA to PAC format
pac2bwt generate BWT from PAC
pac2bwtgen alternative algorithm for generating BWT
bwtupdate update .bwt to the new format
bwt2sa generate SA from BWT and Occ
Note: To use BWA, you need to first index the genome with `bwa index'.
There are three alignment algorithms in BWA: `mem', `bwasw', and
`aln/samse/sampe'. If you are not sure which to use, try `bwa mem'
first. Please `man ./bwa.1' for the manual.
说明¶
- 各种脚本、jar包、编译好的二进制可执行包, e.g. NCBI直接下载编译好的blast
- 采用C/C++等编写, 只有源码,按文档要求编译, e.g. 下载R源码, 自己编译安装
- 系统自带的包管理器(需root权限)
- bioconda, 生信软件包管理器
- docker、singularity容器镜像
- Python、perl、R等的模块或包
环境变量¶
PATH, 自动搜索安装的可执行文件
LD_LIBRARY_PATH,动态链接库搜索位置
PERL5LIB,perl模块位置
PYTHONPATH,python模块位置
export PATH=/opt/bin/:$PATH #或写入~/.bashrc文件
脚本软件¶
采用Perl/Python等解释型语言编写, 下载后有解释器即可运行。
用法:系统安装对应的解释器, 添加x权限
优点:下载就可以直接用, 修改方便
缺点:可能需要很多依赖, 性能差
$ mv N50.pl ~/opt/bin/
$ export PATH="$PATH:$HOME/opt/bin/" #或写入~/.bashrc文件
jar包¶
采用java或类java语言编写, 开发人员已编译打包好, 下载可用。
用法:系统安装java运行环境JDK, java -jar package.jar
优点:下载就可以直接用, 跨平台
缺点: 相比C/C++性能较差, 对内存有一定要求
$ cd ~/opt
$ wget http://www.usadellab.org/cms/uploads/supplementary/Trimmomatic/Trimmomatic-0.39.zip
$ unzip Trimmomatic-0.39.zip
$ java -jar trimmomatic-0.36.jar PE \
-phred33 input_forward.fq.gz input_reverse.fq.gz \
output_forward_paired.fq.gz output_forward_unpaired.fq.gz \
output_reverse_paired.fq.gz output_reverse_unpaired.fq.gz \
ILLUMINACLIP:/usr/local/src/Trimmomatic/Trimmomatic-0.36/adapters/TruSeq3-PE.fa:2:30:10 \
LEADING:3 TRAILING:3 SLIDINGWINDOW:4:15 HEADCROP:8 MINLEN:36
二进制可执行包¶
采用C/C++等编译语言编写, 开发人员已编译好, 下载可用
用法:下载系统对应版本的二进制软件, 添加x权限
优点:下载就可以直接用, 不依赖编译器
缺点:没法看到源码;不能根据需要预编译;依赖预编译系统的底层库; 跨平台性差
$ cd ~/opt
$ mkdir sratoolkit && cd sratoolkit
$ wget http://ftp-trace.ncbi.nlm.nih.gov/sra/sdk/2.6.3/sratoolkit.2.6.3-centos_linux64.tar.gz
$ tar zxvf sratoolkit.2.6.3-centos_linux64.tar.gz
$ chmod +x ~/opt/sratoolkit/sratoolkit.2.6.3-centos_linux64/bin/*
$ ~/opt/sratoolkit/sratoolkit.2.6.3-centos_linux64/bin/fastdump -h
源码编译¶
采用C/C++等编译语言编写, 开发人员提供源代码以及安装文档, 用户根据平台自行编译
用法: 安装好编译器以及依赖库,按文档要求编译
优点: 可以指定预编译选项;使用自己系统的依赖库
缺点: 对新手不友好; 很多软件编译步骤复杂; 自己手动解决依赖;
常用编译器¶
GCC(GNU Compiler Collection,GNU编译器套件)
- 由GNU开发的编程语言编译器,是采用GPL及LGPL协议所发行的自由软件,是Linux及类Unix标准编译器,被认为是跨平台编译器的事实标准
- GCC可处理C、C++、Fortran、Pascal、Objective-C、Java等其他语言
Intel Composer XE (ntel 编译器)
- Intel编译器是Intel公司发布的x86平台(IA32/INTEL64/IA64/MIC)编译器产品,支持C/C++/Fortran编程语言
- Intel编译器针对Intel处理器进行了专门优化,性能优异,在其它x86处理器平台上表现同样出色
LLVM (Low Level Virtual Machine,底层虚拟机)
- Apple资助开发的编译器,支持C、C++、Objective-C和Objective-C++
- 编译速度快;占用内存小;模块化设计,易与IDE集成及其他用途重用;诊断信息可读性强,有利于调试
源代码后缀规范¶
在Linux系统中,可执行文件没有统一的后缀,系统从文件的属性来区分。而源代码、目标文件等后缀名最好保持统一的规范,便于识别区分。
| 文件类型 | 后缀名 |
|---|---|
| C source | .c |
| C++ source | .C, .cc, .cpp, .cxx, .c++ |
| Fortran77 source | .f, .for |
| Fortran90/95 source | .f90 |
| 汇编source | .s |
| 目标文件 | .o |
| 头文件 | .h |
| Fortran90/95模块文件 | .mod |
| 动态链接库 | .so |
| 静态链接库 | .a |
c语言编译¶
# 编译时,指定生成可执行文件的路径或文件名(-o参数)
$ gcc -o hello hello.c
$ file hello
hello: ELF 64-bit LSB executable, x86-64, version 1 (SYSV),
for GNU/Linux 2.6.4, dynamically linked (uses shared libs), not stripped
$ gcc -o /home/test/hello hello.c
#include <stdio.h>
int main()
{
printf("Hello world.\n");
}
#多个源文件同时编译,生成可执行文件sum
$ gcc -o sum main.c function.c
# 源文件分别编译,再将目标文件连接成可执行文件
$ gcc -c main.c
$ gcc -c function.c
$ gcc -o sum main.o function.o
#include <stdio.h>
int main()
{
int sum=0,r,i;
for(i=1;i<=10;i++)
{
r=function(i);
sum=sum+r;
}
printf("sum is %d\n",sum);
}
int function(int x)
{
int result;
result=x*x; return(result);
}
Makefile¶
源文件数量非常多、存放在不同目录下、相互之间有各种依赖关系以及先后顺序关系时,需要使用Makefile进行管理。Makefile定义了一系列的规则来指定哪些文件需要先编译,哪些文件需要后编译,哪些文件需要重新编译,甚至于进行更复杂的功能操作。
- 软件程序的管理工具
- 定义规则,实现自动化编译
- 处理源代码、目标文件、头文件、库文件等依赖关系
- 根据规则和依赖关系,结合时间戳实现精细化控制
make命令执行 Makefile 中的定义的编译流程。make命令默认读取当前目录 Makefile 或 makefile 文件,也可以用 -f 参数指定 Makefile 文件
configure¶
autotool生成configure文件, 有程序不提供configure, 提供autogen.sh
Makefile.am和makefile.in生成Makefile
大型开源程序通常使用configure脚本生成Makefile,Configure脚本作用:
- 检查编译环境 (数据类型长度(int),操作系统,CPU平台)
- 检查依赖头文件及库文件
- 设置安装路径
- 设置编译器及编译参数
configure → make → make install
Configure常用参数:
- --prefix=/opt/software 指定安装路径
- -h 查看configure帮助,configure支持选项
- CC=gcc/icc 设置c语言编译器
- CFLAGS=-O2 –funrool- c编译器参数
- CXX=g++/icpc 设置c++编译器
- CXXFLAGS=-O2 c++编译器参数
- FC=gfortran/ifort 设置fortran编译器
- FCFLAGS=-O2 fortran编译器参数
- --with-XXX 编译时使用XXX包
- --without-XXX 编译时不使用XXX包
- --enable-XXX 启用XXX特性
- --disable-XXX 不启用XXX特性
编译安装samtools
$ wget https://github.com/samtools/samtools/releases/download/1.3.1/samtools-1.3.1.tar.bz2
$ tar xvfj samtools-1.3.1.tar.bz2
$ cd samtools-1.3.1
$ ./configure –prefix=/home/username/opt/samtools/1.3.1
$ make
$ make install
$ echo "export PATH=/home/username/opt/samtools/1.3.1:$PATH" >> ~/.bashrc
cmake¶
cmake:跨平台编译工具,生成makefile。其配置文件为CMakeLists.txt。
cmake → make → make install
cmake常用参数:
- -DCMAKE_INSTALL_PREFIX=/opt/software 指定安装路径
- -DCMAKE_C_COMPILER=/opt/gcc/bin/gcc 设置c语言编译器
- -DCMAKE_CXX_FLAGS ="-O2 –funrool-" c编译器参数
- -DCMAKE_CXX_COMPILER = =/opt/gcc/bin/gcc 设置c++编译器
也可以使用CC CXX 指定gcc
$ export CC=$HOME/opt/gcc/9.4/bin/gcc
$ export CXX=$HOME/opt/gcc/9.4/bin/g++
$ tar –xf gromacs-5.1.4.tar.gz
$ cd gromacs-5.1.5
$ mkdir build && cd build
$ cmake ..
$ make –j 20
$ make install
系统包管理器¶
用法:不同的操作系统用法不大一样 yum: RedHat, CentOS, Fedora; apt-get: Ubuntu, Debian; brew:MacOS
优点:简单, 一键搞定;包管理器自己解决软件依赖
缺点:生物信息软件大部分不在包管理器中, 用于安装底层依赖库;需要root权限
# ubuntu
sudo apt-get -y install libcurl4-gnutls-dev
sudo apt-get -y install libxml2-dev
sudo apt-get -y install libssl-dev
sudo apt-get -y install libmariadb-client-lgpl-dev
# centos
$ yum search openssl
$ yum install -y openssl-devel
conda¶
Anaconda 用于科学计算Python发行版,使用conda 管理包和环境。conda 不仅管理安装python包,还可以是各种其他的应用软件。
优点:不需要root权限;自行解决依赖关系;一键安装, 不需要配置环境
缺点:有些软件conda中没有, 需要自己手动安装;环境混乱、版本管理麻烦
Note
conda 安装软件较慢,可以使用mamba代替,使用方式与conda一直。其配置使用见集群文档 mamba。
配置¶
# 第一步:下载miniconda3
$ wget https://nanomirrors.tuna.tsinghua.edu.cn/anaconda/miniconda/Miniconda3-latest-Linux-$(uname -m).sh
# 第二步:安装miniconda3
$ bash Miniconda3-latest-Linux-x86_64.sh -b -p $HOME/miniconda3
# 第三步:将miniconda3保存到环境路径并启用
$ echo "export PATH=$PREFIX/bin:"'$PATH' >> ~/.bashrc
$ source ~/.bashrc
#第四步:基本配置bioconda,添加清华源镜像
$ conda config --add channels https://mirrors.tuna.tsinghua.edu.cn/anaconda/pkgs/free
$ conda config --add channels https://mirrors.tuna.tsinghua.edu.cn/anaconda/cloud/conda-forge
$ conda config --add channels https://mirrors.tuna.tsinghua.edu.cn/anaconda/cloud/bioconda
$ conda config --set show_channel_urls yes
管理软件包¶
# 搜索需要安装的软件包,获取其完成名字
conda search <package name>
# 安装软件包
conda install <package name>
# 安装特定版本的软件包
conda install <package name>=版本号
# 更新软件包
conda update <package name>
# 移除软件包
conda remove <package name>
# 安装R,及80多个常用的数据分析包, 包括idplyr, shiny, ggplot2, tidyr, caret 和 nnet
conda install -c r r-essentials
管理环境¶
通过conda环境,可以实现软件版本管理、流程环境管理。
# 创建名为env_name的新环境,并在该环境下安装名为 package_name 的包
$ conda create -n env_name package_name
# 可以指定新环境的版本号,例如:创建python2环境,python版本为2.7,同时还安装了numpy pandas包
$ conda create -n python2 python=2 numpy pandas
# 激活 python2环境,通过python -V可以看到是python2.7
$ conda activate python2
# python2 环境中安装相关包
$ conda install pandas
# 退出 python2 环境
$ conda deactivate
# 删除环境
$ conda remove -n env_name --all
# 查看当前存在的虚拟环境
$ conda env list
$ conda info -e
直接使用bioconda内的软件¶
部分编译比较复杂的软件,可以在bioconda内找到该软件,然后点击"Files",在里面下载编译好的软件,执行时如果有库缺失、GCC版本不够的报错,可以载入相应的库和GCC,此方式可以快速安装复杂软件。
docker¶
操作系统之上的虚拟层,提供独立于系统的软件环境;兴起于互联网行业,便于项目开发和交付部署,提高硬件资源利用率。
优点: 简单,对于复杂软件可以一键安装;无需安装任何依赖
缺点: 无法与作业调度软件结合使用;权限要求较高,多用户使用有风险
docker pull quay.io/qiime2/core:2021.8
singularity¶
HPC集群的容器工具,直接使用docker镜像。使用singularity搭建分析流程,可以在所有机器上运行。
优点: 简单;无需安装任何依赖;安全;可结合作业调度系统;高性能;适应性广
缺点: 软件较少;文件比较大
# 从给定的URL下载容器镜像,常用的有URL有Docker Hub(docker://user/image:tag) 和 Singularity Hub(shub://user/image:tag)
$ singularity pull tensorflow.sif docker://tensorflow/tensorflow:latest
# 在容器中执行某个命令
$ singularity exec /share/Singularity/saige_0.35.8.2.sif
# 进入容器
$ singularity shell /share/Singularity/ubuntu.sif
R包安装¶
# 从官方源安装,最常见方式
$ >install.packages("ggplot2")
# 同时安装多个包
$ >install.packages(c("broom", "clusterProfiler", "dorothea", "DOSE", "dplyr"))
# 指定安装源和安装路径
$ >install.packages("ggplot2", repos = "https://mirrors.ustc.edu.cn/CRAN/",lib="~/opt/Rlib")
# 使用Rscript,方便安装包报错时试错,不用每次进入R交互界面,然后又退出
$ Rscript -e 'install.packages(c("RcppArmadillo"), repos="https://mirrors.ustc.edu.cn/CRAN/")'
# 源码安装
$ R CMD INSTALL /path/rpackage.tar.gz
# 或
$ >install.packages("/path/rpackage.tar.gz", repos = NULL, type = "source")
# 安装指定版本的R包
$ >require(devtools)
$ >install_version("limma", version = "1.8.0")
# 或
$ >install.packages("https://cran.r-project.org/src/contrib/Archive/limma/limma_1.8.10.tar.gz", repos=NULL, type="source")
# bioconductor包安装
$ >install.packages("BiocManager")
$ >BiocManager::install("clusterProfiler")
# 指定安装位置和源
$ >options(BioC_mirror="https://mirrors.tuna.tsinghua.edu.cn/bioconductor")
$ >.libPaths(c("~/R/4.2/", .libPaths()))
$ >BiocManager::install("clusterProfiler")
# 也可以使用Rscript
$ Rscript -e 'BiocManager::install("clusterProfiler")'
https://bioconductor.org/packages/3.19/BiocViews.html#___Software
https://bioconductor.org/packages/3.19/bioc/html/XVector.html
# 测试包是否正常安装
$ >library(package)
# 其它包常见操作
# 卸载包
$ >remove.packages("package")
# 更新包
$ >update.packages("package")
# 查看R包安装位置
$ >.libPaths()
# 查看已安装包
$ >installed.packages()
# 查看包版本
$ >packageVersion("package")
# 查看包安装位置
$ >find.package("package")
$ >install.packages("pak")
$ >library(pak)
# 安装CRAN中的包
$ >pak:pak("ggplot2")
# 指定安装路径
$ >pak:pak("ggplot2", lib="PATH")
# 安装Bioconductor中的包
$ >pak::pak("clusterProfiler")
# 安装github上的包
$ >pak::pak("lchiffon/REmap")
# 使用URL
$ >pak::pkg("url::https://cran.r-project.org/src/contrib/Archive/tibble/tibble_3.1.7.tar.gz")
# 本地安装
# shell
$ wget https://cytotrace.stanford.edu/CytoTRACE_0.3.3.tar.gz
# 解压后为CytoTRACE
$ tar -xf CytoTRACE_0.3.3.tar.gz
# R
$ >pak::local_install("CytoTRACE")
# 或
$ >pkg_install("local::./CytoTRACE_0.3.3.tar.gz")
# 安装多个包
$ >pak::pkg(c("BiocNeighbors", "ComplexHeatmap", "circlize", "NMF"))
# 更新包
$ >pak::pkg_install("tibble")
# 更新包的所有依赖,默认不更新依赖
$ >pak::pkg("tibble", upgrade = TRUE)
# 重装包
$ >pak::pkg_install("tibble?reinstall")
# 卸载包
$ >pkg_remove("tibble")
perl包安装¶
# CPAN 模块自动安装
# 如果使用系统的cpan,则需要root权限,因此普通用户建议使用cpanm代替
$ cpan -i Bio::SeqIO
# cpanm 推荐
$ wget https://cpanmin.us -O capnm
$ cpanm --mirror http://mirrors.163.com/cpan --mirror-only Bio::SeqIO
# cpanm 指定安装目录
$ cpanm --mirror http://mirrors.163.com/cpan --mirror-only -l /path/to/perl/5.34.0/ Bio::SeqIO
# 源码安装
$ tar xvzf BioPerl-1.7.5.tar.gz
$ cd BioPerl-1.7.5
$ perl Makefile.PL (PREFIX=/home/opt/perl_modules)
$ make && make install
# 添加环境变量
$ export PERL5LIB=$PERL5LIB:/home/opt/perl_modules/lib/site_perl #或者把该行内容添加到 ~/.bashrc
# 测试perl模块安装正常
$ perl -MBio::SeqIO -e1 或 perldoc Bio::SeqIO
#如下报错,可能是perl版本冲突导致,即安装perl包的版本和使用perl包的版本不一致,建议将~/.bashrc perl相关的部分注释,然后再测试
perl: symbol lookup error: perl5/lib/perl5/x86_64-linux-thread-multi/auto/Cwd/Cwd.so: undefined symbol
python包安装¶
python 源码编译
其中 --enable-loadable-sqlite-extensions 的作用是防止部分应用使用 sqlite3 包时出现报错 No module named '_sqlite3',需要系统安装有 sqlite-devel。
$ tar xf Python-3.11.10.tgz
$ cd Python-3.11.10
$ ./configure --enable-optimizations --enable-loadable-sqlite-extensions --prefix=/path/to/python/
$ make -j20 && make install
# conda
$ conda install biopython
# pip
$ pip install --prefix=/home/opt/ppython_modules/ biopython 或 pip install --user biopython
# 使用国内源
$ pip install --prefix=/home/opt/ppython_modules/ -i https://pypi.tuna.tsinghua.edu.cn/simple biopython
# requirements.txt文件
$ pip install -r requirements.txt --prefix=/home/opt/ppython_modules/
# 源码
$ git clone https://github.com/madmaze/pytesseract.git
$ python setup.py install --prefix=/home/opt/ppython_modules或 pip install . --prefix=$PREFIX_PATH
# 测试python模块安装正常
$ python -c "import Bio"
# 添加环境变量
$ export PYTHONPATH=$PREFIX_PATH:/home/opt/python_modules/lib/python2.7/site-packages/ #或者加到 ~/.bashrc
常用包的所有 whl 文件 https://pypi.tuna.tsinghua.edu.cn/simple/numpy/
centos7中高版本的python,使用pip安装包时可能出现报错 pip is configured with locations that require TLS/SSL, however the ssl module in Python is not available,最快捷的解决办法为:
$ pip3 install pymysql -i http://mirrors.aliyun.com/pypi/simple --trusted-host mirrors.aliyun.com
注意事项¶
-
生信绝大部分软件都可以使用普通用户安装,不需要root权限。普通用户无法在集群上使用yum apt等安装软件;
-
软件安装过程中出现libxx库文件缺失的问题,一般都可以找到相应的源码,编译安装,设置环境变量即可;
-
避免使用conda一键安装软件,时间长了会导致各种环境问题,直至所有软件不可用,推到重来;
-
软件安装或使用过程中出现问题,最好将~/.bashrc中无关部分都临时注释掉,避免其它软件的影响;
-
使用软件前看一下集群公共软件库,尽量使用公共软件,一个软件一个环境,出现问题比较好排查;
介绍¶
毕昇编译器是华为编译器实验室针对鲲鹏等通用处理器架构场景,打造的一款高性能、高可信及易扩展的编译器工具链,增强和引入了多种编译优化技术,支持C/C++/Fortran等编程语言及对应编程语言的OpenMP扩展。
华为高性能通信库(Hyper MPI,hmpi)是整个高性能计算解决方案的关键组件,它实现了并行计算的网络通讯功能,可以用来支持制造、气象、分子动力学等应用场景。
集群上配置了华为毕昇编译器以及Hyper MPI,使用以下命令加载。
module purge
source /share/software/HPCKit/latest/setvars.sh
module use /share/software/HPCKit/latest/modulefiles
module load bisheng/compiler/bishengmodule bisheng/hmpi/hmpi
或
module load arm/hyper-mpi/24.6.30_bisheng
$ mpicc -show
clang -I/share/software/HPCKit/24.6.30/hmpi/bisheng/hmpi/include -I/share/software/HPCKit/24.6.30/hmpi/bisheng/hmpi/include/openmpi -I/share/software/HPCKit/24.6.30/hmpi/bisheng/hmpi/include/openmpi/opal/mca/hwloc/hwloc201/hwloc/include -I/share/software/HPCKit/24.6.30/hmpi/bisheng/hmpi/include/openmpi/opal/mca/event/libevent2022/libevent -I/share/software/HPCKit/24.6.30/hmpi/bisheng/hmpi/include/openmpi/opal/mca/event/libevent2022/libevent/include -L/share/software/HPCKit/24.6.30/hmpi/bisheng/hmpi/lib -Wl,-rpath -Wl,/share/software/HPCKit/24.6.30/hmpi/bisheng/hmpi/lib -Wl,--enable-new-dtags -lmpi
编译 lammps¶
这里以 lammps 编译安装为例,介绍毕昇编译器以及Hyper MPI的使用。
加载编译环境¶
module load arm/hyper-mpi/24.6.30_bisheng
编译配置 fftw¶
安装 fftw,样例安装目录:/path/to/FFTW
wget http://www.fftw.org/fftw-3.3.8.tar.gz
tar -xvf fftw-3.3.8.tar.gz
cd fftw-3.3.8
export CC=`which clang`
export CXX=`which clang++`
export FC=`which flang`
./configure --prefix=/path/to/FFTW --enable-shared --enable-static --enable-fma --enable-neon
make -j 96
make install
配置fftw环境变量
export PATH=/path/to/FFTW/bin:$PATH
export LD_LIBRARY_PATH=/path/to/FFTW/lib:$LD_LIBRARY_PATH
编译 lammps¶
下载lammps源码包:https://www.lammps.org/download.html
tar -xzf lammps-stable.tar.gz
cd lammps-29Aug2024/src
MAKE/Makefile.mpi 文件,修改CCFLAGS、LINKFLAGS、FFT_INC、FFT_PATH、FFT_LIB 这5行的内容:
CC = mpicxx
CCFLAGS = -g -O3 -I/path/to/BISHENG/include/c++/v1 -L/path/to/BISHENG/lib -std=c++11 -stdlib=libc++ -lc++ -lc++abi
SHFLAGS = -fPIC
DEPFLAGS = -M
LINK = mpicxx
LINKFLAGS = -g -O -L/path/to/BISHENG/lib -std=c++11 -stdlib=libc++ -lc++ -lc++abi
LIB =
SIZE = size
……
ARCHIVE = ar
ARFLAGS = -rc
SHLIBFLAGS = -shared
FFT_INC = -DFFT_FFTW -I/path/to/FFTW/include
FFT_PATH = -L/path/to/FFTW/lib
FFT_LIB = -lfftw3
make yes-KSPACE
make yes-MPIIO
make yes-MOLECULE
make yes-MANYBODY
make mpi -j
编译 lapack¶
加载编译环境¶
module load arm/hyper-mpi/24.6.30_bisheng
编译 lapack¶
tar xf lapack-3.12.0.tar.gz
cd lapack-3.12.0
cp INSTALL/make.inc.gfortran make.inc
将 make.inc 中的 CC = gcc 改为 CC = clang,FC = gfortran 改为 FC = flang。
TIMER = INT_ETIME 注释掉,# TIMER = INT_CPU_TIME 取消注释,否则编译报错 etime is not an intrinsic function。
修改后的 make.inc 如下所示。
####################################################################
# LAPACK make include file. #
####################################################################
SHELL = /bin/sh
# CC is the C compiler, normally invoked with options CFLAGS.
#
CC = clang
CFLAGS = -O3
# Modify the FC and FFLAGS definitions to the desired compiler
# and desired compiler options for your machine. NOOPT refers to
# the compiler options desired when NO OPTIMIZATION is selected.
#
# Note: During a regular execution, LAPACK might create NaN and Inf
# and handle these quantities appropriately. As a consequence, one
# should not compile LAPACK with flags such as -ffpe-trap=overflow.
#
FC = flang
FFLAGS = -O2 -frecursive
FFLAGS_DRV = $(FFLAGS)
FFLAGS_NOOPT = -O0 -frecursive
# Define LDFLAGS to the desired linker options for your machine.
#
LDFLAGS =
# The archiver and the flag(s) to use when building an archive
# (library). If your system has no ranlib, set RANLIB = echo.
#
AR = ar
ARFLAGS = cr
RANLIB = ranlib
# Timer for the SECOND and DSECND routines
#
# Default: SECOND and DSECND will use a call to the
# EXTERNAL FUNCTION ETIME
#TIMER = EXT_ETIME
# For RS6K: SECOND and DSECND will use a call to the
# EXTERNAL FUNCTION ETIME_
#TIMER = EXT_ETIME_
# For gfortran compiler: SECOND and DSECND will use a call to the
# INTERNAL FUNCTION ETIME
#TIMER = INT_ETIME
# If your Fortran compiler does not provide etime (like Nag Fortran
# Compiler, etc...) SECOND and DSECND will use a call to the
# INTERNAL FUNCTION CPU_TIME
TIMER = INT_CPU_TIME
# If none of these work, you can use the NONE value.
# In that case, SECOND and DSECND will always return 0.
#TIMER = NONE
...
BLASLIB = $(TOPSRCDIR)/librefblas.a
CBLASLIB = $(TOPSRCDIR)/libcblas.a
LAPACKLIB = $(TOPSRCDIR)/liblapack.a
TMGLIB = $(TOPSRCDIR)/libtmglib.a
LAPACKELIB = $(TOPSRCDIR)/liblapacke.a
# DOCUMENTATION DIRECTORY
# If you generate html pages (make html), documentation will be placed in $(DOCSDIR)/explore-html
# If you generate man pages (make man), documentation will be placed in $(DOCSDIR)/man
DOCSDIR = $(TOPSRCDIR)/DOCS
编译 make -j20 lapacklib。
如编译有报错,修改后 make clean再编译。
参考
x86移植到鲲鹏常见编译脚本、编译选项移植、builtin函数、内联汇编函数替换汇总
在GCC 9.1.0版本,支持了鲲鹏处理器所兼容的ARM v8指令集、TaiShan v110流水线。
使用方法:
在GCC for openEuler编译器、毕昇编译器、GCC编译器高于9.1.0版本上,并在CFLAGS、CPPFLAGS里增加编译选项:
-mtune=tsv110 -march=armv8-a
https://hpc.ilri.cgiar.org/list-of-software
http://ftp.genek.cn:8888/Share/linux_software/
常见报错¶
-
c++: error: unrecognized command-line option ‘-mbmi2’`-mbmi2` 是一个 GCC 编译器的命令行选项,用于启用针对支持 BMI2(Bit Manipulation Instruction Set 2)的处理器架构的优化。BMI2 是 Intel 和 AMD 处理器的一组指令集扩展,旨在提高位操作的性能。 解决:移除该编译选项 -
c++: error: unrecognized command-line option ‘-mpopcnt’-mpopcnt 是 GCC 编译器的一个选项,用于启用对 POPCNT 指令的支持。POPCNT 是一种位操作指令,用于快速计算一个二进制数中“1”的个数,通常可用于优化与位操作相关的算法。 解决:移除该编译选项
公共软件安装规范¶
-
慎用 root 安装各类依赖库、python 包、perl 模块等,以免导致各节点环境不一致、环境混乱,也没法做版本管理。root 非必要不使用。
-
zlib、gsl、zstd、xz、hdf5等各类依赖均使可使用普通账号源码编译,然后写好modulefile,使用时module载入即可。 -
公共环境禁止使用
conda安装任何软件,如果无法避免,则在singularity镜像中使用conda安装所需的软件,用户调用该镜像即可。 -
perl 所需的模块可以安装在 module 的 perl 中,基准版本为
arm/perl/5.34.0。 -
R 包可以安装在 module 的 R 中,基本版本为
arm/r/4.4.1。 -
由于 python 包更新频繁、各个软件依赖的 python 版本区别较大,因此每个应用软件依赖的 python 包均应该安装在该软件目录内(使用
--prefix指定安装目录),而不是在系统或公共 python 目录内,modulefile文件中使用PYTHONPATH环境变量指定路径来引用安装的包。 -
modulefile文件命名均采用小写字母,所有依赖 python 的软件,其modulefile文件名中均应指定 python 版本,如arm/rnaframework/2.9.2-py3.11arm/ntsynt/1.0.2-py3.9arm/snpbinner/1.0.0-py2.7;使用了 MPI 的软件,modulefile文件命名中指定使用了哪个MPI,如arm/gflow/0.1.7-openmpiarm/gromacs/2019.5-hmpi等。 -
每个软件的
modulefile文件中均应包含软件的项目地址,github地址优先,方便跟踪。 -
所有软件源码在 package 目录内均应单独建目录,然后在该目录内下载该软件的各种版本、进行编译等,所有软件源码的
tar.gz源码包尽量保留。 -
软件中若有头文件(
xxx.h),应使用CPATH指定路径。 -
软件中若有静态链接库(
xxx.a),应使用LIBRARY_PATH指定路径。 -
软件中若有动态链接库(
xxx.so),应使用LD_LIBRARY_PATH指定路径。 -
软件中若有 pkgconfig 文件(
xxx.pc),应使用PKG_CONFIG_PATH指定路径。 -
软件中若有 cmake 模块文件(
xxx.cmake),应使用CMAKE_PREFIX_PATH指定路径。 -
java 写的 jar 软件,如果没有调用脚本,可以按如下方式编写调用脚本以方便使用,以 trimmomatic 为例
使用#!/bin/bash SCRIPT_DIR="$(cd "$(dirname "${BASH_SOURCE[0]}")" && pwd)" JAR_FILE="${SCRIPT_DIR}/trimmomatic-0.39.jar" if [[ ! -f "$JAR_FILE" ]]; then echo "错误:找不到 $JARAR_FILE" exit 1 fi java -jar "$JAR_FILE" "$@"$ trimmomatic.sh Usage: PE [-version] [-threads <threads>] [-phred33|-phred64] [-trimlog <trimLogFile>] [-summary <statsSummaryFile>] [-quiet] [-validatePairs] [-basein <inputBase> | <inputFile1> <inputFile2>] [-baseout <outputBase> | <outputFile1P> <outputFile1U> <outputFile2P> <outputFile2U>] <trimmer1>... or: SE [-version] [-threads <threads>] [-phred33|-phred64] [-trimlog <trimLogFile>] [-summary <statsSummaryFile>] [-quiet] <inputFile> <outputFile> <trimmer1>... or: -version
静态编译¶
参考:
$ ./configure
checking for gawk... gawk
checking for gcc... gcc
checking whether the C compiler works... no
configure: error: in '/share/home/software/package/samtools/samtools-1.22.1':
configure: error: C compiler cannot create executables
See 'config.log' for more details
# 解决
yum install glibc-static libxcrypt-static libstdc++-static
在 rocky8 上分别编译动态 samtools 和静态 samtools,然后在 centos7 上运行测试。
可以看到静态的 samtools 文件较大,但可以正常运行,动态的 samtools 文件较小,但依赖高版本的 glibc。
$ ldd samtools_static
not a dynamic executable
$ ldd samtools_dynamic
./samtools_d2: /lib64/libm.so.6: version 'GLIBC_2.23' not found (required by ./samtools_d2)
linux-vdso.so.1 => (0x00007fff33b8a000)
/usr/local/bin/snoopy/lib/libsnoopy.so (0x00007fe3ca389000)
libpthread.so.0 => /lib64/libpthread.so.0 (0x00007fe3ca16d000)
libz.so.1 => /lib64/libz.so.1 (0x00007fe3c9f57000)
libm.so.6 => /lib64/libm.so.6 (0x00007fe3c9c55000)
libbz2.so.1 => /lib64/libbz2.so.1 (0x00007fe3c9a45000)
liblzma.so.5 => /lib64/liblzma.so.5 (0x00007fe3c981f000)
...
libcrypto.so.10 => /lib64/libcrypto.so.10 (0x00007fe3c66ca000)
librt.so.1 => /lib64/librt.so.1 (0x00007fe3c64c2000)
libkrb5support.so.0 => /lib64/libkrb5support.so.0 (0x00007fe3c62b2000)
libkeyutils.so.1 => /lib64/libkeyutils.so.1 (0x00007fe3c60ae000)
libresolv.so.2 => /lib64/libresolv.so.2 (0x00007fe3c5e95000)
libsasl2.so.3 => /lib64/libsasl2.so.3 (0x00007fe3c5c78000)
libselinux.so.1 => /lib64/libselinux.so.1 (0x00007fe3c5a51000)
libcrypt.so.1 => /lib64/libcrypt.so.1 (0x00007fe3c581a000)
libpcre.so.1 => /lib64/libpcre.so.1 (0x00007fe3c55b8000)
libfreebl3.so => /lib64/libfreebl3.so (0x00007fe3c53b5000)
$ ll -h
-rwxr-xr-x 1 software software 14M Sep 21 00:49 samtools_static
-rwxr-xr-x 1 software software 9.2M Sep 21 00:13 samtools_dynamic
$ ./samtools_dynamic
./samtools_d2: /lib64/libm.so.6: version 'GLIBC_2.23' not found (required by ./samtools_d2)
$ ./samtools_static --version
samtools 1.22.1
Using htslib 1.22.1
Copyright (C) 2025 Genome Research Ltd.
Samtools compilation details:
Features: build=configure curses=no
CC: gcc
CPPFLAGS:
CFLAGS: -Wall -g -O2
LDFLAGS: -static
HTSDIR: htslib-1.22.1
LIBS:
CURSES_LIB:
HTSlib compilation details:
Features: build=configure libcurl=no S3=no GCS=no libdeflate=no lzma=yes bzip2=no plugins=no htscodecs=1.6.4
CC: gcc
CPPFLAGS:
CFLAGS: -Wall -g -O2 -fvisibility=hidden
LDFLAGS: -fvisibility=hidden
HTSlib URL scheme handlers present:
built-in: file, data, preload
crypt4gh-needed: crypt4gh
mem: mem
autotools¶
一、编译阶段(生成 .o)
| 选项 / 变量 | 作用 | 典型值 |
|---|---|---|
CFLAGS / CXXFLAGS |
让代码本身与位置无关(后续可能进 .so)或强制静态 | -static -fPIC |
-static |
告诉驱动器 “后续只找静态库” | gcc -static |
-fPIC / -fPIE |
生成位置无关代码(必须 若最终要进 .so) | -fPIC |
-ffunction-sections -fdata-sections |
减小编出静态库体积 | |
CPPFLAGS |
头文件搜索路径 | -I/opt/static/include |
二、链接阶段(生成 可执行文件 / .so / .a)
| 选项 / 变量 | 作用 | 示例 |
|---|---|---|
LDFLAGS |
链接器旗标 + 库搜索路径 | -L/opt/static/lib -static |
-static |
生成 纯静态 ELF(无 .dynamic 段) | gcc -static file.o -o file |
-Wl,-Bstatic |
区间内 只链静态库 | -Wl,-Bstatic -lz -lssl -Wl,-Bdynamic |
-Wl,-Bdynamic |
恢复默认动态链接(与上成对) | 同上 |
-Wl,--whole-archive |
把静态库里 所有 .o 都拉进来(解决循环依赖) | -Wl,--whole-archive -lmylib -Wl,--no-whole-archive |
-Wl,-soname=… |
控制 .so 名字(动态库专用) | |
LIBS |
要链的 具体库简称 | -lreadline -lncursesw |
LIBRARY_PATH |
gcc 驱动搜索静态/动态库路径(链接时) | export LIBRARY_PATH=/opt/static/lib |
LD_LIBRARY_PATH |
运行时搜索 .so(对静态链接无意义) |
三、ar / ranlib(打包静态库)
| 工具 | 变量 | 默认 | 说明 |
|---|---|---|---|
ar |
AR / ARFLAGS |
ar cr |
创建 .a |
ranlib |
RANLIB |
ranlib |
生成索引,加快链接 |
四、configure 常见开关
| 选项 | 含义 |
|---|---|
--enable-static |
把 软件包自己编出静态库(如 libxxx.a) |
--disable-shared / --enable-shared=no |
禁止生成 .so |
--enable-R-static-lib(R 专属) |
让 R 把 blas/lapack 等静态链进可执行文件 |
--with-static-the-system |
少数包提供,等价于 -static 全套 |
五、静态链接“找不到符号”速查
| 症状 | 检查项 |
|---|---|
ld: cannot find -lxxx |
1. 真没 libxxx.a2. 没加 -L/path |
relocation against … can not be used |
库未用 -fPIC 重编(要进 .so 时) |
undefined reference to symbol` |
顺序错,被依赖的放右边;或用 --whole-archive |
一键静态
export CC="gcc -static"
export CPPFLAGS="-I/opt/static/include"
export LDFLAGS="-L/opt/static/lib -static"
export LIBS="-lssl -lcrypto -lz -ldl -pthread"
./configure --disable-shared --enable-static
make
libtool¶
在编译二进制文件时,在 XXX_LINK中$(CCLD)后面添加-all-static即可起效。一般的LDFLAG、CFLAG等对libtool无效
-static` 的方式测试无效
iperf3_LINK = $(LIBTOOL) $(AM_V_lt) --tag=CC $(AM_LIBTOOLFLAGS) \
$(LIBTOOLFLAGS) --mode=link $(CCLD) -all-static $(iperf3_CFLAGS) $(CFLAGS) \
$(iperf3_LDFLAGS) $(LDFLAGS) -o $@
LTCOMPILE = $(LIBTOOL) $(AM_V_lt) --tag=CC $(AM_LIBTOOLFLAGS) \
$(LIBTOOLFLAGS) --mode=compile $(CC) -static $(DEFS) \
$(DEFAULT_INCLUDES) $(INCLUDES) $(AM_CPPFLAGS) $(CPPFLAGS) \
$(AM_CFLAGS) $(CFLAGS)
libtool looks at the parameters of gcc, so you should have something like below
$ cat Makefile
all: libone libtwo
rm *.o
@libtool --mode=link gcc -all-static -o libcombo.a libone.a libtwo.a
libone: one.c
@libtool --mode=compile gcc -c one.c -o one.lo
@libtool --mode=link gcc -static -o libone.a one.lo
libtwo: two.c
@libtool --mode=compile gcc -c two.c -o two.lo
@libtool --mode=link gcc -static -o libtwo.a two.lo
cmake¶
一、控制「库」本身的类型
| 变量 / 指令 | 作用 | 典型值 |
|---|---|---|
BUILD_SHARED_LIBS |
全局开关,ON=动态/OFF=静态 |
-DBUILD_SHARED_LIBS=OFF |
add_library(foo STATIC …) |
显式声明 静态库 | |
add_library(foo SHARED …) |
显式声明 动态库 | |
add_library(foo …) 无参数 |
由 BUILD_SHARED_LIBS 决定 |
二、编译阶段(生成 .o)
| 变量 | 说明 | 示例 |
|---|---|---|
CMAKE_C_FLAGS / CMAKE_CXX_FLAGS |
给 所有目标 加选项 | -static -fPIC |
target_compile_options(foo PRIVATE -fPIC) |
只给指定目标加 | |
add_compile_options(-fPIC) |
全局生效 |
若要把静态库 再链进动态库,静态库本身必须带 -fPIC,否则链接时报 can not be used when making a shared object; recompile with -fPIC
三、链接阶段(生成 可执行文件 / .so / .a)
| 变量 | 说明 | 示例 |
|---|---|---|
CMAKE_EXE_LINKER_FLAGS |
可执行文件 链接旗标 | -static |
CMAKE_SHARED_LINKER_FLAGS |
动态库 链接旗标 | |
CMAKE_STATIC_LINKER_FLAGS |
静态库 链接旗标(很少用) | |
target_link_libraries(foo PRIVATE -static) |
对单个目标加 -static |
|
target_link_libraries(foo PRIVATE -Wl,-Bstatic -lz -Wl,-Bdynamic) |
区间静态链接 |
纯静态可执行文件: set(CMAKE_EXE_LINKER_FLAGS "-static") 即可让 最终 ELF 无 .dynamic 段
四、查找 & 强制使用 静态库
| 命令 / 变量 | 说明 | 示例 |
|---|---|---|
find_package(ZLIB REQUIRED) |
先找到包 | |
set(ZLIB_USE_STATIC_LIBS ON) |
官方 Find 模块识别后只选 .a |
CMake ≥ 3.24 |
find_library(ZLIB_STATIC NAMES z libz PATHS /opt/static/lib) |
手动找 libz.a |
|
target_link_libraries(foo PRIVATE ${ZLIB_STATIC}) |
直接给 绝对路径 链入 |
一键静态
cmake -B build -G Ninja \
-DCMAKE_BUILD_TYPE=Release \
-DBUILD_SHARED_LIBS=OFF \
-DCMAKE_EXE_LINKER_FLAGS="-static" \
-DCMAKE_C_FLAGS="-fPIC" \
-DCMAKE_CXX_FLAGS="-fPIC" \
-DZLIB_USE_STATIC_LIBS=ON \
-DCMAKE_INSTALL_PREFIX=/opt/static
cmake --build build
anchorwave¶
项目地址 https://github.com/baoxingsong/anchorwave
git clone https://github.com/baoxingsong/anchorwave.git
cd anchorwave
mv CMakeLists_arm.txt CMakeLists.txt
cmake ./
make
Amber¶
项目地址 https://ambermd.org/GetAmber.php
参考文档 https://ambermd.org/doc12/Amber24.pdf
module load gcc/compiler/gccmodule gcc/hmpi/hmpi
tar xf Amber24.tar.bz2
tar xf AmberTools24.tar.bz2
pip install --prefix=/path/to/amber/ numpy scipy matplotlib
cd amber24_src
$ ./update_amber --update
cd build
run_cmake 文件中 cmake 的参数,然后再运行 run_cmake。
修改后如下,指定安装目录、启用 MPI、禁用 conda
cmake $AMBER_PREFIX/amber24_src \
-DCMAKE_INSTALL_PREFIX=/share/software/app/arm/amber/24/ \
-DCOMPILER=GNU \
-DMPI=TRUE -DCUDA=FALSE -DINSTALL_TESTS=TRUE \
-DDOWNLOAD_MINICONDA=FALSE \
2>&1 | tee cmake.log
run_cmake 运行完成后,再运行 make install 安装。
source /share/software/app/arm/amber/24/amber.sh 后即可使用。
AnnoPRO¶
https://github.com/idrblab/AnnoPRO
依赖库 profeatpy 在 github 上的源码包被删了,无法编译安装这个包。
apbs¶
项目地址
github.com/Electrostatics/apbs
https://apbs.readthedocs.io/en/latest/index.html
$ tar xf apbs-3.4.1.tar.gz
$ cd apbs-3.4.1
$ mkdir build && cd build
$ export APBS_BUILD_DIR=${PWD}
$ export SuiteSparse_DIR="/path/to/suitesparse/"
$ export CMAKE_PREFIX_PATH="/path/to/suitesparse/:$CMAKE_PREFIX_PATH"
$ cmake .. -DUMFPACK_INCLUDES=/path/to/suitesparse/include/suitesparse/umfpack.h -DUMFPACK_LIBRARIES=/path/to/suitesparse/lib64/libumfpack.a -DAMD_INCLUDE_DIR=/path/to/suitesparse/include/suitesparse/ -DAMD_STATIC=/path/to/suitesparse/lib64/libamd.a -DPython3_EXECUTABLE=/usr/bin/python3 -DCMAKE_C_COMPILER=`which gcc` -DCMAKE_CXX_COMPILER=`which g++` -DCMAKE_C_FLAGS="-fopenmp" -DCMAKE_CXX_FLAGS="-fopenmp"
$ make -j
$ make install
Augustus¶
项目地址:https://github.com/Gaius-Augustus/Augustus/
参考 https://github.com/Gaius-Augustus/Augustus/blob/master/docs/INSTALL.md
$ wget http://bioinf.uni-greifswald.de/augustus/binaries/augustus.current.tar.gz
$ tar -xzf augustus.current.tar.gz
$ cd augustus
common.mk 文件,将 COMPGENEPRED = true 更改为 COMPGENEPRED = false,添加 MYSQL = false
编译 make augustus
如果需要编译 augustus 及其它相关工具。
$ module load arm/bamtools/2.5.2 arm/htslib/1.21
$ tar -xzf augustus.current.tar.gz
$ cd augustus
$ make
ATLAS¶
$ mkdir build && cd build
$ ../configure --prefix=/path/to/atlas/ -Si archdef 0 -b 64 -D c -DATL_ARM_HARDFP=1 --cripple-atlas-performance
# make -j 20 会报错
$ make && make install
# 查看安装的库
$ $ ls -1 /path/to/atlas/
libatlas.a
libcblas.a
libf77blas.a
liblapack.a
libptcblas.a
libptf77blas.a
BatMeth2¶
项目地址:https://github.com/GuoliangLi-HZAU/BatMeth2
$ git clone https://github.com/GuoliangLi-HZAU/BatMeth2.git
$ cp ~/package/sse2neon.h src/mealign/
$ sed -i.bak 's/<emmintrin.h>/"sse2neon.h"/g' ./src/mealign/ksw.c
$ sed -i.bak 's/-m64/ /g' ./src/Makefile
$ ./configure --prefix=/path/to/batmeth2/
$ make -j20 && make install
BLAT¶
最新版 https://hgdownload.soe.ucsc.edu/admin/,下载 jksrc.xxx.zip,BLAT 位于 kent/src/blat,不容易编译成功。
编译好的 x86 版本 https://hgdownload.soe.ucsc.edu/admin/exe/linux.x86_64/blat/
可编译版本
$ wget https://codeload.github.com/djhshih/blat/tar.gz/v35.1
$ mv v35.1 blat_35.1.tar.gz
$ tar xf blat_35.1.tar.gz
$ cd blat-35.1/
$ make
# 或编译静态库版本
$ CFLAGS="-static -static-libgcc -D_STATIC" make
BerkeleyDB¶
项目地址 https://github.com/berkeleydb/libdb/
$ wget https://github.com/berkeleydb/libdb/releases/download/v5.3.28/db-5.3.28.tar.gz
$ tar xf db-5.3.28.tar.gz
$ cd db-5.3.28/build_unix
$ ../dist/configure --build=aarch64-unknown-linux-gnu --prefix=/path/to/berkeley-db/
$ make -j20 && make install
bmtagger¶
https://ftp.ncbi.nlm.nih.gov/pub/agarwala/bmtagger/
$ wget https://ftp.ncbi.nlm.nih.gov/pub/agarwala/bmtagger/bmtagger.sh
$ wget https://anaconda.org/bioconda/bmfilter/3.101/download/linux-aarch64/bmfilter-3.101-h78569d1_5.tar.bz2
$ wget https://anaconda.org/bioconda/srprism/2.4.24/download/linux-aarch64/srprism-2.4.24-h76f4f2e_5.tar.bz2
$ wget https://conda.anaconda.org/bioconda/linux-aarch64/extract_fullseq-3.101-h78569d1_5.tar.bz2
$ tar xf *bz2
$ ls -1 bin/
bmfilter
extract_fullseq
srprism
$ module load arm/blast/2.16.0
bmtagger.conf
#!/bin/sh
echo "bmtagger.sh: You may want to edit bmtagger.conf" >&2
SRPRISM=./bin/srprism
BMFILTER="./bin/bmfilter"
EXTRACT_FA="./bin/extract_fullseq"
#bmfiles="hs/quick_mask/indexes/washu_mask18"
#blastdb="hs/quick_mask/indexes/HUMAN_SCREENING"
#srindex="hs/quick_mask/indexes/HUMAN_SCREENING.srprism"
#TMPDIR="/tmp"
https://ftp.ncbi.nlm.nih.gov/pub/agarwala/bmtagger/bmtools.tar.gz 这个源码包可以独立编译 srprism bmfilter extract_fullseq 但有编译报错。
blast+¶
可以在 https://ftp.ncbi.nlm.nih.gov/blast/executables/blast+/LATEST/ 中下载编译好的版本。
$ wget https://ftp.ncbi.nlm.nih.gov/blast/executables/blast+/LATEST/ncbi-blast-2.16.0+-aarch64-linux.tar.gz
bwa-mem2¶
项目地址 https://github.com/bwa-mem2/bwa-mem2
# 下载二进制版本
wget https://anaconda.org/bioconda/bwa-mem2/2.2.1/download/linux-aarch64/bwa-mem2-2.2.1-h0010869_6.tar.bz2
cactus¶
项目地址 https://github.com/ComparativeGenomicsToolkit/cactus
git clone https://github.com/ComparativeGenomicsToolkit/cactus.git --recursive
cd cactus
setup.py 文件中22行的 subprocess.run([sys.executable, "-m", "pip", "install", "submodules/sonLib"], check=True) 注释掉,以免将 sonLib 安装到了 python 的默认目录。
make -j20
pip install --prefix=/path/to/cactus/ submodules/sonLib
pip install --prefix=/path/to/cactus/ .
pip install --prefix=/path/to/cactus/ -r toil-requirement.txt
cereal¶
https://github.com/USCiLab/cereal
unittests/CMakeLists.txt unittests/boost/CMakeLists.txt unittests/cpp17/CMakeLists.txt 中所有 -m32 删除
删除 CMakeLists.txt 中部分编译选项
add_compile_options(-Wall -Wextra -pedantic -Wshadow -Wold-style-cast) 更改为 add_compile_options(-Wall -Wextra -pedantic )
add_compile_options(-Werror) 更改为 add_compile_options()
$ mkdir build
$ cd build
$ cmake -DCMAKE_INSTALL_PREFIX=/path/to/cereal/ -DBoost_INCLUDE_DIR=/path/to/boost/include/ ..
$ make -j12
$ make install
circos¶
项目地址 https://circos.ca/software/download/circos/
$ wget https://circos.ca/distribution/circos-0.69-10.tgz
$ tar xf circos-0.69-10.tgz
$ cd circos-0.69-10
$ module load arm/perl/5.34.0
# 查看确实的 perl 模块
$ bin/circos -modules|grep missing
missing Font::TTF::Font
missing GD
missing GD::Polyline
missing Math::Bezier
missing Math::Round
missing Math::VecStat
missing Params::Validate
missing Regexp::Common
missing Set::IntSpan
missing Statistics::Basic
missing Text::Format
# 安装缺失的 perl 模块
$ cpanm -l /share/software/app/arm/perl/5.34.0/ Text::Format Statistics::Basic Set::IntSpan Regexp::Common Params::Validate Math::VecStat Math::Round Math::Bezier Font::TTF::Font
# 加载 libgd 用于安装 GD 模块,注意 libgd 版本需要低于 2.3,否则 GD 模块安装会报错
# 报错是高版本的 libgd 中没有 gdlib-config 导致的
$ module load arm/libgd/2.2.5
$ cpanm -l /share/software/app/arm/perl/5.34.0/ GD GD::Polyline
# 测试
$ bin/circos -conf /share/software/app/arm/circos/0.69-10/example/etc/circos.conf
clustalw¶
项目地址 http://www.clustal.org/download/current/
wget http://www.clustal.org/download/current/clustalw-2.1.tar.gz
tar xf clustalw-2.1.tar.gz
cd clustalw-2.1/
./configure --build=arm --prefix=/path/to/clustalw/
make -j20 && make install
cmake¶
直接下载编译好的 aarch64 版本 cmake-3.30.5-linux-aarch64.sh
cppunit¶
https://freedesktop.org/wiki/Software/cppunit/
$ tar xf cppunit-1.13.2.tar.gz
$ cd cppunit-1.13.2
$ ./configure --prefix=/path/to/cppunit/ --build=aarch64-unknown-linux-gnu
$ make -j20 && make install
bowtie2¶
直接下载编译好的二进制文件
https://github.com/BenLangmead/bowtie2/releases
EDTA (todo)¶
https://github.com/liaoherui/AnnoSINE_v2
mamba create -n EDTA2.2 -c conda-forge -c bioconda annosine2 biopython cd-hit coreutils genericrepeatfinder genometools-genometools glob2 tir-learner ltr_finder_parallel ltr_retriever mdust multiprocess muscle openjdk perl perl-text-soundex r-base r-dplyr regex repeatmodeler r-ggplot2 r-here r-tidyr tesorter samtools bedtools LTR_HARVEST_parallel HelitronScanner
glob2
https://github.com/lutianyu2001/TIR-Learner
elai¶
项目地址 https://github.com/haplotype/ELAI
$ git clone https://github.com/haplotype/ELAI
$ cd src
修改 Makefile 文件
CFLAGS += -Wall -fno-math-errno -ffast-math 修改为 CFLAGS += -Wall -fno-math-errno -ffast-math -fsigned-char,即添加 -fsigned-char 编译选项
STATICLIBS += /usr/local/lib/libgsl.a /usr/local/lib/libgslcblas.a 修改为 STATICLIBS += /share/software/app/arm/gsl/2.7/lib/libgsl.a /share/software/app/arm/gsl/2.7/lib/libgslcblas.a
最后编译 make。
Warning
编译时不添加 -fsigned-char 编译选项也能编译成功,但运行不出结果。
测试
cd ../example
$ ../src/elai -g hap.ceu.chr22.inp -p 10 -g hap.yri.chr22.inp -p 11 -g admix-1cm.inp -p 1 -pos hgdp.chr22.pos -o test -s 20 --exclude-nopos --exclude-maf 0.01 --exclude-miss1 -mg 20
emboss¶
项目地址 https://emboss.sourceforge.net/
$ wget https://anaconda.org/bioconda/emboss/6.6.0/download/linux-aarch64/emboss-6.6.0-h70f19aa_11.tar.bz2
eggnog-mapper¶
项目地址 https://github.com/eggnogdb/eggnog-mapper
# 2.1.13
pip install --prefix=/path/to/eggnog-mapper eggnog-mapper
# eggnog-mapper 安装包自带了这几个软件的预编译版,但都是 x86 的,使用时需要载入 arm 版
# eggnog-mapper 会优先使用 $PATH 中有的软件,然后再使用自己安装的
$ module load arm/diamond/2.1.10 arm/prodigal/2.6.3 arm/mmseqs2/16-747c6 arm/hmmer/3.4
$ emapper.py -h
下载数据库
更改代码
将 download_eggnog_data.py 中 BASE_URL = f'http://eggnogdb.embl.de/download/emapperdb-{__DB_VERSION__}' 改为 BASE_URL = f'http://eggnog5.embl.de/download/emapperdb-{__DB_VERSION__}'。
# 2759 是真核数据库,一路 Y 就可以了
$ download_eggnog_data.py -F -f -P -M -H -d 2759
EVM¶
项目地址 https://github.com/EVidenceModeler/EVidenceModeler/
$ tar xf EVidenceModeler-v2.1.0.tar.gz
$ cd EVidenceModeler-v2.1.0
plugins/ParaFly/configure 中第 3123 行的 AM_CXXFLAGS=-m64 注释掉。
$ make
exonerate¶
下载二进制版本
$ wget https://anaconda.org/bioconda/exonerate/2.4.0/download/linux-aarch64/exonerate-2.4.0-he4d9ad3_8.tar.bz2
fastlmm¶
需要替换 Cpp/Inc/BlasLapack.h 中数学库,源码中使用的 intel MKL 数学库。
| intel MKL | openblas | KML |
|---|---|---|
| sscal | cblas_sscal | cblas_sscal |
| dscal | cblas_dscal | cblas_dscal |
| scopy | cblas_scopy | cblas_scopy |
| dcopy | cblas_dcopy | cblas_dcopy |
| sgemv | cblas_sgemv | cblas_sgemv |
| dgemv | cblas_dgemv | cblas_dgemv |
| sgemm | cblas_sgemm | cblas_sgemm |
| dgemm | cblas_dgemm | cblas_dgemm |
| ssyrk | cblas_ssyrk | cblas_ssyrk |
| dsyrk | cblas_dsyrk | cblas_dsyrk |
| spotrf | LAPACKE_spotrf | spotrf_ |
| dpotrf | LAPACKE_dpotrf | dpotrf_ |
| spotrs | LAPACKE_spotrs | spotrs_ |
| dpotrs | LAPACKE_dpotrs | dpotrs_ |
| sgesdd | LAPACKE_sgesdd | sgesdd_ |
| dgesdd | LAPACKE_dgesdd | dgesdd_ |
| ssyevr | LAPACKE_ssyevr | ssyevd_ |
| dsyevr | LAPACKE_dsyevr | dsyevd_ |
| sgelsy | LAPACKE_sgelsy | sgelsy_ |
| dgelsy | LAPACKE_dgelsy | dgelsy_ |
| vsLn | for (BLAS_INT i = 0; i < n; ++i) | vslog2 |
| vdLn | vdlog2 | |
| vsSqrt | for (BLAS_INT i = 0; i < n; ++i) | vssqrt |
| vsSqrt | vdsqrt |
fastqc¶
https://anaconda.org/bioconda/fastqc
直接下载编译好的二进制文件
$ wget https://anaconda.org/bioconda/fastqc/0.12.1/download/noarch/fastqc-0.12.1-hdfd78af_0.tar.bz2
fastp¶
安装依赖库 isa-l,libdeflate,如果没有 libstdc++.a,也需要安装 gcc。最后使用静态库编译fastp,使用时不用另外载入依赖库。
# 设置静态库地址
$ export LIBRARY_PATH=/path/to/isa-l/lib/:/path/to/libdeflate/lib/:/path/to/gcc/lib64/
$ git clone https://github.com/OpenGene/fastp.git
$ cd fastp
$ make -j12 static
fastStructure¶
- python3
项目地址 https://github.com/rajanil/fastStructure/issues/63
原项目基于python2,安装 scipy 出现报错。
因此实际编译安装使用python3的版本 https://github.com/jashapiro/fastStructure
git clone https://github.com/jashapiro/fastStructure.git
cd fastStructure
git checkout py3
部分代码需要更改
parse_bed.pyx 中
25 行的Nbytes = Nindiv/4+(Nindiv%4>0)*1 更改为 Nbytes = Nindiv//4+(Nindiv%4>0)*1
48 行的 print "This is not a valid bed file" 更改为 print("This is not a valid bed file")
fastStructure.pyx 中
305 行的 print "Failed" 更改为 print("Failed")
$ module load arm/gsl/2.4
$ pip install --prefix=/path/to/faststructure/ numpy scipy cython
$ cd var
$ PYTHONPATH=/path/to/faststructure/1.0/lib64/python3.9/site-packages/:/path/to/faststructure/1.0/lib/python3.9/site-packages/ python setup.py build_ext -f --inplace
$ cd ..
$ PYTHONPATH=/path/to/faststructure/1.0/lib64/python3.9/site-packages/:/path/to/faststructure/1.0/lib/python3.9/site-packages/ python setup.py build_ext -f --inplace
# 测试,将 ./var/ 加入 PYTHONPATH,否则出现 allelefreq 相关报错
$ PYTHONPATH=/path/to/faststructure/1.0/lib64/python3.9/site-packages/:/path/to/faststructure/1.0/lib/python3.9/site-packages/:$PWD python structure.py -h
运行测试算例
PYTHONPATH=//path/to/faststructure/faststructure/1.0/lib64/python3.9/site-packages/://path/to/faststructure/faststructure/1.0/lib/python3.9/site-packages/ python structure.py -K 3 --input=test/testdata --output=testoutput_simple --full --seed=100
出现报错,暂时未能解决。
Traceback (most recent call last):
File "//path/to/faststructure/fastStructure/structure.py", line 176, in <module>
Q, P, other = fastStructure.infer_variational_parameters(G, params['K'], \
File "fastStructure.pyx", line 99, in fastStructure.infer_variational_parameters
psi.update(G, pi)
TypeError: Cannot convert allelefreq.AlleleFreq to allelefreq.AlleleFreq
参考
https://github.com/rajanil/fastStructure/issues/63
https://github.com/rajanil/fastStructure/issues/81
- python2
$ module load arm/python/2.7.18
$ git clone https://github.com/rajanil/fastStructure/
$ cd fastStructure
$ pip install --prefix=/path/to/fastStructure/ numpy
# 设置编译参数 -fallow-argument-mismatch,否则 scipy 编译过程中出现报错 Error: Rank mismatch between actual argument at (1) and actual argument at (2) (scalar and rank-1)
$ export FCFLAGS="-fallow-argument-mismatch"
$ export FFLAGS="-fallow-argument-mismatch"
$ PYTHONPATH=/path/to/lib/python2.7/site-packages/ pip install --prefix=/path/to/fastStructure/ scipy Cython matplotlib
parse_bed.pyx 和 fastStructure.pyx 顶部添加 # cython: language_level=2 表示使用 python2 编译 cpython
$ cd vars
$ PYTHONPATH=/path/to/lib/python2.7/site-packages/ python setup.py build_ext -f --inplace
$ cd ..
$ PYTHONPATH=/path/to/lib/python2.7/site-packages/ python setup.py build_ext -f --inplace
# 测试
$ PYTHONPATH=/path/to/lib/python2.7/site-packages/:$PWD/vars/ python structure.py -h
PYTHONPATH=//path/to/faststructure/faststructure/1.0/lib/python2.7/site-packages/ python structure.py -K 3 --input=test/testdata --output=testoutput_simple --full --seed=100
FastTree¶
$ wget http://meta.microbesonline.org/fasttree/FastTree.c
$ gcc -DOPENMP -DUSE_DOUBLE -fopenmp -O3 -march=armv8.2-a -mtune=tsv110 -finline-functions -funroll-loops -Wall -o FastTreePar-2.1.11 FastTree.c -lm
$ export OMP_NUM_THREADS=128
fftw¶
参考
$ module load arm/openmpi/5.0.5
$ wget wget https://fftw.org/pub/fftw/fftw-3.3.8.tar.gz
$ tar xf fftw-3.3.8.tar.gz
$ cd fftw-3.3.8/
$ ./configure --prefix=/path/to/fftw/ --enable-single --enable-float --enable-neon --enable-shared --enable-threads --enable-openmp --enable-mpi CFLAGS="-O3 -fomit-frame-pointer -fstrict-aliasing"
$ make -j20
$ make install
flye¶
项目地址 https://github.com/mikolmogorov/Flye/
wget wget https://anaconda.org/bioconda/flye/2.9.5/download/linux-aarch64/flye-2.9.5-py39h989d1c4_2.tar.bz2
tar xf flye-2.9.5-py39h989d1c4_2.tar.bz2
GATK¶
intel GKL
# 下载这个 arm64 分支
git clone -b devel https://github.com/syalavarthi/GKL.git
cd GKL
gradle-5.6-bin.zip,将其再放在 /share/home/software/.gradle/wrapper/dists/gradle-5.6-bin/gradle-5.6-bin.zip,将 gradle/wrapper/gradle-wrapper.properties 中的 distributionUrl=https\://services.gradle.org/distributions/gradle-3.2.1-all.zip 替换为 distributionUrl=file:/share/home/software/.gradle/wrapper/dists/gradle-5.6-bin/gradle-5.6-bin.zip。
运行 ./gradlew build
git clone https://github.com/IntelLabs/GKL.git
gcta¶
项目地址 https://github.com/jianyangqt/gcta
注意 openblas 的版本,较高的版本中部分 API 有变化(函数 dpotri_)会导致编译报错,测试 0.3.13 可正常编译。
git submodule update --init
git clone https://github.com/jianyangqt/gcta.git
cd gcta
mdir build; cd build
module load arm/gcc/12.2.0
export EIGEN3_INCLUDE_DIR=/path/to/eigen/3.4.0/include/eigen3
export SPECTRA_LIB=/path/to/spectra/1.1.0/include/
export BOOST_LIB=/path/to/boost/1.72.0/include/
export OPENBLAS=/path/to/openblas/0.3.13/
cmake ..
make -j20
静态编译
在 CMAakeList.txt 中 PROJECT 之前 添加 set(CMAKE_TRY_COMPILE_TARGET_TYPE STATIC_LIBRARY),否则会出现 cannot find -lgcc_s 的报错。
添加 gfortran 库依赖,即,将
target_link_libraries(gcta64 mainV1 ${libs_list} Pgenlib z sqlite3 zstd gsl gslcblas m -Wl,--start-group ${BLAS_LIB} -Wl,--end-group gomp -Wl,--whole-archive -lpthread -Wl,--no-whole-archive m dl)
替换为
target_link_libraries(gcta64 mainV1 ${libs_list} Pgenlib z sqlite3 zstd gsl gslcblas m -Wl,--start-group ${BLAS_LIB} -Wl,--end-group gomp -Wl,--whole-archive -lpthread -Wl,--no-whole-archive m dl gfortran)
$ cd build
$ cmake -DCMAKE_CXX_FLAGS="-static"
$ make -j VERBOSE=1
$ ldd gcta64
not a dynamic executable
gcc¶
$ wget https://ftp.gnu.org/gnu/gcc/gcc-10.3.0/gcc-10.3.0.tar.xz
$ tar xf gcc-10.3.0.tar.xz
$ cd gcc-10.3.0
$ contrib/download_prerequisites
$ mkdir build
$ cd build
../configure \
--prefix=/path/to/gcc \
--enable-languages=all \
--enable-threads=posix \
--enable-shared \
--enable-static \
--disable-multilib \
--enable-libatomic \
--enable-libgomp \
--enable-libstdcxx \
$ make -j20 && make install
-
--enable-languages=all启用 GCC 支持的所有语言 -
-enable-threads=posix指定线程模型 -
--enable-shared启用共享库的生成 -
--enable-static启用静态库的生成,否则部分软件编译时报错/usr/bin/ld: cannot find -lstdc++ -
--diskable-multilib禁止多库支持,只编译 64 位库 -
--enable-libatomic启用 libatomic 库,否则部分软件编译时报错/usr/bin/ld: cannot find -latomic。 -
--enable-libgomp启用 libgomp 库(OpenMP 支持) -
--enable-libstdcxx启用 C++ 标准库
GEMMA¶
项目地址 https://github.com/genetics-statistics/GEMMA/
安装依赖库 gsl,openblas,如果没有 libstdc++.a,也需要安装 gcc。最后使用静态库编译 GEMMA,使用时不用另外载入依赖库。
# 使用静态链接库编译,设置环境变量
$ export LIBRARY_PATH=/path/to/gsl/lib/:/path/to/openblas/lib/:/path/to/gcc/lib64/
$ wget https://github.com/genetics-statistics/GEMMA/archive/refs/tags/v0.98.5.tar.gz
$ mv v0.98.5.tar.gz gemma-0.98.5.tar.gz
$ tar xf gemma-0.98.5.tar.gz
$ cd GEMMA-0.98.5/
$ make -j12 static
GenABEL¶
项目地址 https://github.com/GenABEL-Project/GenABEL
$ module load arm/r/4.4.1
$ wget https://cran.r-project.org/src/contrib/Archive/GenABEL.data/GenABEL.data_1.0.0.tar.gz
$ wget https://cran.r-project.org/src/contrib/Archive/GenABEL/GenABEL_1.8-0.tar.gz
$ tar xf GenABEL_1.8-0.tar.gz
$ R CMD INSTALL GenABEL.data_1.0.0.tar.gz
typedef int Sint;,将 GenABEL/src/ctest.h 中的 Sint 替换为 int,sed -i 's/Sint/int/g' GenABEL/src/ctest.h。
修改 GenABEL/src/fexact.c 文件,添加头文件 #include <Rinternals.h> ;
将 PROBLEM "Bug in FEXACT: gave negative key" RECOVER(NULL_ENTRY); 替换为 Rf_error("Bug in FEXACT: gave negative key");;
将 ROBLEM "FEXACT error %d.\n%s", icode, mes RECOVER(NULL_ENTRY); 替换为 Rf_error("FEXACT error %d.\n%s", icode, mes);
安装 R CMD INSTALL GenABEL。
gffread¶
项目地址 https://github.com/gpertea/gffread
$ git clone https://github.com/gpertea/gffread
$ cd gffread/
$ make release
GFlow¶
项目地址:https://github.com/gflow/GFlow
$ git clone https://github.com/gflow/GFlow.git
$ cd GFlow
# 加载 petsc
$ module load arm/petsc/3.22.2-openmpi
# 下载测试算例
$ wget https://github.com/gflow/GFlow/releases/download/v0.1.7-alpha/inputs.tar.gz
在 output.h 中添加一行代码,否则编译时会出现 undefined reference to 'MAX' 的报错。
#define MAX(a, b) ((a) > (b) ? (a) : (b))
make,生成可执行文件 gflow.x 。
运行测试算例
$ tar xf inputs.tar.gz
$ cp input/* .
# 修改 execute_example.sh 中 PETSC_DIR 的位置为 petsc 的安装目录
$ sh execute_example.sh
GLnexus¶
项目地址 https://github.com/dnanexus-rnd/GLnexus
bioconda 中有一个 aarch64 的预编译版本,下载使用报错,看报错应该是在 x86 上编译的。https://anaconda.org/bioconda/glnexus/1.4.1/download/linux-64/glnexus-1.4.1-h17e8430_5.tar.bz2
以下为源码编译。
下载源码
git clone https://github.com/dnanexus-rnd/GLnexus
cd GLnexus
删除 CMakeLists.txt 中所有的 -march=ivybridge -msse4.2 -DHAVE_SSE42 -mpclmul
将 src/genotyper.cc 中的 __asm__(".symver logf,logf@GLIBC_2.2.5");注释。
运行 cmake .
下载所有依赖软件至当前路径下,然后 cp *gz *bz2 *zip external/src/。
https://github.com/giacomodrago/fcmm/archive/v1.0.1.zip 404 了,换成 https://github.com/sigiesec/fcmm/archive/v1.0.1.zip。
https://github.com/samtools/htslib/releases/download/1.9/htslib-1.9.tar.bz2
https://github.com/facebook/rocksdb/archive/v6.29.3.tar.gz
https://github.com/jbeder/yaml-cpp/archive/yaml-cpp-0.6.3.zip
https://github.com/vit-vit/CTPL/archive/v.0.0.2.zip
https://github.com/giacomodrago/fcmm/archive/v1.0.1.zip
https://github.com/gabime/spdlog/archive/v1.8.2.tar.gz
https://capnproto.org/capnproto-c++-0.7.0.tar.gz
https://github.com/philsquared/Catch/archive/v1.12.2.zip
$ ls -1 external/src/{*gz,*zip,*bz2}
external/src/capnproto-c++-0.7.0.tar.gz
external/src/htslib-1.9.tar.bz2
external/src/v.0.0.2.zip
external/src/v1.0.1.zip
external/src/v1.8.2.tar.gz
external/src/v6.29.3.tar.gz
external/src/yaml-cpp-0.6.3.zip
然后将所有cmake中下载相关的动作注释掉
find external/src/ -name "*-impl.cmake" |xargs -i sed -i '1,10s/^/# /' {}
将 src/BCFKeyValueData.cc 中第 467 至 471 行注释,如下所示。
//#ifndef __x86_64__
//if (uint64_t(data.data) % sizeof(::capnp::word)) {
// return Status::Failure("BCFBucketReader: input buffer isn't word-aligned");
//}
//#endif
make 编译,中间会有一次 capnp 相关的报错,继续 make 编译。然后报错
/usr/bin/ld: cannot find -lsnappy
/usr/bin/ld: cannot find -lzstd
/usr/bin/ld: cannot find -lstdc++
加载相关依赖 module load arm/zstd/1.5.6 arm/gcc/12.2.0 arm/snappy/1.2.1,继续 make。这几个依赖不可一开始就是加载,否则依赖 yaml-cpp 编译不通过。
出现如下信息表明 glnexus 编译成功,后面测试部分报错不用管。
[ 76%] Linking CXX executable glnexus_cli
[ 77%] Built target glnexus_cli
[ 78%] Creating directories for 'catch'
[ 79%] Performing download step (download, verify and extract) for 'catch'
测试
$ ./glnexus_cli -h
[2871613] [2025-03-09 19:12:19.710] [GLnexus] [info] glnexus_cli release v1.4.5-0-gfae491b-dirty Mar 9 2025
[2871613] [2025-03-09 19:12:19.710] [GLnexus] [warning] jemalloc absent, which will impede performance with high thread counts. See https://github.com/dnanexus-rnd/GLnexus/wiki/Performance
Usage: ./glnexus_cli [options] /vcf/file/1 .. /vcf/file/N
Merge and joint-call input gVCF files, emitting multi-sample BCF on standard output.
Options:
--dir DIR, -d DIR scratch directory path (mustn't already exist; default: ./GLnexus.DB)
--config X, -c X configuration preset name or .yml filename (default: gatk)
--bed FILE, -b FILE three-column BED file with ranges to analyze (if neither --range nor --bed: use full length of all contigs)
--list, -l expect given files to contain lists of gVCF filenames, one per line
--more-PL, -P include PL from reference bands and other cases omitted by default
--squeeze, -S reduce pVCF size by suppressing detail in cells derived from reference bands
--trim-uncalled-alleles, -a remove alleles with no output GT calls in postprocessing
--mem-gbytes X, -m X memory budget, in gbytes (default: most of system memory)
--threads X, -t X thread budget (default: all hardware threads)
--help, -h print this help message
Configuration presets:
Name CRC32C Description
gatk 1926883223 Joint-call GATK-style gVCFs
gatk_unfiltered 4039280095 Merge GATK-style gVCFs with no QC filters or genotype revision
xAtlas 1991666133 Joint-call xAtlas gVCFs
xAtlas_unfiltered 221875257 Merge xAtlas gVCFs with no QC filters or genotype revision
weCall 2898360729 Joint-call weCall gVCFs
weCall_unfiltered 4254257210 Merge weCall gVCFs with no filtering or genotype revision
DeepVariant 2932316105 Joint call DeepVariant whole genome sequencing gVCFs
DeepVariantWGS 2932316105 Joint call DeepVariant whole genome sequencing gVCFs
DeepVariantWES 1063427682 Joint call DeepVariant whole exome sequencing gVCFs
DeepVariantWES_MED_DP 2412618877 Joint call DeepVariant whole exome sequencing gVCFs, populating 0/0 DP from MED_DP instead of MIN_DP
DeepVariant_unfiltered 3285998180 Merge DeepVariant gVCFs with no QC filters or genotype revision
Strelka2 395868656 [EXPERIMENTAL] Merge Strelka2 gVCFs with no QC filters or genotype revision
GxS 3929547104 [EXPERIMENTAL] merging for GxS
GMAP¶
项目地址 http://research-pub.gene.com/gmap/src/
$ wget http://research-pub.gene.com/gmap/src/gmap-gsnap-2023-12-01.tar.gz
$ tar xf gmap-gsnap-2023-12-01.tar.gz
$ cd gmap-2023-12-01/
$ ./configure --prefix=/path/to/gmap/ --with-simd-level=arm
gnuplot¶
$ wget http://nchc.dl.sourceforge.net/project/gnuplot/gnuplot/5.4.9/gnuplot-5.4.9.tar.gz
$ tar xf gnuplot-5.4.9.tar.gz
$ cd gnuplot-5.4.9
$ module load arm/lua/5.4.7
$ ./configure --prefix=/path/gnuplot/ --with-qt=no
$ make -j20 && make install
gromacs¶
参考
$ module load arm/openmpi/5.0.5 arm/openblas/0.3.28
$ wget http://ftp.gromacs.org/pub/gromacs/gromacs-2019.5.tar.gz
$ tar xf gromacs-2019.5.tar.gz
$ cd gromacs-2019.5/
$ mkdir build && cd build/
$ cmake .. -DCMAKE_INSTALL_PREFIX=/path/to/gromacs/ -DGMX_SIMD=ARM_NEON_ASIMD -DGMX_OPENMP=on -DGMX_MPI=on -DGMX_X11=off -DGMX_GPU=off -DFFTWF_LIBRARY=/path/to/lib//libfftw3f.so -DFFTWF_INCLUDE_DIR=/path/to/include
$ make -j20
$ make install
$ cmake .. -DGMX_GPU=CUDA -DGMX_BUILD_OWN_FFTW=ON -DCMAKE_INSTALL_PREFIX=/path/to/gromacs/
$ make -j20
$ make install
http://www.fftw.org/fftw-3.3.8.tar.gz。
$ cmake .. -DGMX_GPU=CUDA -DGMX_BUILD_OWN_FFTW=ON -DCMAKE_C_COMPILER=`which gcc` -DCMAKE_INSTALL_PREFIX=/path/to/GROMACS/ -DGMX_BUILD_OWN_FFTW_URL=/path/to/fftw-3.3.8.tar.gz -DGMX_BUILD_OWN_FFTW_MD5=8aac833c943d8e90d51b697b27d4384d
$ make -j20 && make install
https://github.com/amd/amd-fftw/issues/3
$ module load arm/plumed/2.9.2-bisheng arm/hyper-mpi/24.6.30_bisheng
$ wget http://ftp.gromacs.org/pub/gromacs/gromacs-2021.7.tar.gz
$ tar xf gromacs-2021.7.tar.gz
$ cd gromacs-2021.7/
# 为 plumed 打 patch
$ plumed-patch -p -e gromacs-2021.7
$ mkdir build && cd build/
$ cmake .. -DCMAKE_INSTALL_PREFIX=/path/to/2021.7-plumed -DGMX_SIMD=ARM_NEON_ASIMD -DGMX_THREAD_MPI=OFF -DGMX_OPENMP=on -DGMX_MPI=on -DGMX_X11=off -DGMX_GPU=off -DBUILD_SHARED_LIBS=OFF -DGMX_PREFER_STATIC_LIBS=ON -DGMXAPI=OFF -DCMAKE_C_COMPILER=`which mpicc` -DCMAKE_CXX_COMPILER=`which mpic++` -DGMX_FFT_LIBRARY=fftw3 -DCMAKE_PREFIX_PATH=/path/to/fftw3/
$ make -j96 && make install
hal2vg¶
项目地址 https://github.com/glennhickey/hal2vg
git clone https://github.com/glennhickey/hal2vg.git --recursive
cd hal2vg
sed -i '1,$s/std=c++11/std=c++17/` deps/libbdsg-easy/deps/DYNAMIC/CMakeLists.txt
# 需要运行 3 次 make
make -j
make -j
make -j
./hal2vg
HiCPlotter¶
项目地址 https://github.com/akdemirlab/HiCPlotter
$ module load arm/python/2.7.18
$ pip install --prefix=/path/to/hicplotter/ bx-python==1.16.6
# 设置这2个环境变量,否则 scipy 安装编译报错,Error: Rank mismatch between actual argument at (1) and actual argument at (2) (scalar and rank-1)
$ export FCFLAGS="-fallow-argument-mismatch"
$ export FFLAGS="-fallow-argument-mismatch"
$ pip install --prefix=/path/to/hicplotter/ scipy==1.2.3
$ pip install --prefix=/path/to/hicplotter/ matplotlib==1.4.3
# 在 mpl_toolkits 目录下添加 __init__.py,否则会出现报错 ImportError: No module named mpl_toolkits.axes_grid1
$ touch /path/to/hicplotter/lib/python2.7/site-packages/mpl_toolkits/__init__.py
$ tar xf HiCPlotter-0.6.6.tar.gz
$ cd HiCPlotter-0.6.6/
# 编辑 HiCPlotter.py,在首行添加 #!/bin/env python
$ chmod +x HiCPlotter.py
$ cp HiCPlotter.py /path/to/hicplotter/bin/
$ /path/to/hicplotter/bin/HiCPlotter.py -h
HiC-Pro¶
HiC-Pro-2.11.4
module load arm/python/2.7.18 arm/lzo/2.10
pip install --prefix=/path/to/hic-pro/ bx-python==0.8.1 numpy==1.8.2 pysam==0.8.3
# 设置这2个环境变量,否则 scipy 安装编译报错,Error: Rank mismatch between actual argument at (1) and actual argument at (2) (scalar and rank-1)
export FCFLAGS="-fallow-argument-mismatch"
export FFLAGS="-fallow-argument-mismatch"
pip install --prefix=/path/to/hic-pro/ scipy==1.2.3
tar xf HiC-Pro-2.11.4.tar.gz
cd HiC-Pro-2.11.4/
# 载入相关依赖软件,需要安装这3个R包,ggplot2 (>2.2.1) RColorBrewer grid
module load arm/r/4.4.1 arm/bowtie2/2.5.4 arm/samtools/1.21
# 编辑 config-install.txt,PREFIX = /path/to/hic-pro/
make configure
make CONFIG_SYS=config-install.txt
pip install --prefix=/path/to/hic-pro/ "numpy<2"
HiC-Pro-3.1.0
wget https://files.pythonhosted.org/packages/e2/3f/c2747248931fc997d2fe303c4e18827cc0c94114fba3e2c709e530db7226/iced-0.5.13.tar.gz
pip install --prefix=/path/to/hic-pro/ numpy==1.19.5 scipy==1.9.3 pysam==0.17.0 bx-python==0.9.0 cython
tar xf iced-0.5.13.tar.gz
cd iced-0.5.13
cython iced/normalization/_normalization_.pyx
python setup.py install --prefix=/path/to/hic-pro/
tar xf hic-pro-3.1.0.tar.gz
HiC-Pro-3.1.0
# 载入相关依赖软件,需要安装这3个R包,ggplot2 (>2.2.1) RColorBrewer grid
module load arm/r/4.4.1 arm/bowtie2/2.5.4 arm/samtools/1.21
# 编辑 config-install.txt,PREFIX = /path/to/hic-pro/
make configure
make CONFIG_SYS=config-install.txt
hisat2¶
项目地址 https://github.com/DaehwanKimLab/hisat2
下载源码及依赖库
$ git clone https://github.com/DaehwanKimLab/hisat2.git
cd hisat2
$ wget https://raw.githubusercontent.com/lh3/ksw2/refs/heads/master/ksw2.h
$ wget https://raw.githubusercontent.com/DLTcollab/sse2neon/refs/heads/master/sse2neon.h
alphabet.cpp 文件的 404 行,将 -1 , 更改为 char(-1) ,
更改后为
char mask2iupac[16] = {
char(-1),
'A', // 0001
'C', // 0010
'M', // 0011
'G', // 0100
'R', // 0101
'S', // 0110
'V', // 0111
'T', // 1000
'W', // 1001
'Y', // 1010
'H', // 1011
'K', // 1100
'D', // 1101
'B', // 1110
'N', // 1111
};
sse_util.h aligner_sw.h 文件中的 #include <emmintrin.h> 替换为 #include "sse2neon.h"
Makefile 文件中的 EXTRA_FLAGS += -DPOPCNT_CAPABILITY 注释掉
Makefile 166-175 行的如下内容
BITS_FLAG =
ifeq (32,$(BITS))
BITS_FLAG = -m32
endif
ifeq (64,$(BITS))
BITS_FLAG = -m64
endif
SSE_FLAG=-msse2
BITS_FLAG =
BITS=64
ifeq (32,$(BITS))
BITS_FLAG = -m32
endif
ifeq (64,$(BITS))
ifeq (0, $(AARCH64))
BITS_FLAG = -m64
endif
endif
SSE_FLAG =
ifeq (0, $(AARCH64))
SSE_FLAG=-msse2
BITS_FLAG = -m64
endif
#SSE_FLAG=-msse2
make -j12
hifiasm¶
项目地址 https://github.com/chhylp123/hifiasm
-
下载二进制文件
$ wget https://anaconda.org/bioconda/hifiasm/0.19.9/download/linux-aarch64/hifiasm-0.19.9-hf1d9491_0.tar.bz2 -
手动编译
下载源码及依赖库
$ git https://github.com/chhylp123/hifiasm.git
$ cd hifiasm
$ wget https://raw.githubusercontent.com/DLTcollab/sse2neon/refs/heads/master/sse2neon.h
Levenshtein_distance.h 中的 以下头文件都注释,添加 #include "sse2neon.h"
#include "emmintrin.h"
#include "nmmintrin.h"
#include "smmintrin.h"
#include <immintrin.h>
//#include "emmintrin.h"
//#include "nmmintrin.h"
//#include "smmintrin.h"
//#include <immintrin.h>
#include "sse2neon.h"
Makefile 中的
#CXXFLAGS= -g -O3 -msse4.2 -mpopcnt -fomit-frame-pointer -Wall
替换为
CXXFLAGS= -g -O3 -fomit-frame-pointer -Wall
最后 make -j12
IBSpy¶
项目地址 https://github.com/Uauy-Lab/IBSpy
module load arm/python/3.11.10
pip install --prefix=/path/to/ibspy/ numpy pandas cython biopython pyfaidx pandas psutil matplotlib pyranges multiprocess pysam scikit-learn
wget https://github.com/Uauy-Lab/IBSpy/archive/refs/tags/0.4.6.tar.gz
tar 0.4.6.tar.gz
cd IBSpy-0.4.6
pip install --prefix=/path/to/ibspy/
# /path/to/ibspy/lib/python3.11/site-packages/IBSpy/window_affinity_propagation.py 中 `from numpy import NaN, ndarray` 改为 `from numpy import nan, ndarray`
# KMC api 安装
cd ..
wget https://github.com/refresh-bio/KMC/archive/refs/tags/v3.1.1.tar.gz
tar xf v3.1.1.tar.gz
cd KMC-3.1.1
# 将 makefile 中的 -I $(PY_KMC_API_DIR)/libs/pybind11/include 替换为 -I /share/software/app/arm/python/3.11.10/lib/python3.11/site-packages/pybind11/include/
make py_kmc_api
cp bin/*so /path/to/ibspy/lib/python3.11/site-packages/
# 测试
IBSpy -h
sh tests/integration_python.sh
k8¶
项目地址 https://github.com/attractivechaos/k8
直接使用 bioconda 中编译好的版本
$ wget https://anaconda.org/bioconda/k8/1.2/download/linux-aarch64/k8-1.2-h7f4e536_0.tar.bz2
$ tar xf k8-1.2-h7f4e536_0.tar.bz2
$ module load arm/gcc/12.2.0
$ ./bin/k8
Usage: k8 [options] <script.js> [arguments]
Options:
-e STR execute STR
-E STR execute STR and print results
-m INT v8 max size of the old space (in Mbytes) [16384]
-v print version number
--help show v8 command-line options
LACHESIS¶
项目地址 https://github.com/shendurelab/LACHESIS
# 注意 samtools 的版本不高于 0.1.19,samtools的include目录结构为 include/bam/xxxx.h
# boost 版本不高于 1.70.0
$ module load arm/samtools/0.1.19 arm/boost/1.60.0
$ tar xf shendurelab-LACHESIS-2e27abb.tar.gz
$ cd shendurelab-LACHESIS-2e27abb
$ ./configure --with-samtools=/path/to/samtools/0.1.19/ --prefix=/path/to/lachesis/ --with-boost=/path/to/boost/1.60.0
$ make -j 20
$ make install
lammps¶
$ module load arm/openmpi/5.0.5
$ wget https://download.lammps.org/tars/lammps.tar.gz
$ #tar xf lammps.tar.gz
$ cd lammps-29Aug2024
$ cd src
$ mkdir build
# 添加所需的包
$ make yes-manybody yes-meam yes-extra-fix yes-mc
$ make -j80 g++_openmpi
lapack¶
编译静态库
将 librefblas.a、libcblas.a、liblapack.a、liblapacke.a 都编译出来。
$ wget http://www.netlib.org/lapack/lapack.tgz
$ tar lapack
$ cd lapack-3.10.1
$ cp make.inc.example make.inc
$ make blaslib
$ make cblaslib
$ make lapacklib
$ make lapackelib
1、 静态库路径
检查下 lapack-3.10.1 下是否有 librefblas.a、libcblas.a、liblapack.a、liblapacke.a 这几个文件,没有的话重新编译一次。
- 头文件路径
cblas头文件路径: lapack-3.10.1/CBLAS/include
lapacke头文件路径: lapack-3.10.0/LAPACKE/include
cblas 是 blas 的 c 接口,lapacke 是 lapack 的 c 接口,由于 blas、lapack 是 fortran 代码所以这两个没有头文件,只有 .a 文件。
参考
https://www.cnblogs.com/babyclass/p/16358589.html
编译动态库
动态库编译需要使用 cmake 编译
$ wget http://www.netlib.org/lapack/lapack.tgz
$ tar lapack
$ cd lapack-3.10.1
$ mkdir build && cd build
$ cmake .. -DCMAKE_BUILD_TYPE=RELEASE -DBUILD_SHARED_LIBS=ON -DCBLAS=ON -DLAPACKE=ON
$ make -j
$ ls -1 lib/
libblas.so -> libblas.so.3
libblas.so.3 -> libblas.so.3.10.0
libblas.so.3.10.0
libcblas.so -> libcblas.so.3
libcblas.so.3 -> libcblas.so.3.10.0
libcblas.so.3.10.0
liblapacke.so -> liblapacke.so.3
liblapacke.so.3 -> liblapacke.so.3.10.0
liblapacke.so.3.10.0
liblapack.so -> liblapack.so.3
liblapack.so.3 -> liblapack.so.3.10.0
liblapack.so.3.10.0
option(BUILD_SHARED_LIBS "Build shared libraries" OFF)
option(BUILD_INDEX64 "Build Index-64 API libraries" OFF)
option(BUILD_TESTING "Build tests" ${_is_coverage_build})
option(BUILD_DEPRECATED "Build deprecated routines" OFF)
option(BUILD_SINGLE "Build single precision real" ON)
option(BUILD_DOUBLE "Build double precision real" ON)
option(BUILD_COMPLEX "Build single precision complex" ON)
option(BUILD_COMPLEX16 "Build double precision complex" ON)
option(USE_OPTIMIZED_BLAS "Whether or not to use an optimized BLAS library instead of included netlib BLAS" OFF)
option(CBLAS "Build CBLAS" OFF)
option(USE_XBLAS "Build extended precision (needs XBLAS)" OFF)
option(USE_OPTIMIZED_LAPACK "Whether or not to use an optimized LAPACK library instead of included netlib LAPACK" OFF)
option(LAPACKE "Build LAPACKE" OFF)
option(LAPACKE_WITH_TMG "Build LAPACKE with tmglib routines" OFF)
option(BLAS++ "Build BLAS++" OFF)
option(LAPACK++ "Build LAPACK++" OFF)
option(BUILD_HTML_DOCUMENTATION "Create and install the HTML based API
option(BUILD_MAN_DOCUMENTATION "Create and install the MAN based documentation (requires Doxygen) - command: make man" OFF)
libdb¶
https://github.com/berkeleydb/libdb
$ git clone https://github.com/berkeleydb/libdb.git
$ cd libdb/build_unix
$ ../dist/configure --prefix=/path/to/libdb/ --build=arm-linux-gnueabi
$ make -j20
$ make install
Liftoff¶
项目地址 https://github.com/agshumate/Liftoff
git clone https://github.com/agshumate/Liftoff
cd Liftoff
pip install --prefix=/path/to/liftoff/ numpy pysam
PYTHONPATH=//path/to/liftoff/lib/python3.9/site-packages/://path/to/liftoff/lib64/python3.9/site-packages/ python setup.py install --prefix=/path/to/liftoff/
llvm¶
项目地址 https://github.com/llvm/llvm-project
git clone https://github.com/llvm/llvm-project.git
cd llvm-project
cmake -S llvm -B build -DCMAKE_INSTALL_PREFIX=/path/to/llvm/ -DCMAKE_BUILD_TYPE=Release -DLLVM_ENABLE_PROJECTS="clang;lld"
cmake --build build -j 80
mafft¶
参考 https://mafft.cbrc.jp/alignment/software/installation_without_root.html
mash¶
项目地址 github.com/marbl/Mash/
源码编译
$ module load capnproto/1.1.0 gsl/2.8 gcc/10.3.0
$ tar xf Mash-2.3.tar.gz
$ cd Mash-2.3
$ sed -i '8,10s/^/#/' Makefile.in
$ autoreconf -i
$ ./configure --prefix=/path/to/mash/ --with-capnp=/path/to/capnproto/ --with-gsl=/path/to/gsl/ --enable-static-gsl
$ make -j
$ make install
$ /path/to/mash/bin/mash -h
Mash version 2.3
Type 'mash --license' for license and copyright information.
Usage:
mash <command> [options] [arguments ...]
Commands:
bounds Print a table of Mash error bounds.
dist Estimate the distance of query sequences to references.
info Display information about sketch files.
paste Create a single sketch file from multiple sketch files.
screen Determine whether query sequences are within a larger mixture of
sequences.
sketch Create sketches (reduced representations for fast operations).
taxscreen Create Kraken-style taxonomic report based on mash screen.
triangle Estimate a lower-triangular distance matrix.
使用 bioconda 中编译好的版本。
先编译 cblas 动态库
tar xf lapack-3.10.0.tgz
cd lapack-3.10.0
mkdir build && cd build
cmake .. -DCMAKE_INSTALL_PREFIX=/path/to/mash/ -DCMAKE_BUILD_TYPE=RELEASE -DCBLAS=ON
make -j
make install
$ wget https://anaconda.org/bioconda/mash/2.3/download/linux-aarch64/mash-2.3-heca2785_9.tar.bz2
$ tar xf mash-2.3-heca2785_9.tar.bz2
$ module load arm/gsl/2.7 arm/gcc/14.2.0
$ cp -r bin/ /path/to/mash/
$ export LD_LIBRARY_PATH=/path/to/mash/lib64/:$LD_LIBRARY_PATH
$ /path/to/mash/bin/mash -h
Mash version 2.3
Type 'mash --license' for license and copyright information.
Usage:
mash <command> [options] [arguments ...]
Commands:
bounds Print a table of Mash error bounds.
dist Estimate the distance of query sequences to references.
info Display information about sketch files.
paste Create a single sketch file from multiple sketch files.
screen Determine whether query sequences are within a larger mixture of
sequences.
sketch Create sketches (reduced representations for fast operations).
taxscreen Create Kraken-style taxonomic report based on mash screen.
triangle Estimate a lower-triangular distance matrix.
megacc¶
项目地址 https://github.com/KumarMEGALab/MEGA-source-code https://www.megasoftware.net/
mega 使用 pascal 语言编写,Lazarus 编译。这里只编译命令行工具 megacc 。
cd ~/megacc
# 准备编译环境
# wget https://downloads.freepascal.org/fpc/beta/3.2.4-rc1/aarch64-linux/fpc-3.2.4-rc1.aarch64-linux.tar
wget https://sourceforge.net/projects/lazarus-ccr/files/FPSpreadsheet/fpspreadsheet-1.16.zip
wget https://download.lazarus-ide.org/Lazarus%20Linux%20arm64%20DEB/Lazarus%204.2/lazarus-project_4.2.0-0_arm64.deb
wget https://download.lazarus-ide.org/Lazarus%20Linux%20arm64%20DEB/Lazarus%204.2/fpc-laz_3.2.3-240813_arm64.deb
$ mkdir lazarus-project
$ mv lazarus-project_4.2.0-0_arm64.deb lazarus-project
$ ar -vx lazarus-project_4.2.0-0_arm64.deb
$ tar xf data.tar.xz
$ mkdir fpc-laz
$ mv fpc-laz_3.2.3-240813_arm64.deb fpc-laz
$ cd fpc-laz
$ ar -vx fpc-laz_3.2.3-240813_arm64.deb
$ tar xf data.tar.xz
$ cd usr/bin
$ mkdir aarch64-linux
$ mv * aarch64-linux
$ cd aarch64-linux
$ ln -s ../../lib/fpc/3.2.3/ppca64 ppca64
$ cd ~/megacc/lazarus-project
$ mv ../fpc-laz/usr usr/share/lazarus/4.2.0/fpc
export PATH=$HOME/megacc/lazarus-project/usr/bin/:$HOME/megacc/lazarus-project/usr/share/lazarus/4.2.0/fpc/bin/aarch64-linux:$PATH
$ which lazbuild fpc fpcres
~/megacc/lazarus-project/usr/bin/lazbuild
~/megacc/lazarus-project/usr/share/lazarus/4.2.0/fpc/bin/aarch64-linux/fpc
~/megacc/lazarus-project/usr/share/lazarus/4.2.0/fpc/bin/aarch64-linux/fpcres
$ mkdir ~/megacc/fpspreadsheet
$ cd ~/megacc/fpspreadsheet
$ cp ../fpspreadsheet-1.16.zip .
$ unzip fpspreadsheet-1.16.zip
# 编译生成 laz_fpspreadsheet.pas
$ lazbuild --lazarusdir="$HOME/megacc/lazarus-project/usr/share/lazarus/4.2.0" --pcp="$HOME/.lazarus" laz_fpspreadsheet.lpk
# 下载mega源码并编译megacc
$ cd ~/megacc
$ git clone https://github.com/KumarMEGALab/MEGA-source-code/
$ cd MEGA-source-code/MEGA12-source/MEGA-CC
$ cp resources_windows.lrs resources_unix.lrs
将 megaccxplatform.lpi 中的以下三行删除
<ExecuteAfter>
<Command Value="test_cc_dependencies.sh"/>
</ExecuteAfter>
$ lazbuild --lazarusdir="$HOME/megacc/lazarus-project/usr/share/lazarus/4.2.0" megaccxplatform.lpi --build-mode=release-Linux --cpu=aarch64 > megacc-build.log 2>&1
# 确认编译成功
$ ../megacc -v
Molecular Evolutionary Genetics Analysis
Molecular Evolutionary Genetics Analysis - Computational Core
Version: 12.0.4
Build: 12241219-arm64
Authors: Koichiro Tamura, Glen Stecher, Sudhir Kumar
Copyright 2011-2025
Web: http://www.megasoftware.net
megahit¶
项目地址 https://github.com/voutcn/megahit
-
下载二进制文件
https://anaconda.org/bioconda/megahit/1.2.9/download/linux-aarch64/megahit-1.2.9-hf1d9491_5.tar.bz2 -
手动编译
$ git clone https://github.com/voutcn/megahit.git $ cd megahit $ wget https://raw.githubusercontent.com/DLTcollab/sse2neon/refs/heads/master/sse2neon.h $ cp sse2neon.h src
将 src/parallel_hashmap/phmap_config.h 中 618 行的 #include <emmintrin.h> 和 623 行的 #include <tmmintrin.h> 分别替换为 #include "../sse2neon.h",如下所示。
#if PHMAP_HAVE_SSE2
//#include <emmintrin.h>
#include "../sse2neon.h"
#endif
#if PHMAP_HAVE_SSSE3
//#include <tmmintrin.h>
#include "../sse2neon.h"
#endif
src/xxhash/xxh3.h 中 618 行的 #include <emmintrin.h> 和 623 行的 #include <tmmintrin.h> 分别替换为 #include "../sse2neon.h",如下所示。
#if defined(__GNUC__)
# if defined(__AVX2__)
//# include <immintrin.h>
#include "../sse2neon.h"
# elif defined(__SSE2__)
//# include <emmintrin.h>
#include "../sse2neon.h"
src/main.cpp 中 包含 HasPopcnt() 和 HasBmi2() 的行注释掉,如下所示。
} else if (strcmp(argv[1], "checkcpu") == 0) {
// pprintf("{}\n", HasPopcnt() && HasBmi2());
} else if (strcmp(argv[1], "checkpopcnt") == 0) {
// pprintf("{}\n", HasPopcnt());
} else if (strcmp(argv[1], "checkbmi2") == 0) {
// pprintf("{}\n", HasBmi2());
} else if (strcmp(argv[1], "dumpversion") == 0) {
将 src/kmlib/kmrns.h 中 11 行的 #include <x86intrin.h> 注释掉,如下所示。
// #include <x86intrin.h>
CMakeLists.txt 中 76-77 两行注释掉,即禁用 -mbmi2 -DUSE_BMI2 和 -mpopcnt 相关的编译选项,如下所示。
set_target_properties(megahit_core PROPERTIES COMPILE_FLAGS "-mbmi2 -DUSE_BMI2 -mpopcnt")
set_target_properties(megahit_core_popcnt PROPERTIES COMPILE_FLAGS "-mpopcnt")
编译
$ mkdir build
$ cd build
$ cmake ../ .. -DCMAKE_BUILD_TYPE=Release
$ make -j12
$ make install
metabat¶
下载二进制文件
$ module load arm/libdeflate/1.8 arm/boost/1.85.0 arm/gcc/12.2.0
$ wget https://anaconda.org/bioconda/metabat2/2.17/download/linux-aarch64/metabat2-2.17-hb490ba1_0.tar.bz2
$ tar xf metabat2-2.17-hb490ba1_0.tar.bz2
$ ./bin/metabat1
$ git clone https://bitbucket.org/berkeleylab/metabat.git
$ cd metabat
$ mkdir build && cd build
$ cmake -DCMAKE_INSTALL_PREFIX=/path/to/metabat ..
# make 时会从 github 下载依赖,需保持 github 网络通畅
$ make -j20
$ make install
MCScanX¶
项目地址 https://github.com/wyp1125/MCScanX
$ tar xf MCScanX-1.0.0.tar.gz
$ cd MCScanX-1.0.0/
$ make
# 测试
$ ./MCScanX -h
# 使用下游分析脚本
$ cd downstream_analyses/
$ java bar_plotter -h
Usage: java bar_plotter -g gff_file -s collinearity_file -c control_file -o output_PNG_file
minigraph¶
项目地址 https://github.com/lh3/minigraph
$ git clone https://github.com/lh3/minigraph
$ cd minigraph
# 去掉 -msse4 编译选项
$ sed -i 's/-msse4//g' Makefile
$ make
minimap2¶
项目地址 https://github.com/lh3/minimap2
$ git clone https://github.com/lh3/minimap2
$ cd minimap2
$ make arm_neon=1 aarch64=1
miniprot¶
项目地址 https://github.com/lh3/miniprot
$ git clone https://github.com/lh3/miniprot
$ cd miniprot && make
msmc2¶
项目地址 https://github.com/stschiff/msmc2
使用 ldmd2 代替 dmd
$ module load arm/ldc2/1.41.0
$ cd /path/to/ldc2/1.41.0/bin/
$ ln -s ldmd2 dmd
$ tar xf msmc2-2.1.4.tar.gz
$ cd msmc2-2.1.4
# 将 Makefile 中的 GSLDIR=/usr/local/lib 替换为 GSLDIR=/path/to/gsl/2.4/lib/
$ make -j
$ build/release/msmc2
muscle¶
项目地址:https://www.drive5.com/muscle/
github上编译好的aarch64版本,运行会报错 myutils.cpp(106) assert failed: sizeof(void *) == 4,查看源码,其原因为代码未能正确识别 64 位CPU,因此需要下载源码重新编译。
muscle-5.3.tar.gz
tar xf muscle-5.3.tar.gz
cd muscle-5.3/src/
myutils.h 第 13 行代码
13 #if defined(__x86_64__) || defined(_M_X64) || defined(__arm64__)
修改为
13 #if defined(__x86_64__) || defined(_M_X64) || defined(__arm64__) || defined(__aarch64__)
然后 sh build_linux.bash 编译,编译后生成可执行文件 ../bin/muscle。
也可以直接在 bioconda 中下载编译的 aarch64 版本
https://anaconda.org/bioconda/muscle/files
$ wget https://anaconda.org/bioconda/muscle/5.3/download/linux-aarch64/muscle-5.3-h78569d1_0.tar.bz2
$ tar xf muscle-5.3-h78569d1_0.tar.bz2
$ ls bin/muscle
nanom6A¶
项目地址:https://github.com/gaoyubang/nanom6A
打开链接 https://drive.google.com/drive/folders/1Dodt6uJC7lBihSNgT3Mexzpl_uqBagu0
下载 nanom6A_2022_12_22.tar.gz 并解压
安装 python 依赖库
# 注意固定 numpy 和 xgboost 的版本,否则可能会安装或运行出错
$ pip install --prefix=/path/to/nanom6a/ joblib numpy==1.21 pysam scikit-learn scipy tqdm xgboost==0.80 h5py statsmodels
安装 jvarkit-sam2tsv
$ wget https://anaconda.org/HCC/jvarkit-sam2tsv/1.0/download/linux-64/jvarkit-sam2tsv-1.0-0.tar.bz2
tar xf jvarkit-sam2tsv-1.0-0.tar.bz2
mkdir /path/to/nanom6a/jar/
cp dist/sam2tsv.jar /path/to/nanom6a/jar/
cp lib/com/beust/jcommander/1.64/jcommander-1.64.jar /path/to/nanom6a/jar/
cp lib/com/github/samtools/htsjdk/2.9.1/htsjdk-2.9.1.jar /path/to/nanom6a/jar/
# 创建sam2tsv脚本
$ cat /path/to/nanom6a/bin/sam2tsv
#!/bin/bash
java -Djvarkit.log.name=sam2tsv -Dfile.encoding=UTF8 -Xmx500m -cp "/path/to/nanom6a/jar/htsjdk-2.9.1.jar:/path/to/nanom6a/jar/jcommander-1.64.jar:/path/to/nanom6a/jar/sam2tsv.jar" com.github.lindenb.jvarkit.tools.sam2tsv.Sam2Tsv $*
测试
cd nanom6A_2022_12_22
$ export PYTHONPATH=/path/to/nanom6a/lib/python3.9/site-packages/:/path/to/nanom6a/lib64/python3.9/site-packages/
$ export PATH="/path/to/nanom6a/bin/:$PATH"
$ module load arm/samtools/1.21 arm/minimap2/2.28 arm/bedtools/2.31.1
$ sh run_source_code.sh
如果 run_source_code.sh 运行正常没有报错,则表示相关依赖安装完成。
ncurses¶
$ wget https://ftp.gnu.org/pub/gnu/ncurses/ncurses-6.5.tar.gz
$ tar xf ncurses-6.5.tar.gz
$ cd ncurses-6.5
$ ./configure --prefix=/path/to/ncurses/ \
--with-shared \
--enable-overwrite \
--without-ada \
--enable-symlinks \
--with-versioned-syms \
--enable-pc-files \
--enable-ext-colors \
--enable-widec \
$ make
$ make install
$ mkdir /path/to/ncurses/lib/pkconfig
$ cp misc/*pc /path/to/ncurses/lib/pkconfig
# 作用说明
--with-shared 生成动态链接库,允许程序运行时动态加载
--enable-overwrite 允许安装时覆盖现有文件,避免冲突
--without-ada 禁用 Ada 语言支持,减少编译时间和依赖
--enable-symlinks 创建符号链接,提供版本兼容性
--with-versioned-syms 使用版本化符号,支持多版本共存
--enable-pc-files 生成 pkg-config 文件,方便其他软件查找依赖
--enable-widec 重要:启用宽字符支持,支持 Unicode 和 UTF-8
--enable-ext-colors 启用扩展颜色支持,提供更多颜色选项
openblas¶
项目地址 https://github.com/OpenMathLib/OpenBLAS
$ wget https://github.com/OpenMathLib/OpenBLAS/releases/download/v0.3.28/OpenBLAS-0.3.28.tar.gz
$ tar xf OpenBLAS-0.3.28.tar.gz
$ cd OpenBLAS
$ make -j20
$ make install PREFIX=/path/to/openblas/
export OPENBLAS_NUM_THREADS=4
export GOTO_NUM_THREADS=4
export OMP_NUM_THREADS=4
odgi¶
项目地址 https://github.com/pangenome/odgi
$ git clone --recursive https://github.com/pangenome/odgi.git
$ cd odgi
# 去掉 -msse4.2 编译选项
$ sed -i 's/-msse4.2//g' ./CMakeLists.txt
$ sed -i 's/-msse4.2//g' ./deps/sdsl-lite/CMakeLists.txt
$ sed -i 's/-msse4.2//g' ./deps/mmmulti/deps/sdsl-lite/CMakeLists.txt
$ sed -i 's/arch=x86-64/arch=armv8-a/g' src/main.cpp
$ module load arm/jemalloc/5.2.1
# 使用自己编译好的 gcc,否则会出现报错 /usr/bin/ld: cannot find -latomic
$ module load arm/gcc/10.3.0
$ cmake -H. -Bbuild
$ cmake --build build -- -j 20
$ bin/odgi -h
ORCA¶
项目地址 https://orcaforum.kofo.mpg.de/app.php/dlext/?cat=25
ORCA 官方只有编译好的二进制版本,其依赖的 glibc 和 gcc 版本较高,这里手动处理这 2 个依赖。
$ cd /path/to/orca/
$ tar xf orca_6_0_1_linux_arm64_shared_openmpi418.tar.xz
$ mv orca_6_0_1_linux_arm64_shared_openmpi418 orca
$ git clone https://github.com/matrix1001/glibc-all-in-one
$ cd glibc-all-in-one
# 下载适合版本的 libc6,此处的 glibc 版本为 2.41
$ wget https://ftp.debian.org/debian/pool/main/g/glibc/libc6_2.41-6_arm64.deb
$ mkdir glibc
# 解压 deb
$ ./extract libc6_2.41-6_arm64.deb glibc
$ mv glibc ../orca
# 使用 patchelf 更改可执行文件的 interpreter
module load arm/patchelf/0.18.0
$ ls -1 /path/to/orca/orca/orca* |xargs -i patchelf --set-interpreter /path/to/orca/orca/glibc/ld-linux-aarch64.so.1 --add-rpath /path/to/orca/orca/glibc/ {}
$ ls -1 /path/to/orca/orca/autoci_* |xargs -i patchelf --set-interpreter /path/to/orca/orca/glibc/ld-linux-aarch64.so.1 --add-rpath /path/to/orca/orca/glibc/ {}
# 更改 orca 动态库的 rpath
$ patchelf --add-rpath /path/to/orca/orca/glibc/ /path/to/orca/orca/lib/liborca_tools_6_0_1.so.6
$ patchelf --add-rpath /path/to/orca/orca/glibc/ /path/to/orca/orca/lib/liborca_tools_6_0_1_mpi.so.6
# 加载 gcc 和 openmpi 依赖
$ module load arm/gcc/14.2.0 arm/openmpi/5.0.5
# 测试
$ orca
This program requires the name of a parameterfile as argument
For example ORCA TEST.INP
# 可用
$ #singularity build --sandbox orca_openmpi2 docker://dockerpull.org/fenicsproject/test-env:current-openmpi
# 不可用
$ #singularity build --sandbox orca_openmpi3 docker://dockerpull.org/mfisherman/openmpi:4.1.6
OrthoFinder¶
项目地址 https://github.com/davidemms/OrthoFinder
$ wget https://github.com/davidemms/OrthoFinder/archive/refs/tags/3.0.1b1.tar.gz
$ tar xf 3.0.1b1.tar.gz
$ cd OrthoFinder-3.0.1b1
$ pip install numpy scipy --prefix=/path/to/orthofinder/
$ pip install . --prefix=/path/dir/orthofinder/
# 载入依赖
$ module load arm/diamond/2.1.10 arm/mcl/22-282 arm/fastme/2.1.6.4 arm/mafft/7.525 arm/fasttree/2.1.11
# 测试
$ orthofinder -f ExampleData/
nextdenovo¶
$ module load arm/curl/8.10.1 arm/openssl/3.3.2
$ git clone git@github.com:Nextomics/NextDenovo.git
$ cd NextDenovo && make arm_neon=1 aarch64=1
PanDepth¶
项目地址https://github.com/HuiyangYu/PanDepth
软件编译时使用了在x86平台上预编译好的静态库,使用自己编译的静态库时报错较多,故这里使用依赖的动态库编译。
$ module load arm/libdeflate/1.8 arm/htslib/1.21
$ git clone https://github.com/HuiyangYu/PanDepth.git
$ cd PanDepth
$ mkdir include2
$ cp include/{comm.h,DataClass.h,gzstream.*} include2/
$ g++ --std=c++11 -g -O3 src/PanDepth.cpp -lhts -ldeflate -lz -pthread -Iinclude2 -o bin/pandepth
$ cd bin
$ ./pandepth -h
htslib 使用默认编译参数出来的静态库 libhts.a ,PanDepth 在使用时出现很多类似 undefined reference to 'BZ2_bzBuffToBuffCompress' 的报错。这里 htslib 编译时需要添加编译参数 --disable-lzma --disable-bz2。
$ git clone https://github.com/HuiyangYu/PanDepth.git
$ cd PanDepth
$ rm -rf lib/*a
$ cp /path/to/zlib/lib/libz.a /path/to/libdeflate/lib/libdeflate.a /path/to/htslib/lib/libhts.a lib
$ make
# 编译出的是动态文件
$ ldd bin/pandepth
$ linux-vdso.so.1 (0x0000400038237000)
libstdc++.so.6 => /share/software/app/arm/gcc/10.3.0//lib64/libstdc++.so.6 (0x0000400038254000)
libm.so.6 => /usr/lib64/libm.so.6 (0x000040003843a000)
libgcc_s.so.1 => /share/software/app/arm/gcc/10.3.0//lib64/libgcc_s.so.1 (0x00004000384db000)
libc.so.6 => /usr/lib64/libc.so.6 (0x000040003850c000)
/lib/ld-linux-aarch64.so.1 (0x0000400038212000)
# 编译静态文件
# 在 Makefile 文件中给gcc 添加静态编译选项 -static
$ module load gcc/10.3.0
$ make
$ ldd bin/pandepth
not a dynamic executable
PanGenie¶
编译安装 jellyfish,设置环境变量 PKG_CONFIG_PATH。
$ export PKG_CONFIG_PATH=/path/to/jellyfish/lib/pkgconfig
下载 cereal,设置环境变量 CPATH。
$ git clone https://github.com/USCiLab/cereal
$ export CPATH=/path/to/cereal/include/
$ git clone https://github.com/eblerjana/pangenie.git
$ cd pangenie
tests/catch.hpp 中 所有的 SIGSTKSZ 更改为 32768 (6631行、6599行)。
编译安装
$ mkdir build
$ cd build
$ cmake ..
$ make -j12
$ ls src/
libPanGenieLib.so
PanGenie-index
PanGenie
paragraph¶
项目地址 https://github.com/Illumina/paragraph/
编译boost,因为需要用特殊的编译选项,故不使用已安装好的。
cd ~
wget http://downloads.sourceforge.net/project/boost/boost/1.65.0/boost_1_65_0.tar.bz2
tar xf boost_1_65_0.tar.bz2
cd boost_1_65_0
./bootstrap.sh
./b2 --prefix=/path/to/boost/ link=static install
tar xf paragraph-2.4a.tar.gz
cd paragraph-2.4a
# 注释x86编译选项 -msse4.1
sed -i.bak 's/set_source_files_properties/#set_source_files_properties/g' ./external/CMakeLists.txt
# 将 x86的 sse2 替换为 arm 的相关指令
cp ~/package/sse2neon.h ./external/gssw/
cp ~/package/sse2neon.h ./external/klib/
sed -i.bak 's/<emmintrin.h>/"sse2neon.h"/g' ./external/gssw/gssw.c
sed -i.bak 's/<smmintrin.h>/"sse2neon.h"/g' ./external/gssw/gssw.h
sed -i.bak 's/<emmintrin.h>/"sse2neon.h"/g' ./external/klib/ksw.c
# 编译 warning 报错
sed -i.bak 's/-Werror/ /g' src/cmake/cxx.cmake
mkdir build && cd build
BOOST_ROOT=/path/to/boost/ ../configure --prefix=//path/to/paragrpah
paragraph-2.4a/build)
在 external/graphtools-src/src/graphcore/GraphCoordinates.cpp 中 31 行添加 #include <limits>
将 external/graphtools-src/src/graphIO/GraphJson.cpp 中 71 行的for (const string& label : *labels) 改为 for (const auto& label : *labels)
将 external/graphtools-src/src/graphIO/../../external/include/nlohmann/json.hpp 中 2997 行的 if ('\x00' <= c and c <= '\x1F') 改为 if (static_cast<unsigned char>(c) <= 0x1F)
最后 make,以下报错不用管。
[ 97%] Linking CXX executable ../../../bin/test_blackbox
/usr/bin/ld: CMakeFiles/test_blackbox.dir/test_paragraph.cpp.o:/share/home/software/package/paragrpah/tmp/paragraph-2.4a/src/c++/test-blackbox/test_paragraph.cpp:47: multiple definition of `compare_values'; CMakeFiles/test_blackbox.dir/test_multithreading.cpp.o:/share/home/software/package/paragrpah/tmp/paragraph-2.4a/src/c++/test-blackbox/test_multithreading.cpp:39: first defined here
collect2: error: ld returned 1 exit status
make[2]: *** [src/c++/test-blackbox/CMakeFiles/test_blackbox.dir/build.make:174: bin/test_blackbox] Error 1
make[1]: *** [CMakeFiles/Makefile2:784: src/c++/test-blackbox/CMakeFiles/test_blackbox.dir/all] Error 2
make: *** [Makefile:136: all] Error 2
$ ls -1 bin/
addVariants.py
compare-alignments.py
findgrm.py
graph-to-fasta
grmpy
grmpy-vcf-merge.py
idxdepth
__init__.py
kmerstats
msa2vcf.py
multigrmpy.py
multiparagraph.py
pam
paragraph
paragraph2dot.py
test_grm
vcf2paragraph.py
$ ./bin/paragraph -h
安装python依赖
pip install --prefix=/path/to/paragrpah/ -r requirements.txt
perl¶
需要先编译安装 berkeley-db,否则使用报错 DB_File/DB_File.so: undefined symbol: db_create at ... 。
$ cpanm --reinstall --configure-args='INC="-I/path/to/berkeley-db/5.3.21/include" LIBS="-L/path//berkeley-db/5.3.21/lib -ldb"' -l /path/to/perl/5.34.0/ DB_File
petsc¶
项目地址 https://gitlab.com/petsc/petsc
$ wget https://web.cels.anl.gov/projects/petsc/download/release-snapshots/petsc-3.22.2.tar.gz
$ tar xf petsc-3.22.2.tar.gz
$ cd petsc-3.22.2
$ module load arm/openmpi/5.0.5 arm/openblas/0.3.28
$ ./configure --prefix=//path/to/petsc/ --with-hypre=1 --download-hypre=yes
$ make all check
$ make install
plink¶
项目地址 https://github.com/chrchang/plink-ng https://www.cog-genomics.org/plink/2.0/
- 2.0 版本
将 Makefile 文件的
tar xf plink-ng-2.0.0-a.6.5.tar.gz cd plink-ng-2.0.0-a.6.5/2.0
#BLASFLAGS64 ?= -llapack
改为
BLASFLAGS64 ?= -O2 -L/share/software/app/arm/lapack/3.12.0/ -I/share/software/app/arm/lapack/3.12.0/include/ -llapack -lblas -lgfortran
然后编译 make -j20,生成目标文件 bin/plink2。
- 1.9 版本
tar xf plink-ng-2.0.0-a.6.5.tar.gz
cd plink-ng-2.0.0-a.6.5/1.9
module load arm/zlib/1.3
CFLAGS ?= -Wall -O2 -g -I../2.0/simde
改为
CFLAGS ?= -Wall -O2 -g -I../2.0/simde -D DYNAMIC_ZLIB
将
BLASFLAGS ?= -L/usr/lib64/atlas -llapack -lblas -lcblas -latlas
改为
BLASFLAGS ?= -L/share/software/app/arm/lapack/3.12.0/lib -I/share/software/app/arm/lapack/3.12.0/include/ -llapack -lblas -lcblas -lgfortran
将
#ZLIB ?= -L. ../zlib-1.3.1/libz.so.1.3.1
改为
ZLIB ?= -L. /share/software/app/arm/zlib/1.3/lib/libz.a
然后编译 make -j20,生成目标文件 plink。
plotsr¶
项目地址 https://github.com/schneebergerlab/plotsr
$ git clone https://github.com/schneebergerlab/plotsr.git
$ pip install --prefix=/path/to/plotsr/ numpy==1.21.2 pandas==1.2.4 matplotlib setuptools
$ PYTHONPATH=/path/to/plotsr/lib/python3.9/site-packages/:/path/to/plotsr/lib64/python3.9/site-packages/ python setup.py install --prefix=/path/to/plotsr/
$ cd dist/
$ unzip plotsr-0.0.0-py3.9.egg
$ cp -r plotsr /path/to/plotsr/lib64/python3.9/
$ export PYTHONPATH=/path/to/plotsr/lib/python3.9/site-packages/:/path/to/plotsr/lib64/python3.9/site-packages/
$ export PATH="/path/to/plotsr/bin/:$PATH"
$ plotsr -h
plumed¶
项目地址 https://github.com/plumed/plumed2
# 使用hmpi编译
# 使用 FFTW_ROOT 设置使用 fftw3,否则默认可能使用fftw,导致后面编译gromacs使用fftw3时报错
$ module load arm/hyper-mpi/24.6.30_bisheng arm/jq/1.7.1
$ FFTW_ROOT=/path/to/fftw3/ ./configure --prefix=/path/to/plumed2 --enable-fftw --enable-zlib --enable-mpi CXX=`which mpicxx` CC=`which mpicc`
$ make -j96 && install
python¶
静态编译
安装最新版的zlib、bzip2、xz、readline、ncurse、libffi、sqlite、openssl到同一个目录,然后删掉动态库,只保留静态库,将所有 lib64下的静态库拷贝到lib下。
$ tar xf Python-3.9.19.tgz
$ cd Python-3.9.19
$ ./configure --enable-optimizations --prefix=/python/to/path/ --with-openssl=/libpath/to/lib/ CPPFLAGS="I/libpath/to/include" LDFLAGS="I/libpath/to/lib" LIBS="-Wl,-Bstatic -lncursesw -lpanelw -lz -lbz2 -lreadline -lffi -Wl,-Bdynamic"
$ make -j
....
# make 结束后查看是否有预期未编译的模块
Python build finished successfully!
The necessary bits to build these optional modules were not found:
_dbm _gdbm _tkinter
_uuid nis
To find the necessary bits, look in setup.py in detect_modules() for the module's name.
The following modules found by detect_modules() in setup.py, have been
built by the Makefile instead, as configured by the Setup files:
_abc atexit pwd
time
# 查看这些模块是否链接额外的动态库
$ ldd ./build/lib.linux-x86_64-3.9/{zlib.cpython-39-x86_64-linux-gnu.so,readline.cpython-39-x86_64-linux-gnu.so,_bz2.cpython-39-x86_64-linux-gnu.so,readline.cpython-39-x86_64-linux-gnu.so,_ctypes.cpython-39-x86_64-linux-gnu.so,_sqlite3.cpython-39-x86_64-linux-gnu.so,_curses.cpython-39-x86_64-linux-gnu.so}
./build/lib.linux-x86_64-3.9/zlib.cpython-39-x86_64-linux-gnu.so:
linux-vdso.so.1 (0x00007fff3a9fc000)
libpthread.so.0 => /lib64/libpthread.so.0 (0x00001486580e9000)
libc.so.6 => /lib64/libc.so.6 (0x0000148657d13000)
/lib64/ld-linux-x86-64.so.2 (0x0000148658524000)
./build/lib.linux-x86_64-3.9/readline.cpython-39-x86_64-linux-gnu.so:
linux-vdso.so.1 (0x00007ffd46f94000)
libpthread.so.0 => /lib64/libpthread.so.0 (0x0000152f99393000)
libc.so.6 => /lib64/libc.so.6 (0x0000152f98fbd000)
/lib64/ld-linux-x86-64.so.2 (0x0000152f9982f000)
./build/lib.linux-x86_64-3.9/_bz2.cpython-39-x86_64-linux-gnu.so:
linux-vdso.so.1 (0x00007ffc9b4bf000)
libpthread.so.0 => /lib64/libpthread.so.0 (0x000015043136b000)
libc.so.6 => /lib64/libc.so.6 (0x0000150430f95000)
/lib64/ld-linux-x86-64.so.2 (0x00001504317a1000)
./build/lib.linux-x86_64-3.9/readline.cpython-39-x86_64-linux-gnu.so:
linux-vdso.so.1 (0x00007ffda3bb5000)
libpthread.so.0 => /lib64/libpthread.so.0 (0x000014700bde9000)
libc.so.6 => /lib64/libc.so.6 (0x000014700ba13000)
/lib64/ld-linux-x86-64.so.2 (0x000014700c285000)
./build/lib.linux-x86_64-3.9/_ctypes.cpython-39-x86_64-linux-gnu.so:
linux-vdso.so.1 (0x00007ffcdd2f3000)
libdl.so.2 => /lib64/libdl.so.2 (0x00001466222fc000)
libpthread.so.0 => /lib64/libpthread.so.0 (0x00001466220dc000)
libc.so.6 => /lib64/libc.so.6 (0x0000146621d06000)
/lib64/ld-linux-x86-64.so.2 (0x000014662272a000)
./build/lib.linux-x86_64-3.9/_sqlite3.cpython-39-x86_64-linux-gnu.so:
linux-vdso.so.1 (0x000014dfe9518000)
libpthread.so.0 => /lib64/libpthread.so.0 (0x000014dfe8d76000)
libc.so.6 => /lib64/libc.so.6 (0x000014dfe89a0000)
/lib64/ld-linux-x86-64.so.2 (0x000014dfe92eb000)
./build/lib.linux-x86_64-3.9/_curses.cpython-39-x86_64-linux-gnu.so:
linux-vdso.so.1 (0x00007ffcaf4f1000)
libpthread.so.0 => /lib64/libpthread.so.0 (0x00001531e9136000)
libc.so.6 => /lib64/libc.so.6 (0x00001531e8d60000)
/lib64/ld-linux-x86-64.so.2 (0x00001531e95c6000)
# 测试使用
$ ./python -c "
import sys, zlib, bz2, readline
print('zlib 版本:', zlib.ZLIB_VERSION)
print('bz2 库版本:', bz2.__doc__)
print('readline 动态库路径:', readline.__file__)
"
# 安装
$ make install
$
R¶
项目地址 https://cran.r-project.org/
install=/path/to/r/
export LDFLAGS="-L${install}/lib"
export CFLAGS="-I${install}/include"
export LD_LIBRARY_PATH="${install}/lib/"
# 编译安装 readline
$ wget https://ftp.gnu.org/gnu/readline/readline-8.2.tar.gz
$ tar xf readline-8.2.tar.gz
$ cd readline-8.2/
$ ./configure --prefix=$install
$ make -j20 && make install
# 编译安装 libpsl
$ wget https://github.com/rockdaboot/libpsl/releases/download/0.21.5/libpsl-0.21.5.tar.gz
$ tar xf libpsl-0.21.5.tar.gz
$ cd libpsl-0.21.5/
$ ./configure --prefix=$install
$ make -j20 && make install
# 编译安装 curl
$ wget https://curl.se/download/curl-8.13.0.tar.gz
$ tar xf curl-8.13.0.tar.gz
$ cd curl-8.13.0/
$ ./configure --prefix=$install --with-ssl
$ make -j20 && make install
# 编译 R
$ wget https://cran.r-project.org/src/base/R-4/R-4.5.0.tar.xz
$ tar xf R-4.5.0.tar.xz
$ cd R-4.5.0/
# 如果系统没有安装图形界面,添加选项 --with-x=no
$ ./configure --enable-R-shlib --with-x=no --with-pcre1 --prefix=$install CPPFLAGS="-I$install/include" LDFLAGS="-L$install/lib"
$ make -j20 && make install
将所有依赖库以静态库的形式引用
依赖库 zlib readline libpsl curl ncurses pcre2 openssl bzip2 xz zstd 编译时 加上 -fPIC 选项
export CFLAGS="-O2 -fPIC"
export FFLAGS="-O2 -fPIC"
export FCFLAGS="-O2 -fPIC"
tar xf R-4.4.1.tar.gz
cd R-4.4.1
export PATH="/libpath/to/bin/:$PATH"
$ ./configure --prefix=/path/to/R/ --enable-R-static-lib --enable-static --with-x=no --with-cairo --with-readline=no LDFLAGS="-L/libpath/to/libs -static" CPPFLAGS="-I/libpath/to/include/" LIBS="-lreadline -lncursesw -lcurl -lssl -lcrypto -lpsl -llzma -lbz2 -lz -lrt -lzstd -lm -ldl -lpthread"
configure 执行完成之后,修改生成的 Makeconf 和 etc/Makeconf 文件
将所有的 -static 去掉
将 LIBS = -L/libpath/to/lib -lpcre2-8 -llzma -lbz2 -lz -lrt -ldl -lm -lreadline -lncursesw -lcurl -lssl -lcrypto -lpsl -llzma -lbz2 -lz -lrt -lzstd -lm -ldl -lpthread
替换为
LIBS = -L/libpath/to/lib -Wl,-Bstatic -lpcre2-8 -lreadline -lncursesw -lcurl -lssl -lcrypto -lpsl -llzma -lbz2 -lz -lzstd -Wl,-Bdynamic -lrt -lm -ldl -lpthread
最后在 make -j
RGT¶
项目地址:https://github.com/CostaLab/reg-gen
$ wget wget https://github.com/CostaLab/reg-gen/archive/refs/tags/RGT-1.0.2.tar.gz
$ tar xf RGT-1.0.2.tar.gz
$ cd RGT-1.0.2
# 将 hint THOR 依赖的 hmmlearn==0.2.2 改为 hmmlearn==0.2.7,添加 numpy<1.24
# 将 ./rgt/THOR/neg_bin_rep_hmm.py 中的 from hmmlearn.hmm import _BaseHMM 改为 from hmmlearn.base import _BaseHMM
$ pip install --prefix=/path/to/rgt/ .
rmats¶
项目地址 https://github.com/Xinglab/rmats-turbo
$ tar xf rmats_turbo_v4_3_0.tar.gz
$ cd rmats_turbo_v4_3_0/
$ export FC=gfortran
$ sed -i 's/-msse2//g' rMATS_C/lbfgs_scipy/Makefile
$ sed -i 's/-msse2//g' rMATS_C/Makefile
$ pip install --prefix=/path/to/rmats-turbo/ cython
$ module load arm/r/4.4.1
$ ./build_rmats
# 使用
run_rmats --b1 Y_Adipose_file.txt --b2 E_Adipose_file.txt --gtf Mus_musculus.GRCm39.114.gtf --od rmats/rMATs_Y_E/ --tmp rmats/tmp_WT_CRC/ -t paired --readLength 150 --variable-read-length --cstat 0.0001 --nthread 20
也可以参考 conda 中 rmats 的编译脚本
#!/bin/bash
set -xe
mkdir $PREFIX/rMATS
GSL_LDFLAGS="$(gsl-config --libs)"
GSL_CFLAGS="$(gsl-config --cflags)"
export GSL_LDFLAGS
export GSL_CFLAGS
export LD_LIBRARY_PATH=${PREFIX}/lib
case $(uname -m) in
aarch64)
sed -i.bak -e "s/-msse2//" rMATS_C/Makefile
;;
*)
;;
esac
make -j ${CPU_COUNT}
cp rmats.py $PREFIX/rMATS
cp cp_with_prefix.py $PREFIX/rMATS
mkdir $PREFIX/rMATS/rMATS_C
cp rMATS_C/rMATSexe $PREFIX/rMATS/rMATS_C
cp -R rMATS_P $PREFIX/rMATS
cp -R rMATS_R $PREFIX/rMATS
cp *.so $PREFIX/rMATS
chmod +x $PREFIX/rMATS/rmats.py
ln -s $PREFIX/rMATS/rmats.py $PREFIX/bin/rmats.py
ln -s $PREFIX/rMATS/rMATS_P/FDR.py $PREFIX/bin/FDR.py
ln -s $PREFIX/rMATS/rMATS_P/inclusion_level.py $PREFIX/bin/inclusion_level.py
ln -s $PREFIX/rMATS/rMATS_P/joinFiles.py $PREFIX/bin/joinFiles.py
ln -s $PREFIX/rMATS/rMATS_P/paste.py $PREFIX/bin/paste.py
# for backwards compatibility with the previous recipe, create a symlink named
# for the previously-used executable
ln -s $PREFIX/rMATS/rmats.py $PREFIX/bin/RNASeq-MATS.py
rmblast¶
项目地址:https://www.repeatmasker.org/rmblast/
$ wget https://www.repeatmasker.org/rmblast/rmblast-2.14.0+-x64-linux.tar.gz
$ wget https://www.repeatmasker.org/rmblast/isb-2.14.1+-rmblast.patch.gz
$ tar xf rmblast-2.14.0+-x64-linux.tar.gz
$ gzip -d isb-2.14.1+-rmblast.patch.gz
$ cd ncbi-blast-2.14.1+-src/
$ patch -p1 < ../isb-2.14.1+-rmblast.patch
$ cd c++/
$ ./configure --with-mt --without-debug --without-krb5 --without-openssl --with-projects=scripts/projects/rmblastn/project.lst --prefix=/path/to/rmblast
$ make -j20
$ make install
RNAstructure¶
项目地址 https://rna.urmc.rochester.edu/RNAstructureDownload.html
$ wget https://rna.urmc.rochester.edu/Releases/current/RNAstructureSource.tgz
$ tar xf RNAstructureSource.tgz
$ cd RNAstructure/
$ make all -j20
sambamba¶
项目地址 https://github.com/biod/sambamba
$ wget https://anaconda.org/bioconda/sambamba/1.0.1/download/linux-aarch64/sambamba-1.0.1-hc099486_2.tar.bz2
$ tar xf sambamba-1.0.1-hc099486_2.tar.bz2
$ bin/sambamba
samtools¶
静态编译
注意 ncurses 的编译,见 编译 ncurses。
$ tar xf samtools-1.22.1.tar.gz
$ cd samtools-1.22.1
$ export LDFLAGS="-static"
$ ./configure --prefix=/path/to/samtools/ CURSES_LIB="-L/path/to/ncurses//lib -lncursesw"
$ make -j
$ make install
$ ldd samtools
not a dynamic executable
smcpp¶
项目地址 https://github.com/popgenmethods/smcpp
smcpp 1.15.4 需要使用低版本 numpy ,pip 安装低版本 numpy 时出现下面的报错,使用 --ignore-installed 选项。
Permission denied: '/usr/lib/python3.9/site-packages/__pycache__/six.cpython-39.pyc'
Consider using the `--user` option or check the permissions.
$ module load arm/mpfr/4.1.0
$ pip install --prefix=/path/to/smcpp/1.15.4/ --ignore-installed --no-cache-dir --no-build-isolation "numpy<1.20" appdirs matplotlib pandas pysam scipy setuptools tqdm scikit-learn
$ module load arm/smcpp/1.15.4-py3.9
$ tar xf smcpp-1.15.4.tar.gz
$ cd smcpp-1.15.4
$ python setup.py install --prefix=/path/to/smcpp/1.15.4/
# 去掉告警
$ export PYTHONWARNINGS="ignore::UserWarning:smcpp.version"
$ /path/to/smcpp/1.15.4/bin/smc++ -h
usage: smc++ [-h] {chunk,cite,cv,estimate,plot,posterior,simulate,split,vcf2smc,version} ...
positional arguments:
{chunk,cite,cv,estimate,plot,posterior,simulate,split,vcf2smc,version}
chunk Sample randomly with replacement chunks from data file(s)
cite Print citation information for SMC++
cv Perform cross-validated estimation procedure.
estimate Estimate size history for one population
plot Plot size history from fitted model
posterior Store/visualize posterior decoding of TMRCA
simulate Simulate from a fitted model
split Estimate split time in two population model
vcf2smc Convert VCF to SMC++ format
version Print version string
optional arguments:
-h, --help show this help message and exit
smoothxg¶
项目地址 https://github.com/pangenome/smoothxg
$ git clone --recursive https://github.com/pangenome/smoothxg.git
# 提前编译 abPOA 静态库,否则后面使用cmake编译会出错
$ cd smoothxg/deps/abPOA/
# 更改代码,否则后面编译 smoothxg 会报错
$ sed -i 's/static const char/static const signed char/' src/abpoa_output.c
$ make
$ ls lib/libabpoa.a
# 更改 odgi 相关代码,否则编译 odgi 时会报错
$ cd ../odgi
# 去掉 -msse4.2 编译选项
$ sed -i 's/-msse4.2//g' ./CMakeLists.txt
$ sed -i 's/-msse4.2//g' ./deps/sdsl-lite/CMakeLists.txt
$ sed -i 's/-msse4.2//g' ./deps/mmmulti/deps/sdsl-lite/CMakeLists.txt
$ sed -i 's/arch=x86-64/arch=armv8-a/g' src/main.cpp
CMakeLists.txt,禁止编译 abPOA,使用上面编译好的 abPOA。
将 280 至 291 abPOA 相关的行注释掉,然后在下面添加这 3 行。
set(abpoa_SOURCE_DIR "${CMAKE_SOURCE_DIR}/deps/abPOA")
set(abpoa "${CMAKE_SOURCE_DIR}/deps/abPOA/lib/libabpoa.a")
include_directories("${CMAKE_SOURCE_DIR}/deps/abPOA/include")
将 268 行的 add_dependencies(smoothxg_objs abpoa) 注释掉。
module load arm/jemalloc/5.2.1 arm/gcc/10.3.0
mkdir build && cd build
$ cmake -DCMAKE_C_COMPILER=`which gcc` -DCMAKE_CXX_COMPILER=`which g++` ..
$ make -j20
$ ../bin/smoothxg -h
snoscan¶
wget http://eddylab.org/software/snoscan/snoscan.tar.gz
tar xf snoscan.tar.gz
cd snoscan-0.9.1
cd squid-1.5.11
make clean
make
cd ..
make clean
make
SPAdes¶
项目地址 https://github.com/ablab/spades
tar xf SPAdes-4.1.0.tar.gz
cd SPAdes-4.1.0
PREFIX=/path/to/spades/ ./spades_compile.sh -j 10
sratools¶
sratools编译需要 libstdc++.a 静态链接库,否则编译过程中会出现报错。
/usr/bin/ld: cannot find -lstdc++
collect2: error: ld returned 1 exit status
$ find /usr/ -name "*libstdc++.a*"
libstdc++.a,可自行编译安装 gcc。
sratools 安装依赖 ncbi-vdb。
$ git clone https://github.com/ncbi/ncbi-vdb
$ cd ncbi-vdb
$ ./configure --prefix=/path/to/ncbi-vdb/
$ make -j12 && make install
$ git clone https://github.com/ncbi/sra-tools
$ cd sra-tools
$ ./configure --with-ncbi-vdb-prefix=/path/to/ndcbi-vdb/ --prefix=/path/to/sra-tools/
$ make -j12 && make install
STAR¶
项目地址 https://github.com/alexdobin/STAR
$ git clone https://github.com/alexdobin/STAR
$ cd STAR/source
Makefile 文件中 CXXFLAGS_SIMD ?= -mavx2 整行注释,如下所示。
#CXXFLAGS_SIMD ?= -mavx2
然后 make
stringtie¶
项目地址 https://github.com/gpertea/stringtie
wget https://anaconda.org/bioconda/stringtie/3.0.0/download/linux-aarch64/stringtie-3.0.0-hac7b671_0.tar.bz2
$ tar xf stringtie-3.0.0-hac7b671_0.tar.bz2
$ ./bin/stringtie -h
suiteparse¶
项目地址 https://github.com/DrTimothyAldenDavis/SuiteSparse
tar xf c-7.10.2.tar.gz
cd SuiteSparse-7.10.2/
module load arm/gcc/12.2.0
cmake ../ -DCMAKE_INSTALL_PREFIX=/path/to/suitesparse/ -DCMAKE_C_COMPILER=`which gcc` -DCMAKE_CXX_COMPILER=`which g++` -DMPFR_INCLUDE_DIR=/path/to/mpfr/include/ -DMPFR_LIBRARY=/path/to/mpfr/lib/
make -j20
make install
syri¶
项目地址 https://github.com/schneebergerlab/syri
$ tar xf v1.7.0.tar.gz
$ cd syri-1.7.0
$ pip install --prefix=/path/to/syri/ numpy==1.21 cython==0.29.24 scipy pandas python-igraph psutil pysam matplotlib
$ PYTHONPATH=/path/to/syri/lib/python3.9/site-packages/:/path/to/syri/lib64/python3.9/site-packages/ python setup.py install --prefix=/path/to/syri/
$ export PYTHONPATH=/path/to/syri/lib/python3.9/site-packages/:/path/to/syri/lib64/python3.9/site-packages/:/path/to/syri/lib64/python3.9/site-packages/syri-0.0.0-py3.9-linux-aarch64.egg/
$ export PATH="/path/to/syri/bin/:$PATH"
$ syri -h
TE_finder¶
https://github.com/urgi-anagen/TE_finder
$ tar xf TE_finder-release-2.31.1.tar.gz
$ cd TE_finder-release-2.31.1/
$ mkdir build; cd build
$ cmake -DCPPUNIT_INCLUDE_DIR=/path/to/cppunit/include -DCPPUNIT_LIBRARY=/path/to/cppunit/lib/libcppunit.so ..
$ make -j20
$ make install
TIR-Learner¶
项目地址 https://github.com/lutianyu2001/TIR-Learner
$ tar xf TIR-Learner-3.0.7.tar.gz
$ cd TIR-Learner-3.0.7
$ cp -r TIR-Learner3/ /path/to/tir-learner/
$ pip install --prefix=/path/to/tir-learner/ biopython keras multiprocess pandas regex scikit-learn swifter torch torchvision torchaudio psutil
$ module load arm/blast/2.16.0 arm/genericrepeatfinder/1.0.2 arm/genometools/1.6.5
$ PYTHONPATH=//path/to//tir-learner/lib/python3.9/site-packages/://path/to//tir-learner/lib64/python3.9/site-packages/ /path/to/tir-learner/TIR-Learner3/TIR-Learner.py -h
trinity¶
$ vim ./Inchworm/CMakeLists.txt
set(CMAKE_CXX_FLAGS "${CMAKE_CXX_FLAGS} -pipe -W -Wall -Wpedantic -fopenmp ")
$ vim ./Chrysalis/CMakeLists.txt
set(CMAKE_CXX_FLAGS "${CMAKE_CXX_FLAGS} -pipe -W -Wall -Wpedantic -fopenmp -pthread -m64")
$ vim trinity-plugins/ParaFly/configure
AM_CXXFLAGS=-m64 $OPENMP_CXXFLAGS
cd trinity-plugins/bamsifter/
rm -rf htslib/
wget https://github.com/samtools/htslib/releases/download/1.21/htslib-1.21.tar.bz2
tar xf htslib-1.21.tar.bz2
mv htslib-1.21 htslib
cd -
make
ucsc kent utils¶
$ module load arm/libpng/1.6.44 arm/mariadb/11.6.2
# 不使用 mysql 的 so 动态库
$ cd /dir/to/mariadb/lib
$ mkdir bak; mv *so* bak
# 下载编译 ucsc kent
$ wget https://hgdownload.cse.ucsc.edu/admin/exe/userApps.src.tgz
$ tar xf userApps.src.tgz
$ cd userApps
$ make -j20
$ ls /bin
# 恢复 mysql 的 so 动态库
$ cd /dir/to/mariadb/lib
$ mv bak/* .
$ module purge
ls -1 bin/* >> app_test.sh
# 测试命令,然后在日志中用error关键字搜索,查看是否有执行失败的命令
sh app_test.sh > app_test_log
vasp¶
使用 openblas、scalapack、fftw、gcc、华为MPI(hmpi) 编译 vasp。
openblas编译安装
# 载入 hmpi、gcc
module load gcc/hmpi/hmpi gcc/compiler/gccmodule
mkdir /path/to/vasp/dep/openblas/
tar xf OpenBLAS-0.3.28.tar.gz
cd OpenBLAS-0.3.28
export CC=`which gcc`
export CXX=`which g++`
export FC=`which gfortran`
make
make PREFIX=/path/to/vasp/dep/openblas/ install
使用常规方式安装 scalapack 会有类似 Error: Type mismatch between actual argument at (1) and actual argument at (2) (REAL(8)/REAL(4)) 的报错,因此这里使用专门的安装工具安装 scalapack。
# 安装工具使用python2编写的
module load arm/python/2.7.18
# 载入 hmpi、gcc
module load gcc/hmpi/hmpi gcc/compiler/gccmodule
mkdir /path/to/vasp/dep/scalapack/
wget http://www.netlib.org/scalapack/scalapack_installer.tgz
tar xf scalapack_installer.tgz
cd scalapack_installer/
python ./setup.py --prefix=/path/to/vasp/dep/scalapack/ --mpiincdir=/path/to/hmpi/gcc/hmpi/include/ --downall --notesting
# 载入 hmpi、gcc
module load gcc/hmpi/hmpi gcc/compiler/gccmodule
mkdir /path/to/vasp/dep/fftw/
tar xf fftw-3.3.8.tar.gz
cd fftw-3.3.8/
./configure --prefix=/path/to/vasp/dep/fftw/ --enable-shared --enable-static --enable-fma --enable-neon
make
make install
vasp编译安装
# 载入 hmpi、gcc
module load gcc/hmpi/hmpi gcc/compiler/gccmodule
tar xf vasp.5.4.4.tar.gz
cd vasp.5.4.4
makefile.include 文件,文件内容如下。
这里面加了一个 -fallow-argument-mismatch 编译选项,否则会出现类似 Error: Type mismatch between actual argument at (1) and actual argument at (2) (REAL(8)/REAL(4)) 的报错,导致编译失败。
重点关注高亮的行。
最后 make,编译时间较长。编译成功后可执行文件在 ./bin/ 目录内。
# Precompiler options
CPP_OPTIONS= -DHOST=\"LinuxGNU\" \
-DMPI -DMPI_BLOCK=8000 \
-Duse_collective \
-DscaLAPACK \
-DCACHE_SIZE=4000 \
-Davoidalloc \
-Duse_bse_te \
-Dtbdyn \
-Duse_shmem
CPP = gcc -E -P -C -w $*$(FUFFIX) >$*$(SUFFIX) $(CPP_OPTIONS)
FC = mpif90
FCL = mpif90
FREE = -ffree-form -ffree-line-length-none
FFLAGS = -w -fallow-argument-mismatch
OFLAG = -O3 -ffp-contract=fast -fdec-math -ffpe-trap=invalid,zero,overflow -ffpe-summary=none
OFLAG_IN = $(OFLAG)
DEBUG = -O0
BLAS = -L$(LIBDIR) -lrefblas
LAPACK = -L$(LIBDIR) -ltmglib -llapack
BLACS =
SCALAPACK = -L$(LIBDIR) -lscalapack $(BLACS)
LLIBS = -L//path/to/vasp/dep/scalapack/lib/ -lscalapack -L/path/to/vasp/dep/openblas/lib/ -lopenblas
FFTW ?= /path/to/vasp/dep/fftw/
LLIBS += -L$(FFTW)/lib -lfftw3
INCS = -I$(FFTW)/include
OBJECTS = fftmpiw.o fftmpi_map.o fftw3d.o fft3dlib.o
OBJECTS_O1 += fftw3d.o fftmpi.o fftmpiw.o
OBJECTS_O2 += fft3dlib.o
# For what used to be vasp.5.lib
CPP_LIB = $(CPP)
FC_LIB = $(FC)
CC_LIB = gcc
CFLAGS_LIB = -O
FFLAGS_LIB = -O1
FREE_LIB = $(FREE)
OBJECTS_LIB= linpack_double.o getshmem.o
# For the parser library
CXX_PARS = g++
LIBS += parser
LLIBS += -Lparser -lparser -lstdc++
# Normally no need to change this
SRCDIR = ../../src
BINDIR = ../../bin
参考
https://www.vasp.at/wiki/index.php/Installing_VASP.5.X.X
VCF2Dis¶
项目地址 https://github.com/hewm2008/VCF2Dis
$ tar xf VCF2Dis-1.53.tar.gz
$ cd VCF2Dis-1.53/
$ sh make.sh
$ bin/VCF2Dis -help
vcflib¶
项目地址 https://github.com/vcflib/vcflib
git clone --recursive https://github.com/vcflib/vcflib
cd vcflib
mkdir build
cd build
$ export CMAKE_PREFIX_PATH=/path/to/pybind11/lib/python3.9/site-packages/pybind11/share/cmake/pybind11/:$CMAKE_PREFIX_PATH
# 这里载入之前安装的 htslib,否则 cmake 会自动下载编译 htslib,然后会出现 libcurl 相关的报错
$ module load arm/htslib/1.21 arm/pybind11/2.13.6-py3.9
$ cmake -DCMAKE_BUILD_TYPE=Debug -DZIG=OFF -DWFA_GITMODULE=ON -DCMAKE_INSTALL_PREFIX=/path/to/vcflib/ ..
$ cmake --build . -- -j 20
$ cmake --install .
静态编译
在 CMAakeList.txt 中 PROJECT 之前 添加 set(CMAKE_TRY_COMPILE_TARGET_TYPE STATIC_LIBRARY)
$ git clone --recursive https://github.com/vcflib/vcflib
$ cd vcflib
$ mkdir build && cd build
$ cmake -DCMAKE_BUILD_TYPE=Debug -DZIG=OFF -DWFA_GITMODULE=ON -DCMAKE_INSTALL_PREFIX=/path/to/vcflib/ -DBZIP2_LIBRARIES=/path/to/bzip2/lib/ -DBZIP2_INCLUDE_DIR=/path/to/bzip2/include -DBUILD_STATIC=ON -DCMAKE_CXX_FLAGS=" -pthread -fopenmp -lgomp" ..
# 将 CMakeFiles 下所有的 libwfa2.so.0 替换为 libwfa2.a
$ find CMakeFiles -type f -name link.txt -exec sed -i 's|libwfa2\.so\.0|libwfa2.a|g' {} +
# 将 libwfa2.so.0 替换为 libwfa2.a
$ sed -i 's|libwfa2\.so\.0|libwfa2.a|g' contrib/WFA2-lib/CMakeFiles/align_benchmark.dir/link.txt
# 文件末尾添加编译选项 -Wl,--whole-archive -lpthread -Wl,--no-whole-archive -fopenmp -lgomp
$ sed -i '${s|$| -Wl,--whole-archive -lpthread -Wl,--no-whole-archive -fopenmp -lgomp|}' contrib/WFA2-lib/CMakeFiles/align_benchmark.dir/link.txt
$ make -j VERBOSE=1
# 测试
$ ldd vcfwave
not a dynamic executable
vcfhub¶
项目地址 https://github.com/pangenome/vcfbub
module load arm/llvm/18.1.8 arm/rust/1.85.1
git clone https://github.com/pangenome/vcfbub.git
cd vcfhub
cargo install --path .
./target/release/vcfbub -h
ExaGear 是一款二进制指令动态翻译软件,运行在Arm64服务器上,通过将x86的指令在运行时翻译为Arm64指令并执行,使得绝大部分x86应用无需重新编译就可运行在Arm64服务器上,实现低成本、快速迁移x86应用到Arm64服务器。
集群 arm 各计算节点配置了 ExaGear 二进制指令动态翻译服务,可以在 arm 节点运行 x86 软件,注意软件需要是使用 module 载入的才可以正常使用。
以 admixture 为例,官方只提供了二进制包。
#DSUB -n admixture
#DSUB -R 'cpu=8'
#DSUB -aa
#DSUB --label arm
#DSUB -o admixture_arm_%J.out
module load x86/admixture/1.3.0
admixture --cv filtered_snps.548.maf.ld_prune_in.bed 4 -j8 > filtered_snps.548.maf.ld_prune_in.log.txt
运行时间比较
| 架构 | 运行时间(s) |
|---|---|
| arm | 302 |
| x86 | 176 |
集群主要有2种CPU,arm 128核;intel,64核,型号为 8358P。
hifiasm¶
基因组组装
https://github.com/chhylp123/hifiasm
hifi+ont+hic 组装
$ module load arm/hifiasm/0.19.9
$ time hifiasm -o ath.asm -t 128 --ul CRR302667.fastq.gz --ul-cut 500 --h1 CRR302669_R1.fastq.gz --h2 CRR302669_R2.fastq.gz CRR302668.fastq.gz
| CPU | 核数 | 运行时间(sec) | cputime | 最大内存(MB) |
|---|---|---|---|---|
| arm | 128(整节点) | 59796 | 6144573 | 77027 |
| intel 8358P | 64(整节点) | 115400 | 6620428 | 65546 |
hifi 组装
$ module load arm/hifiasm/0.19.9
$ time hifiasm -o ath.asm -t 128 CRR302668.fastq.gz
| CPU | 核数 | 运行时间(sec) | cputime | 最大内存(MB) |
|---|---|---|---|---|
| arm | 16 | 28497 | 430202 | 41591 |
| arm | 128 | |||
| intel 8358P | 16 | 26689 | 430054 | 91667 |
| AMD 9654 | 192 | 4457 | 445924 |
原生 ARM 和 repack 包
$ export REPACK_EXTRA_OPTS='--bind /path/to/ath_data/ /data'
$ ./repack_ARM/hifiasm-0.19.8-aarch64-conda.rpk -- hifiasm -o ath4.asm -t 16 /data/CRR302668/CRR302668.fastq.gz
| 软件 | 核数 | 运行时间(sec) | cputime | 最大内存(MB) |
|---|---|---|---|---|
| hifiasm | 16 | 28497 | 430202 | 41591 |
| hifiasm-0.19.8-aarch64-conda.rpk | 16 | 28584 | 400963 | 91667 |
BWA¶
短序列比对
$ module load arm/bwa/0.7.18 arm/samtools/1.21
$ bwa mem -t 128 -R '@RG\tID:test146\tPL:illumina\tLB:library\tSM:humen146' hg38.fa ERR194146_1.fastq.gz ERR194146_2.fastq.gz |samtools sort -@ 128 -o $ERR194146_srt.bam"
| CPU | 核数 | 运行运行时间(sec) | cputime | 最大内存(MB) |
|---|---|---|---|---|
| arm | 16 | 34082 | 519789 | 66291 |
| arm | 128(整节点) | 7136 | 602206 | 259437 |
| intel 8358P | 16 | 28999 | 458316 | 66132 |
| intel 8358P | 64(整节点) | 10123 | 510047 | 129309 |
原生 ARM 和 repack 包
$ export REPACK_EXTRA_OPTS='--bind /path/to/WGS/ /data'
$ /share/home/software/tmp/repack_ARM/bwa-0.7.17-aarch64-openEuler20.03.rpk -- bwa mem -t 128 /data/hg38.fa /data/ERR194146_1.fastq.gz /data/ERR194146_2.fastq.gz > ERR194146.sam
| 软件 | 核数 | 运行时间(sec) | cputime | 最大内存(MB) |
|---|---|---|---|---|
| hifiasm | 64(整节点) | 6557 | 586457 | 151514 |
| hifiasm-0.19.8-aarch64-conda.rpk | 64(整节点) | 6655 | 597742 | 151157 |
hisat2¶
RNASeq短序列比对
https://github.com/DaehwanKimLab/hisat2
$ module load arm/hisat/2.1.0 arm/samtools/1.21
$ time hisat2 -p ${thread} -x genome -1 sample_1.fq.gz -2 sample_2.fq.gz | samtools sort -@${thread} -o sample.sorted.bam
| CPU | 核数 | 运行时间(sec) | cputime | 最大内存(MB) |
|---|---|---|---|---|
| arm | 128(整节点) | 378 | 3597 | 23351 |
| arm | 16 | 371 | 3499 | 17031 |
| intel 8358P | 64(整节点) | 499 | 29164 | 23153 |
| intel 8358P | 16 | 288 | 4262 | 17070 |
GATK HaplotypeCaller¶
变异检测
https://github.com/broadinstitute/gatk
$ module load arm/gatk/4.6.0.0
$ gatk HaplotypeCaller --native-pair-hmm-threads $thread -R hg38.fa -L chr20 -I ERR194146_sort_redup.bam -O ERR194146_chr20_arm.vcf.gz
| CPU | 核数 | 运行时间(sec) | cputime | 最大内存(MB) |
|---|---|---|---|---|
| arm | 128(整节点) | 14458 | 14503 | 1938 |
| arm(GKL优化) | 128 | 1476 | 10124 | 1938 |
| intel 8358P | 64(整节点) | 1181 | 26289 | 1851 |
| process/runtime(sec) | AMD 9654, 192c | intel 8358P, 64c | intel 6150, 36c | ARM, 128c |
|---|---|---|---|---|
| bwa + samtools sort | 661 | 922 | 1575 | 729 |
| gatk markduplicates | 393 | 871 | 859 | 984 |
| gatk haplotypecaller | 403 | 515 | 886 | 4503 |
| total | 1457 | 2308 | 3320 | 6216 |
vg¶
泛基因组比对和变异检测等
$ module load arm/vg/1.60.0
$ time vg giraffe -p -t $thread -Z ref.giraffe.gbz -d ref.dist -m ref.min -f sampleid_clean_1.fq.gz -f sampleid_clean_2.fq.gz > sampleid.gam
| CPU | 核数 | 运行时间(sec) | cputime | 最大内存(MB) |
|---|---|---|---|---|
| arm | 128(整节点) | 222 | 25889 | 9771 |
| arm | 16 | 1243 | 20284 | 5313 |
| intel 8358P | 64(整节点) | 327 | 19687 | 7018 |
| intel 8358P | 16 | 996 | 16260 | 5312 |
原生 ARM 和 repack 包
| 软件 | 核数 | 运行时间(sec) | cputime | 最大内存(MB) |
|---|---|---|---|---|
| vg(1.60.0) | 128 | 222 | 25889 | 9771 |
| vg-1.56.0-aarch64-conda.rpk | 128 | 194 | 22179 | 11437 |
STAR¶
RNASeq 短序列比对
https://github.com/alexdobin/STAR
$ module load arm/star/2.7.11b arm/samtools/1.21
$ STAR --genomeDir star_index/ --runThreadN 128 --readFilesIn CRR232282_f1.fastq.gz CRR232282_r2.fastq.gz --readFilesCommand zcat --outFileNamePrefix CRR232282 --outSAMtype BAM SortedByCoordinate --outBAMsortingThreadN $128
| CPU | 核数 | 运行时间(sec) | cputime | 最大内存(MB) |
|---|---|---|---|---|
| arm | 128(整节点) | 219 | 4424 | 39745 |
| arm | 16 | 280 | 4176 | 23199 |
| intel 8358P | 64(整节点) | 154 | 4852 | 30225 |
| intel 8358P | 16 | 314 | 4768 | 23141 |
megahit¶
宏基因组组装
https://github.com/voutcn/megahit
$ module load arm/megahit/1.2.9
$ megahit -1 sampleid_clean_R1.fq.gz -2 sampleid_clean_R2.fq.gz -o out --presets meta-large --no-mercy --kmin-1pass -t 128
| CPU | 核数 | 运行时间(sec) | cputime | 最大内存(MB) |
|---|---|---|---|---|
| arm | 128(整节点) | 48623 | 4575263 | 69953 |
| intel 8358P | 64(整节点) | 61315 | 3362805 | 70090 |
minimap2¶
三代长序列比对
https://github.com/lh3/minimap2
$ module load arm/minimap2/2.28 arm/samtools/1.21
$ minimap2 -ax map-pb -t 128 MH63.fa MH63WGS_all_subreads.fasta.gz |samtools sort -@128 -o MH63WGS_all_subreads.bam
| CPU | 核数 | 运行时间(sec) | cputime | 最大内存(MB) |
|---|---|---|---|---|
| arm | 128(整节点) | 371 | 32222 | 89353 |
| arm | 16 | 1974 | 31654 | 23143 |
| intel 8358P | 64(整节点) | 452 | 25266 | 72995 |
| intel 8358P | 16 | 1408 | 22713 | 23402 |
lammps¶
分子动力学模拟
https://github.com/lammps/lammps
参考 https://docs.hpc.sjtu.edu.cn/app/engineeringscience/lammps.html
运行时间越短越好
| CPU | 核数 | 运行时间(sec) | cputime(sys) | 最大内存(MB) |
|---|---|---|---|---|
| arm | 64(单节点) | 198 | 12410(169) | 1012 |
| arm | 128(单节点) | 98 | 12083(265) | 1360 |
| arm | 128(4节点) | 194 | 10985(3883) | 1360 |
| arm | 256(2节点) | 103 | 8587(4172) | 1114 |
| arm | 512(多节点) | 96 | 5067(3272) | 642 |
gromacs¶
分子动力学模拟
https://github.com/gromacs/gromacs
参考 https://docs.hpc.sjtu.edu.cn/app/engineeringscience/gromacs.html
单位:ns/day,越高越好
| 核数 | 64(单节点) | 128(单节点) | 256(2节点) | 512 |
|---|---|---|---|---|
| Performance | 10.160 | 18.745 | 16.256 |
vllm¶
参考
https://vllm-ascend.readthedocs.io/en/latest/installation.html
安装 nnal,否则后面 vllm 运行报错 libatb.so: cannot open shared object file: No such file or directory。
# root 运行
# https://github.com/vllm-project/vllm-ascend/issues/152
$ wget https://ascend-repo.obs.cn-east-2.myhuaweicloud.com/CANN/CANN%208.0.0/Ascend-cann-nnal_8.0.0_linux-aarch64.run
$ chmod +x ./Ascend-cann-nnal_8.0.0_linux-aarch64.run
# root安装
$ ./Ascend-cann-nnal_8.0.0_linux-aarch64.run --install-for-all --install
$ source /usr/local/Ascend/nnal/atb/set_env.sh
$ source /usr/local/Ascend/ascend-toolkit/set_env.sh
# 普通账号安装
# install torch_npu
$ wget https://pytorch-package.obs.cn-north-4.myhuaweicloud.com/pta/Daily/v2.5.1/20250218.4/pytorch_v2.5.1_py39.tar.gz
$ tar xf pytorch_v2.5.1_py39.tar.gz
$ pip install --upgrade torch_npu-2.5.1.dev20250218-cp39-cp39-manylinux_2_17_aarch64.manylinux2014_aarch64.whl
# Install vllm from source
$ git clone --depth 1 --branch v0.7.1 https://github.com/vllm-project/vllm
$ cd vllm
# VLLM_TARGET_DEVICE=empty pip install . --extra-index-url https://download.pytorch.org/whl/cpu/
# Install vllm-ascend from source
git clone --depth 1 --branch v0.7.1rc1 https://github.com/vllm-project/vllm-ascend.git
cd vllm-ascend
pip install -e . --extra-index-url https://download.pytorch.org/whl/cpu/
测试是否可用
# 设置进程可用的卡ID
$ export ASCEND_RT_VISIBLE_DEVICES=4,5,6,7
$ export NPU_VISIBLE_DEVICES=4,5,6,7
# 从 MODELSCOPE 下载模型
$ VLLM_USE_MODELSCOPE=true
$ vllm serve Qwen/Qwen2.5-0.5B-Instruct
.
.
.
INFO 02-26 01:36:24 launcher.py:19] Available routes are:
INFO 02-26 01:36:24 launcher.py:27] Route: /openapi.json, Methods: GET, HEAD
INFO 02-26 01:36:24 launcher.py:27] Route: /invocations, Methods: POST
INFO: Started server process [1923472]
INFO: Waiting for application startup.
INFO: Application startup complete.
INFO: Uvicorn running on http://0.0.0.0:8000 (Press CTRL+C to quit)
$ curl http://localhost:8000/v1/models
测试 DeepSeek-R1-Distill-Qwen-32B,速度非常慢,3.2 tokens/s,curl 命令测试将近 3 分钟才返回结果。同节点上 mindie 部署的 DeepSeek 70B 模型推理服务,1 秒钟不到即可返回结果。
$ source /usr/local/Ascend/nnal/atb/set_env.sh
$ VLLM_USE_MODELSCOPE=true NPU_VISIBLE_DEVICES=4,5,6,7 ASCEND_RT_VISIBLE_DEVICES=4,5,6,7 vllm serve --tensor-parallel-size 4 --dtype bfloat16 deepseek-ai/DeepSeek-R1-Distill-Qwen-32B
$ curl http://localhost:8000/v1/completions \
-H "Content-Type: application/json" \
-d '{
"model": "deepseek-ai/DeepSeek-R1-Distill-Qwen-32B",
"prompt": "描述一下北京的秋天",
"max_tokens": 512
}'
ascend life science 相关仓库¶
https://modelers.cn/user/Ascend-AI4S
https://ai.gitcode.com/zone/ai4s/models
https://gitcode.com/AI4Science/LifeScience
Warning
以下所有项目,如无特殊说明,均在 AI-01 节点运行,使用前向管理员申请 AI 队列使用权限,并在群共享表格 超算GPU-NPU使用登记 中登记使用情况。
xfold¶
evo2¶
项目核心定位¶
Evo2 是一款先进的基因组建模与设计工具,基于前沿深度学习架构(StripedHyena 2)构建,专注于 DNA、RNA 及蛋白质的序列预测与设计任务,支持单核苷酸分辨率建模,最大上下文窗口可达 100 万碱基对,是生物信息学研究与实践的高效辅助工具。
核心功能¶
-
序列生成:通过 DNA 序列或物种提示,生成目标序列并标注编码区;
-
序列评分:对输入 DNA 序列进行困惑度与核苷酸熵值评估;
-
辅助可视化:支持原核生物序列的 3D 蛋白质可视化(基于 ESMFold)。
链接¶
-
线上试用地址:Evo Designer(无需部署,直接体验序列生成与评分)
-
项目开源地址:GitHub - ArcInstitute/evo2(含完整代码、示例笔记本、本地部署教程)
- 昇腾版项目地址: 待补充
使用¶
# 登录节点下载适配好的的evo2镜像 (已下载则忽略此步骤)
$ wget https://obs-whaicc-fae-public.obs.cn-central-221.ovaijisuan.com:443/singularity-img/ai4s/singularity-evo2.03.tar
# 解压
$ tar -xvf singularity-evo2.03.tar && cd singularity-evo2.03
# 交互模式进入ai节点
$ dsub -R 'npu=1,cpu=4' -aa -q AI -I bash
# 载入 singularity
$ module load arm/singularity/4.1.5
# 测试运行
$ bash run.sh
esm2¶
原版项目地址 https://github.com/facebookresearch/esm
昇腾版项目地址 https://modelers.cn/models/Ascend-AI4S/ESM2
自行安装环境¶
# 交互模式进入ai节点
$ dsub -R 'npu=1,cpu=4' -aa -q AI -I bash
# 载入 npu 版 pytorch
$ module load arm/python/3.11.11 arm/torch_npu/2.3.1.post4-python311
# 安装基础依赖,默认安装路径为 ~/.local/lib/python3.11/site-packages/
$ pip3.11 install pyyaml decorator attrs matplotlib
# 安装 ESM 推理模块
$ pip3.11 install fair-esm
$ pip3.11 install "fair-esm[esmfold]"
$ pip3.11 install torch_npu/2.3.1.post4-python311
$ pip3.11 install "numpy<2.0"
使用集群环境¶
# 交互模式进入ai节点
$ dsub -R 'npu=1,cpu=4' -aa -q AI -I bash
$ module load arm/esm2/1.0-py3.11
测试¶
示例1¶
提取蛋白质序列的残基级与整链级特征,并画出自注意力预测的残基接触图。
import torch
import torch_npu
import esm
# use ascend npu
device = torch.device('npu' if torch_npu.npu.is_available() else 'cpu')
print(f"using device:{device}")
# Load ESM-2 model
model, alphabet = esm.pretrained.esm2_t33_650M_UR50D()
model = model.to(device)
batch_converter = alphabet.get_batch_converter()
model.eval() # disables dropout for deterministic results
# Prepare data (first 2 sequences from ESMStructuralSplitDataset superfamily / 4)
data = [
("protein1", "MKTVRQERLKSIVRILERSKEPVSGAQLAEELSVSRQVIVQDIAYLRSLGYNIVATPRGYVLAGG"),
("protein2", "KALTARQQEVFDLIRDHISQTGMPPTRAEIAQRLGFRSPNAAEEHLKALARKGVIEIVSGASRGIRLLQEE"),
("protein2 with mask","KALTARQQEVFDLIRD<mask>ISQTGMPPTRAEIAQRLGFRSPNAAEEHLKALARKGVIEIVSGASRGIRLLQEE"),
("protein3", "K A <mask> I S Q"),
]
batch_labels, batch_strs, batch_tokens = batch_converter(data)
batch_tokens = batch_tokens.to(device)
batch_lens = (batch_tokens != alphabet.padding_idx).sum(1)
# Extract per-residue representations (on CPU)
with torch.no_grad():
results = model(batch_tokens, repr_layers=[33], return_contacts=True)
token_representations = results["representations"][33]
# Generate per-sequence representations via averaging
# NOTE: token 0 is always a beginning-of-sequence token, so the first residue is token 1.
sequence_representations = []
for i, tokens_len in enumerate(batch_lens):
sequence_representations.append(token_representations[i, 1 : tokens_len - 1].mean(0))
# Look at the unsupervised self-attention map contact predictions
import matplotlib.pyplot as plt
for idx,((_, seq), tokens_len, attention_contacts) in enumerate(zip(data, batch_lens, results["contacts"])):
plt.figure()
plt.matshow(attention_contacts[: tokens_len, : tokens_len].cpu().numpy())
plt.title(seq)
plt.savefig(f"plot_{idx}.png")
python pretrain.py,当前目录下会生成几个 png 文件。
示例2¶
提取蛋白质序列特征
import torch
import torch_npu
import esm
device = torch.device('npu' if torch_npu.npu.is_available() else 'cpu')
#device = torch.device('npu')
print(f"using device:{device}")
# 加载预训练模型
model, alphabet = esm.pretrained.esm2_t33_650M_UR50D()
model = model.to(device)
model.eval() # 推理模式
batch_converter = alphabet.get_batch_converter()
# 准备蛋白质序列数据
data = [("protein1", "MKTVRQERLKSIVRILERSKEPVSGAQLAEELSVSRQVIVQDIAYLRSLGYNIVATPRGYVLAGG")]
batch_labels, batch_strs, batch_tokens = batch_converter(data)
batch_tokens = batch_tokens.to(device) # <= 输入也要 to(device)
# 提取序列特征
with torch.no_grad():
results = model(batch_tokens, repr_layers=[33])
token_representations = results["representations"][33]
print(f"提取的特征形状: {token_representations.shape}")
python feature.py。
模型说明¶
ESM-2 一共有多个版本,主要区别在于:层数(depth)、参数量(size)、推理速度和精度权衡。这些版本都遵循相同的 Transformer 编码器架构,只是在大小和计算能力上有差异。
模型命名:esm2_t<层数>_<参数量>_UR50D
t<层数>:比如 t12 表示有 12 层 Transformer 编码器。<参数量>:大概的参数数量,比如 35M, 3B 等。UR50D:代表训练用的数据集(Uniref50),是去冗余后的蛋白质数据库。
| 模型名称(Hugging Face 名) | 层数 | 参数量 | 说明 |
|---|---|---|---|
| esm2_t6_8M_UR50D | 6 | 8M | 极小模型,适合快速原型,快速测试 / 教学 / CPU 调用 |
| esm2_t12_35M_UR50D | 12 | 35M | 中等小型,推荐用于入门任务,快速测试 / 教学 / CPU 调用 |
| esm2_t30_150M_UR50D | 30 | 150M | 中等模型,效果与效率平衡,标准下游任务建模 |
| esm2_t33_650M_UR50D | 33 | 650M | 较大模型,适合更复杂任务,结构预测 / 蛋白功能预测 |
| esm2_t36_3B_UR50D | 36 | 3B | 超大模型,强大建模能力,结构预测 / 蛋白功能预测 |
| esm2_t48_15B_UR50D | 48 | 15B | 最大模型,性能最强但最重,结构预测 / 蛋白功能预测 |
参考资料¶
esmfold¶
项目地址 https://ai.gitcode.com/zzhihaoo/ESMfold
使用¶
# 交互模式进入ai节点
$ dsub -R 'npu=1,cpu=4' -aa -q AI -I bash
$ module load arm/singularity/4.1.5
$ singularity exec -B /usr/local/Ascend/driver:/usr/local/Ascend/driver:ro -B /usr/local/Ascend/ascend-toolkit/8.2.RC1/opp:/usr/local/Ascend/opp:ro /share/software/image/arm/esmfold.2.0.1.sif esm-fold -i test.fasta -o .
>test
MKTVRQERLKSIVRILERSKEPVSGAQLAEELSVSRQVIVQDIAYLRSLGYNIVATPRGYVLAGG
esm3¶
LigandMPNN¶
项目地址 https://gitcode.com/AI4Science/LifeScience/blob/main/PyTorch/LigandMPNN
自行安装环境¶
# 交互模式进入ai节点
$ dsub -R 'npu=1,cpu=4' -aa -q AI -I bash
# 默认安装路径 ~/.local/lib/python3.11/site-packages/
$ module load arm/python/3.11.11
# 安装torch-npu
$ pip3.11 install torch-npu==2.3.1.post4
# 拉取模型代码
git clone https://gitcode.com/AI4Science/LifeScience.git
cd LifeScience/PyTorch/LigandMPNN
git clone https://github.com/dauparas/LigandMPNN.git && cd LigandMPNN
git checkout 26ec57ac976ade5379920dbd43c7f97a91cf82de
git apply ../patch/LigandMPNN.patch
# 安装环境依赖
$ pip3.11 install -r requirements.txt
bash get_model_params.sh "./model_params"。
测试数据目录 inputs。
推理 bash run_examples.sh。
使用集群环境¶
使用集群预装好的环境
# 交互模式进入ai节点
$ dsub -R 'npu=1,cpu=4' -aa -q AI -I bash
# 下载推理代码
git clone https://gitcode.com/AI4Science/LifeScience.git
cd LifeScience/PyTorch/LigandMPNN
# 加载环境
$ module load arm/ligandmpnn/1.0-py3.11
# 下载模型权重
$ bash get_model_params.sh "./model_params"
# 推理,数据在 inputs 下
$ bash run_examples.sh
BioGPT¶
项目地址 https://modelers.cn/models/Ascend-AI4S/BioGPT
使用集群环境¶
# 交互模式进入ai节点
$ dsub -R 'npu=1,cpu=4' -aa -q AI -I bash
# 将运行脚本拷贝至自己的目录下
$ cp /share/software/image/arm/singularity-biogpt-02/run.sh .
# 运行
$ sh run.sh "Immunotherapy for breast cancer"
...
输入文本:Immunotherapy for breast cancer
开始生成(最大长度:1024,束宽:5)
==================================================
生成结果:
==================================================
Immunotherapy for breast cancer: current status and future prospects for the treatment of metastatic disease. (1) Breast Cancer Immunotherapy: Current Status and Future Prospects for The Treatment of Metastatic Disease: (2) The Role of Immunotherapy in the Management of Early-Stage and Locally Advanced Breast Cancers: A Systematic Review and Meta-Analysis of the Literature (3) Immunotherapy as an Adjuvant Therapy for Patients with High-Risk, Hormone Receptor-Positive, Human Epidermal Growth Factor Receptor 2 (HER2) -Negative, Estrogen Receptor (ER) -negative, and Her2 / neu-Overexpressing (HR + / HER2-) Invasive Breast Carcinomas.
==================================================
RFdiffusion¶
原项目地址 https://github.com/RosettaCommons/RFdiffusion
昇腾项目地址 https://modelers.cn/models/Ascend-AI4S/RFdiffusion
使用集群环境¶
# 交互模式进入ai节点
$ dsub -R 'npu=1,cpu=4' -aa -q AI -I bash
$ module load arm/rfdiffsion/ascend
$ ln -s /share/software/app/arm/rfdiffsion_pyg_ascend/{examples,ops,rfdiffusion,scripts,config,models} .
# 测试运行
$ sh examples/design_unconditional.sh
ProteinMPNN¶
项目地址 https://modelers.cn/models/Ascend-AI4S/ProteinMPNN
自行安装环境¶
# 交互模式进入ai节点
$ dsub -R 'npu=1,cpu=4' -aa -q AI -I bash
# 安装 torch-npu 及相关依赖
$ module load arm/python/3.11.11
$ pip3.11 install torch-npu==2.6.0
$ pip3.11 install "numpy<2.0" pyyaml
# 安装mx_driving,用于训练,推理可以不要
$ pip3.11 install wheel setuptools decorator scipy attrs psutil cloudpickle synr==0.5.0 tornado protobuf==4.25.8
$ git clone https://gitee.com/ascend/DrivingSDK.git
$ cd DrivingSDK
$ source /usr/local/Ascend/ascend-toolkit/set_env.sh
$ export CPATH="$HOME/.local/lib/python3.11/site-packages/torch_npu/include/"
$ export LIBRARY_PATH="$HOME/.local/lib/python3.11/site-packages/torch_npu/lib:$LIBRARY_PATH"
$ bash ci/build.sh --python=3.11
$ pip3.11 install --force-reinstall dist/mx_driving-1.0.0+git3e3c655-cp311-cp311-linux_aarch64.whl
# 下载 ProteinMPNN
$ git clone https://modelers.cn/Ascend-AI4S/ProteinMPNN.git
使用集群环境¶
# 交互模式进入ai节点
$ dsub -R 'npu=1,cpu=4' -aa -q AI -I bash
$ module load arm/proteinmpnn/1.0-py3.11
推理/训练¶
下载 ProteinMPNN 相关代码和权重
$ git clone https://modelers.cn/Ascend-AI4S/ProteinMPNN.git
$ cd ProteinMPNN/training/
$ bash test_inference.sh
# 下载数据集
$ wget https://files.ipd.uw.edu/pub/training_sets/pdb_2021aug02.tar.gz
# 解压数据集
$ tar -xzf pdb_2021aug02.tar.gz
$ cd ProteinMPNN/training/
$ bash submit_exp_020.sh
报错¶
重装 torch_npu¶
报错
$ python test.py
Traceback (most recent call last):
File "/share/home/software/package/esm2/pretrained.py", line 2, in <module>
import torch_npu
File "/share/software/app/arm/esm2/1.0/lib/python3.11/site-packages/torch_npu/__init__.py", line 12, in <module>
from torch.distributed.fsdp import sharded_grad_scaler
File "/share/software/app/arm/esm2/1.0/lib/python3.11/site-packages/torch/distributed/fsdp/__init__.py", line 1, in <module>
from ._flat_param import FlatParameter as FlatParameter
File "/share/software/app/arm/esm2/1.0/lib/python3.11/site-packages/torch/distributed/fsdp/_flat_param.py", line 30, in <module>
from torch.distributed.fsdp._common_utils import (
File "/share/software/app/arm/esm2/1.0/lib/python3.11/site-packages/torch/distributed/fsdp/_common_utils.py", line 35, in <module>
from torch.distributed.fsdp._fsdp_extensions import FSDPExtensions
File "/share/software/app/arm/esm2/1.0/lib/python3.11/site-packages/torch/distributed/fsdp/_fsdp_extensions.py", line 8, in <module>
from torch.distributed._tensor import DeviceMesh, DTensor
File "/share/software/app/arm/esm2/1.0/lib/python3.11/site-packages/torch/distributed/_tensor/__init__.py", line 6, in <module>
import torch.distributed._tensor.ops
File "/share/software/app/arm/esm2/1.0/lib/python3.11/site-packages/torch/distributed/_tensor/ops/__init__.py", line 2, in <module>
from .embedding_ops import * # noqa: F403
^^^^^^^^^^^^^^^^^^^^^^^^^^^^
File "/share/software/app/arm/esm2/1.0/lib/python3.11/site-packages/torch/distributed/_tensor/ops/embedding_ops.py", line 8, in <module>
import torch.distributed._functional_collectives as funcol
File "/share/software/app/arm/esm2/1.0/lib/python3.11/site-packages/torch/distributed/_functional_collectives.py", line 12, in <module>
from . import _functional_collectives_impl as fun_col_impl
File "/share/software/app/arm/esm2/1.0/lib/python3.11/site-packages/torch/distributed/_functional_collectives_impl.py", line 36, in <module>
from torch._dynamo import assume_constant_result
File "/share/software/app/arm/esm2/1.0/lib/python3.11/site-packages/torch/_dynamo/__init__.py", line 2, in <module>
from . import convert_frame, eval_frame, resume_execution
File "/share/software/app/arm/esm2/1.0/lib/python3.11/site-packages/torch/_dynamo/convert_frame.py", line 40, in <module>
from . import config, exc, trace_rules
File "/share/software/app/arm/esm2/1.0/lib/python3.11/site-packages/torch/_dynamo/trace_rules.py", line 50, in <module>
from .variables import (
File "/share/software/app/arm/esm2/1.0/lib/python3.11/site-packages/torch/_dynamo/variables/__init__.py", line 34, in <module>
from .higher_order_ops import (
File "/share/software/app/arm/esm2/1.0/lib/python3.11/site-packages/torch/_dynamo/variables/higher_order_ops.py", line 13, in <module>
import torch.onnx.operators
File "/share/software/app/arm/esm2/1.0/lib/python3.11/site-packages/torch/onnx/__init__.py", line 46, in <module>
from ._internal.exporter import ( # usort:skip. needs to be last to avoid circular import
ImportError: cannot import name 'DiagnosticOptions' from 'torch.onnx._internal.exporter' (/share/software/app/arm/esm2/1.0/lib/python3.11/site-packages/torch/onnx/_internal/exporter/__init__.py)
$ pip3.11 uninstall -y torch torch_npu
$ rm -rf /share/software/app/arm/esm2/1.0/lib/python3.11/site-packages/torch*
$ pip3.11 install torch-npu==2.3.1.post4
程序被终止¶
交互模式下(dsub -R 'npu=1,cpu=4' -aa -q AI -I bash) 运行 AI 相关程序,如果程序运行一段时间后突然被终止并被退出了 AI 节点,同时出现如下 killed 的提示,则可能是程序内存(非NPU显存)使用较大,超过了作业能使用的内存限制,建议调高作业核数,如 dsub -R 'npu=1,cpu=10' -aa -q AI -I bash,多瑙作业内存限制见 多瑙使用-内存限制。
Killed
[username@AI-01 esmfold]
安装¶
由于 glibc 版本的问题,ORCA安装会有点麻烦,参考 ORCA 安装
算例¶
算例1 test1.inp,简单算例
! BLYP D3 def2-TZVP def2/J noautostart miniprint nopop
* xyz 0 1
C 0.0 0.0 0.0
O 0.0 0.0 1.13
*
算例2 benzene_opt_freq.inp,复杂算例,设置使用120个进程。
! B3LYP 6-31G* Opt Freq
%pal
nprocs 120
end
* xyz 0 1
C 0.000000 0.000000 0.000000
C 1.400000 0.000000 0.000000
C 2.100000 1.210000 0.000000
C 0.700000 2.420000 0.000000
C -0.700000 2.420000 0.000000
C -2.100000 1.210000 0.000000
C -2.100000 -1.210000 0.000000
C -0.700000 -2.420000 0.000000
C 0.700000 -2.420000 0.000000
H 1.400000 2.420000 0.000000
H -1.400000 2.420000 0.000000
H -2.800000 1.210000 0.000000
H -2.800000 -1.210000 0.000000
H -1.400000 -2.420000 0.000000
H 1.400000 -2.420000 0.000000
H 2.800000 -1.210000 0.000000
H 2.800000 1.210000 0.000000
*
运行测试¶
单节点单进程¶
运行算例1,默认只能单进程
#DSUB -n orca
#DSUB -N 1
#DSUB -nn 1
#DSUB --mpi openmpi
#DSUB -aa
#DSUB -q arm
#DSUB -o orca_arm_%J.out
module load arm/orca/6.0.1
/share/software/app/arm/orca/6.0.1/orca test1.inp
单节点多进程¶
运行算例2,利用 openmpi 单节点运行 120 个进程。
注意算例里设置的进程数需要与作业申请的核数一致,否则运行报错。
#DSUB -n orca
#DSUB -N 120
#DSUB -nn 1
#DSUB --mpi openmpi
#DSUB -aa
#DSUB -q arm
#DSUB -o orca_arm_%J.out
module load arm/orca/6.0.1
# 以下三行打印hostfile文件,用于debug,正常运行可以不需要
echo $CCS_MPI_OPTIONS
hostfile=$(echo $CCS_MPI_OPTIONS| sed s'/-hostfile //g')
cat $hostfile
/share/software/app/arm/orca/6.0.1/orca benzene_opt_freq.inp
多节点多进程¶
运行算例2,利用 openmpi 多节点运行 120 个进程,这里使用了 2 个节点,每个节点 60 个进程。
注意算例里设置的进程数需要与作业申请的进程数一直,否则运行报错。
#DSUB -n orca
#DSUB -N 120
#DSUB -nn 2
#DSUB --mpi openmpi
#DSUB -o orca_arm_%J.out
module load arm/orca/6.0.1
# 以下三行打印hostfile文件,用于debug,正常运行可以不需要
echo $CCS_MPI_OPTIONS
hostfile=$(echo $CCS_MPI_OPTIONS| sed s'/-hostfile //g')
cat $hostfile
# ORCA 运行目录内需要有与输入文件同名的nodes文件才能启动多节点
# 相当于给mpirun 添加了参数 --hosftfile benzene_opt_freq.nodes
cp $hostfile benzene_opt_freq.nodes
# 其它 mpirun 运行参数可以通过orca命令最后的 "arg1 arg2 arg3 ... " 传给 mpirun
/share/software/app/arm/orca/6.0.1/orca benzene_opt_freq.inp "-x LD_LIBRARY_PATH -x UCX_TLS=shm,ud"
参考
nextdenovo 已适配多瑙调度器,由于计算节点目前没有安装 dsub 提交命令,因此需要在计算节点运行 nextdenovo 程序。
$ module load arm/nextdenovo/2.5.2
$ nohup nextDenovo run.cfg &
run.cfg 如下
[General]
job_type = donau
job_prefix = nextDenovo
task = all # 'all', 'correct', 'assemble'
rewrite = yes # yes/no
rerun = 3
parallel_jobs = 200
input_type = raw
input_fofn = input.fofn
read_type = ont
workdir = 01_rundir
[correct_option]
read_cutoff = 1k
genome_size = 650m
blocksize = 3g
pa_correction = 250
sort_options = -m 20g -t 8 -k 40
minimap2_options_raw = -x ava-ont -t 8
correction_options = -p 8
[assemble_option]
random_round = 20
minimap2_options_cns = -x ava-ont -t 8 -k17 -w17
nextgraph_options = -a 1
由于VASP为商业软件,需要用户自行申请license、在官网自行下载源码包。
编译安装¶
如用户需要在 arm 机器上自行编译,参考 在 arm 上编译 vasp。
测试运行¶
下载测试算例 http://sobereva.com/attach/455/benchmark.Hg.tar.gz,这是个含50个Hg原子的标准测试任务。
解压后有一个 IN-short 和 IN-long,分别是一个耗时较短和一个耗时较长任务的 INCAR 文件。
#DSUB -n vasp
#DSUB -N 256
#DSUB -nn 2
#DSUB --mpi hmpi
#DSUB -o vasp_%J.out
# print hostfile
echo $CCS_MPI_OPTIONS
hostfile=$(echo $CCS_MPI_OPTIONS| sed s'/-hostfile //g')
cat $hostfile
which mpirun
tar xf benchmark.Hg.tar.gz
cd vasp.Hg
# 这里使用 IN-short
cp IN-short INCAR
mpirun $CCS_MPI_OPTIONS --bind-to core -x OMP_NUM_THREADS=1 vasp_std
作业运行完成后,检查输出文件 OUTCAR 内容是否正常!
参考
| CPU | 核数 | 运行时间(sec) | cputime(sys) |
|---|---|---|---|
| arm | 256(2节点) | 976 | |
| arm | 256(4节点) | 1213 | |
| arm | 128(2节点) | 476 | |
| arm | 128(1节点) | 422 | |
| arm | 64(1节点) | 278 | |
| arm | 32(1节点) | 352 | |
| x86 | 32(1节点) | 263 | 8077(241) |
| x86 | 64(1节点) | 240 | 14753(342) |
Ended: 应用软件
Linux ↵
参考:
基本概念¶
操作系统¶
计算机是一台机器,它按照用户的要求接收信息、存储数据、处理数据,然后再将处理结果输出(文字、图片、音频、视频等)。计算机由硬件和软件组成:
- 硬件是计算机赖以工作的实体,包括显示器、键盘、鼠标、硬盘、CPU、主板等;
- 软件会按照用户的要求协调整台计算机的工作,比如 Windows、Linux、Mac OS、Android 等操作系统,以及 Office、QQ、微信等应用程序。
操作系统(Operating System,OS)是软件的一部分,它是硬件基础上的第一层软件,是硬件和其它软件沟通的桥梁。
操作系统会控制其他程序运行,管理系统资源,提供最基本的计算功能,如管理及配置内存、决定系统资源供需的优先次序等,同时还提供一些基本的服务程序。
Linux历史¶
- 1970年,Unix 诞生于贝尔实验室,开放源码,此后诞生了AIX、Solaris、HP-UX、BSD等Unix系统
- 1979年,从Unix V7开始,贝尔实验室严格了版本 ,禁止其源码用于教学
- 1987年,Tanenbaum 发布了其从头开发、兼容Unix V7的操作系统 Miniux(mini-UNIX)用于教学
- 1991年,Minix功能有限,Linus借鉴Minix开发了Linux(内核)
- 1983年,Stallman发起GNU(GNU is not Unix)计划,目标是创建一套完全自由的操作系统,并陆续开发了Emacs、GCC等各种工具,但缺乏操作系统内核
- 1991年Linus开发出Linux内核之后,GNU软件和Linux内核结合, 形成现代Linux生态环境。Stallman发布的GPL协议保障了开源软件的发展。
根据不同目的和用途,整合Linux内核和各类应用软件形成不同的操作系统,称之为Linux发行版。常见的Linux发行版有Ubuntu、Dedian、RHEL、Fedora、CentOS、SLE等。
Linux特点¶
- 免费
- 一切皆文件
- 由目的单一的小程序组成,组合小程序完成复杂任务
- 多任务、多用户系统
- 大量的可用软件
- 良好的可移植性及灵活性
- 优秀的稳定性和安全性
Linux组成¶
- Kernel(内核)
- 系统启动时将内核装入内存
- 管理系统各种资源
- Shell
- 保护操作系统
- 用户界面,提供用户与内核交互处理接口,各种命令运行的地方
- bash, ash, pdksh, tcsh, ksh, sh, csh, zsh….
- Utility
- 提供各种管理工具,应用程序
文件¶
linux哲学核心思想是"一切皆文件"。"一切皆文件"指的是,对所有文件(目录、字符设备、块设备、套接字、打印机、进程、线程、管道等)操作,读写都可用 fopen()/fclose()/fwrite()/fread() 等函数进行处理。屏蔽了硬件的区别,所有设备都抽象成文件,提供统一的接口给用户。虽然类型各不相同,但是对其提供的却是同一套操作界面。
文件具有多种属性,如类型、所属用户、用户组、权限、修改时间、大小等。
所有文件的起始为 / (根目录),采用树形结构存放。各类文件存放目录遵循FHS (Filesystem Hierarchy Standard)。
文件的位置称之为路径,如 /var/log/messages。
- 从
/写起的为绝对路径,如/var/log/messages - 从当前位置写起的为
相对路径,如log/messages
https://www.runoob.com/wp-content/uploads/2014/06/d0c50-linux2bfile2bsystem2bhierarchy.jpg
/
├── bin # binaries(二进制文件),存放着最常用的程序和指令
├── boot # 系统启动相关文件,如内核,initrd,以及grub(BootLoader)
├── dev # device,设备文件 — 体现了LInux的“一切皆文件”思想
├── etc # 配置文件。大多数为纯文本文件
├── home # 用户的家目录
├── lib # library,公共库文件(不能单独执行, 只能被调用)
├── media # 挂载点目录,挂载移动设备(如U盘)
├── mnt # 挂载额外的临时文件(如第二块硬盘)
├── opt # optional,存放额外安装软件
├── proc # processes,伪文件系统,内存映射文件,系统启动后才出现文件, 关机就空
├── root # 管理员的家目录
├── run # 是一个临时文件系统,存储系统启动以来的信息
├── sbin # superuser binaries,存放系统管理所需要的命令
├── srv # 该目录存放一些服务启动之后需要提取的数据
├── tmp # temporary,临时文件,所有用户都可以操作,但只能删自己的,不能删别人的
├── usr # unix shared resources,存放只能读的命令和其他文件
└── var # variable,存放运行时需要改变数据的文件,如日志等
用户和用户组¶
多个用户可以在同一时间内登录同一个Linux系统执行各自不同的任务,互不影响。每个用户有唯一的用户名、用户id(uid),并通过各自的密码登录使用,每个用户不能干涉其它用户的活动、访问修改其它用户的文件。
root用户(超级用户)拥有系统最高权限。
用户组是具有相同特征用户的逻辑集合。有时需要让多个用户具有相同的权限,比如查看、修改某一个文件的权限等,通过用户组可以方便地实现。将用户分组是Linux 系统中对用户进行管理及控制访问权限的一种手段,通过定义用户组,简化了管理工作。每个用户组有唯一的名称和id(gid)。
用户和用户组的对应关系有:
- 一对一:即一个用户可以存在一个组中,也可以是组中的唯一成员
- 一对多:即一个用户可以存在多个用户组中。那么此用户具有多个组的共同权限
- 每个用户拥有唯一的主用户组(primary group)
- 每个用户可以拥有多个附加用户组(supplementary groups)
- 多对一:多个用户可以存在一个组中,这些用户具有和组相同的权限
- 多对多:多个用户可以存在多个组中。其实就是上面三个对应关系的扩展
权限¶
在Linux中,多用户之间通过权限实现资源的隔离和共享,以保障整个系统的安全。权限的主体为用户,对象为文件。用户对自己的文件有绝对的权限,本用户的文件默认禁止其它用户访问,可通过更改文件权限向其它用户共享特定文件,使其它用户可以访问和修改该文件。
root用户对所有文件拥有访问和修改的权限。
命令¶
命令(command)是在命令行上运行的程序或实用程序。命令行(CLI, command line interface),是一种命令提示符界面,它接受文本行并将其处理为计算机的指令。
命令提示符(prompt):登陆成功后显示的东西,如[username@localhost ~]$ ,其中 #:root用户,$:普通用户。
命令格式: 命令 -选项 参数
- 命令:shell传递给内核,并由内核判断该程序是否有执行权限,以及是否能执行,从什么时候开始执行
- 选项(options):命令所含的一些选项,通过选项控制命令的细节行为(可选,非必须)
- 短选项:
-character,多个选项可以组合 ,可以写ls -l -a或者ls -la - 长选项:
–word,不能组合,要分开
- 短选项:
- 参数(arguments),多数用于指向命令的指向目标(可选,非必须),多个参数要用空格隔开,参数顺序决定命令的作用顺序
命令类型:
- 内置命令(shell builtin 内置)
- 外部命令:某个路径下有一个与命令名称相应的可执行文件
命令补全:使用 tab键 补全功能,可提升输入效率,减少输入错误
- 命令补全:tab 接在一串指令的第一个字的后面
- 文件补全:tab 接在一串指令的第二个字以后时
命令历史:shell会记录执行过的命令,退出后写入~/.bash_history。使用history 命令查看使用过的命令
↑和↓可以上下翻看使用过的命令ctrl + r输入字符,可以快速定位到使用过的命令
命令帮助:man 命令 打开命令手册信息,包含命令的详细用法及各选项解释;命令 -h 显示命令用法
命令存放在$PATH的路径中,命令执行时会在这些路径中逐一搜索,并执行第一个搜索的命令。可以使用绝对路径准确执行命令。
$ echo $PATH
/usr/local/bin:/bin:/usr/bin:/usr/local/sbin:/usr/sbin
命令行常用快捷键:
ctrl+a:跳到命令行首
ctrl+e:跳到命令行尾
ctrl+u:删除光标至命令行首的内容, 命令行下清除输错的密码很管用
ctrl+k:删除光标至命令行尾的内容
ctrl+l:清屏
文件操作¶
文件属性¶
$ ls -lh
total 3.0K
drwxr-xr-x 2 username usergroup 4.0K Sep 17 2020 code
drwxr-xr-x 5 username usergroup 4.0K Sep 23 2020 data
drwxr-xr-x 2 username usergroup 4.0K Sep 6 2019 data2
-rw-r--r-- 1 username usergroup 50 May 18 09:09 file
drwxr-xr-x 2 username usergroup 4.0K Sep 6 2016 MPI
drwxr-xr-x 14 username usergroup 4.0K Jun 3 2019 software
- 第一字段:文件类型;
- 第二字段:文件访问权限;
- 第三字段:硬链接个数;
- 第四字段:属主(owner),拥有该文件或目录的用户账号;
- 第五字段:所归属的组(group),拥有该文件或目录的组账号;
- 第六字段:文件或目录的大小, 默认单位 bytes;
- 第七字段:最后访问或修改时间;
- 第八字段:文件名或目录名
文件类型¶
linux中文件后缀不能表示文件类型,只是方便识别
| 类型 | 简称 | 描述 |
|---|---|---|
| 普通文件 | -,Normal File | 如mp4、pdf、html log;用户可以根据访问权限对普通文件进行查看、更改和删除,包括 纯文本文件(ASCII);二进制文件(binary);数据格式的文件(data);各种压缩文件.第一个属性为 - |
| 目录文件 | d,directory file | /usr/ /home/,目录文件包含了各自目录下的文件名和指向这些文件的指针,打开目录事实上就是打开目录文件,只要有访问权限,就可以随意访问这些目录下的文件。能用 cd 命令进入的。第一个属性为 d,例如 drwxrwxrwx |
| 硬链接 | -,hard links | 若一个inode号对应多个文件名,则称这些文件为硬链接。硬链接就是同一个文件使用了多个别名删除时,只会删除链接, 不会删除文件;硬链接的局限性:1.不能引用自身文件系统以外的文件,即不能引用其他分区的文件;2.无法引用目录; |
| 符号链接(软链接) | l,symbolic link | 若文件用户数据块中存放的内容是另一文件的路径名的指向,则该文件就是软连接,克服硬链接的局限性, 类似于快捷方式,使用与硬链接相同 |
| 字符设备文件 | c,char | 文件一般隐藏在/dev目录下,在进行设备读取和外设交互时会被使用到,即串行端口的接口设备,例如键盘、鼠标等等。第一个属性为 [c]。/dev/tty 的属性是 crw-rw-rw-,前面第一个字 c,这表示字符设备文件 |
| 块设备文件 | b,block | 存储数据以供系统存取的接口设备,简单而言就是硬盘。/dev/hda1 的属性是 brw-r—– ,注意前面的第一个字符是 b,这表示块设备,比如硬盘,光驱等设备。系统中的所有设备要么是块设备文件,要么是字符设备文件,无一例外 |
| FIFO管道文件 | p,pipe | 管道文件主要用于进程间通讯。FIFO解决多个程序同时存取一个文件所造成的错误。比如使用 mkfifo 命令可以创建一个FIFO文件,启用一个进程A从FIFO文件里读数据,启动进程B往FIFO里写数据,先进先出,随写随读。 |
| 套接字 | s,socket | 以启动一个程序来监听客户端的要求,客户端就可以通过套接字来进行数据通信。用于进程间的网络通信,也可以用于本机之间的非网络通信,第一个属性为 [s] ,这些文件一般隐藏在 /var/run 目录下,证明着相关进程的存在 |
日常使用中接触最多的为普通文件、目录文件、软连接,其中对于普通文件而言,根据其可读性可分为文本文件(ASCII text)、其它文件等。
文本文件类似于windows下的txt文件,可以使用文本操作命令 cat less 等直接读取,如各种序列文件、C源代码、程序脚本等;
其它文件有各类命令、压缩文件等,一般不可直接读取,直接 cat 读取会显示为乱码。
一般使用 file 命令可以查看普通文件的类型:
# 显示ASCII text为文本文件
$ file IRGSP-1.0_genome.fasta
IRGSP-1.0_genome.fasta: ASCII text
# C代码源文件也是ASCII text,即文本文件
$ file hello.c
hello.c: C source, ASCII text
# shell脚本也是ASCII text,即文本文件
$ file hello.sh
hello.sh: POSIX shell script, ASCII text executable
# python脚本也是ASCII text,即文本文件
$ file test.py
test.py: a /bin/python script, ASCII text executable
# 这种为ELF文件,即Linux中的可执行文件,一般为C/C++编译后生成
$ file /bin/ls
/bin/ls: ELF 64-bit LSB executable, x86-64, version 1 (SYSV), dynamically linked (uses shared libs), for GNU/Linux 2.6.32, BuildID[sha1]=c5ad78cfc1de12b9bb6829207cececb990b3e987, stripped
# gzip压缩文件
$ file HW84_NDSW31815_1.clean.fq.gz
HW84_NDSW31815_1.clean.fq.gz: gzip compressed data, was "NDSW31815_NDSW31815_1.clean.fq", from Unix, last modified: Fri Jul 6 16:17:47 2018
# 可以看到bam文件也是一种gzip压缩文件
$ file HW84_NDSW31815_sorted.bam
HW84_NDSW31815_sorted.bam: gzip compressed data, extra field
访问权限¶
文件属性的权限字段中,有9个字符,3个字符一组,共三组,分别代表属主(u, user)、属组(g, group)、其它人(o, other)的权限;
每组权限字符中,从第一位到第三位分别为下面4个字符中的一个:
图片来源 booleanworld.com
文件和目录的权限作用不同;
- 读取(r, read): 允许查看文件内容;允许
ls显示目录列表。数字表示为4, - 写入(w, write): 允许修改文件内容;允许在目录中新建、删除、移动文件或者子目录。数字表示为2
- 可执行(x, execute): 允许运行程序;允许
cd进入目录。数字表示为1 - 无权限(-): 没有权限,数字表示为0
| 类型 | r | w | x |
|---|---|---|---|
| 文件 | 读取文件内容 | 修改文件内容 | 执行文件内容 |
| 目录 | 读取文件列表 | 修改文件或目录 | 进入该目录的权限 |
为方便表示,每组权限字符的3个数字表示可以相加
图片来源 booleanworld.com
如 rwxr-xr-x 可以表示为 4+2+1=7,4+1=5, 4+1=5,即755
文件扩展名¶
linux 文件扩展名无特殊意义,一般用于标识文件内容,如:
- .sh:shell脚本
- Z、.tar、.tar.gz、.zip、.tgz:经过打包的压缩文件。不同的压缩软件压缩的扩展名不同如,gunzip、tar
- .html、.php:网页相关文件
- .pl、.py: Perl、Python 脚本
常用命令¶
- ls 列出文件,-a -h -l -t -r –d
- tree 以树形列出文件
- cd 切换目录 cd; cd ~; cd -; cd ..; cd ../..
- pwd 显示当前目录
- mkdir 创建目录, -p
- rm 删除文件 –r
- mv 移动文件或者重命名文件
- cp 复制文件 –r
- touch 创建空文件或更新文件时间
- rename 文件重命名, 支持正则 rename fasta fa *.fasta
- find 查找文件, -name -type –size
- which 查找可执行文件的绝对路径
- ln 创建链接 –S 删除软链接时要特别小心
- du 显示文件和目录的磁盘使用情况 -sh
- chmod 更改文件权限 755 -R
- tar 文件目录压缩、解压 –xf file.tgz; -zcvf file.tgz file; -tvf file.tgz
- gzip 文件压缩、解压 –d, 推荐 pigz, 支持多线程
# 列出 ~/training 目录内的文件
$ ls ~/training
code data data2 file MPI software
# -a, 列出 ~/training 目录内的所有文件,包含隐藏文件
$ ls -a ~/training
. .. code .config data data2 file MPI software
# -l, 以长格式显示目录下的内容列表, 日常使用可以使用ll代替ls -l。
# 输出的信息从左到右依次包括文件名,文件类型、权限模式、硬连接数、所有者、组、文件大小和文件的最后修改时间等
$ ls -l ~/training
total 3
drwxr-xr-x 2 username usergroup 4096 Sep 17 2020 code
drwxr-xr-x 5 username usergroup 4096 Sep 23 2020 data
drwxr-xr-x 2 username usergroup 4096 Sep 6 2019 data2
-rw-r--r-- 1 username usergroup 50 May 18 09:09 file
drwxr-xr-x 2 username usergroup 4096 Sep 6 2016 MPI
drwxr-xr-x 14 username usergroup 4096 Jun 3 2019 software
# -h, 打印大小,K表示千字节,M表示兆字节,G表示吉字节
$ ll -h
total 3.0K
drwxr-xr-x 2 username usergroup 4.0K Sep 17 2020 code
drwxr-xr-x 5 username usergroup 4.0K Sep 23 2020 data
drwxr-xr-x 2 username usergroup 4.0K Sep 6 2019 data2
-rw-r--r-- 1 username usergroup 50 May 18 09:09 file
drwxr-xr-x 2 username usergroup 4.0K Sep 6 2016 MPI
drwxr-xr-x 14 username usergroup 4.0K Jun 3 2019 software
#-t, 用文件和目录的更改时间排序
ls -lt
total 3
-rw-r--r-- 1 username usergroup 50 May 18 09:09 file
drwxr-xr-x 5 username usergroup 4096 Sep 23 2020 data
drwxr-xr-x 2 username usergroup 4096 Sep 17 2020 code
drwxr-xr-x 2 username usergroup 4096 Sep 6 2019 data2
drwxr-xr-x 14 username usergroup 4096 Jun 3 2019 software
drwxr-xr-x 2 username usergroup 4096 Sep 6 2016 MPI
# -r, 输出目录内容列表
$ ls -lr
total 3
drwxr-xr-x 14 username usergroup 4096 Jun 3 2019 software
drwxr-xr-x 2 username usergroup 4096 Sep 6 2016 MPI
-rw-r--r-- 1 username usergroup 50 May 18 09:09 file
drwxr-xr-x 2 username usergroup 4096 Sep 6 2019 data2
drwxr-xr-x 5 username usergroup 4096 Sep 23 2020 data
drwxr-xr-x 2 username usergroup 4096 Sep 17 2020 code
# –d, 仅显示目录名,而不显示目录下的内容列表。显示符号链接文件本身,而不显示其所指向的目录列表
# 注意-d和不加-d参数的区别
$ ls -ld software
drwxr-xr-x 14 username usergroup 4096 Jun 3 2019 software
$ ls -l software
total 221717
drwxr-xr-x 5 username usergroup 4096 Sep 6 2016 blast-2.2.31
drwxr-xr-x 5 username usergroup 4096 Sep 6 2016 bowtie2-2.1.0
-rw-r----- 1 username usergroup 15631689 Sep 6 2016 bowtie2-2.1.0-linux-x86_64.zip
drwxrwxr-x 5 username usergroup 4096 Sep 9 2016 bowtie2-2.2.6
drwxr-xr-x 5 username usergroup 8192 Sep 6 2016 hisat2-2.0.4
-rw-r--r-- 1 username usergroup 3875287 Sep 6 2016 hisat2-2.0.4-source.zip
# -f, 不进行排序输出。ls默认会将所有文件名进行排序之后再输出。
# 当目录内文件数据量非常多时,如几千到几万个文件,ls会非常耗时,建议加上-f参数节省时间
$ls -f
. .. .config code MPI software file data data2
# 列出 data2目录下的文件
$ tree data2/
data2/
├── blast.bak.pbs
├── blast.e1692608
├── blast.lsf
├── blast.o1692608
├── blast.pbs
├── example.lsf
├── MH63_cds.fa
├── MH63_cds.nhr
├── MH63_cds.nin
├── MH63_cds.nog
├── MH63_cds.nsd
├── MH63_cds.nsi
├── MH63_cds.nsq
├── prosss.sh
├── ZS97_cds
└── ZS97_cds.fa
0 directories, 16 files
# 从当前目录切换到 '~/app' 目录
$ cd ~/app
# 从任何位置切换回用户主目录 '/home/User'
$ cd
# 回退上一个访问目录
$ cd -
# 返回上一级目录
$ cd ..
# 返回上上一级目录
$ cd ../../
# 使用相对路径,进入另一个目录"new directory",这里 '\' 是转义符,将空格键正确转义
$ cd ../../../new\ directory
# 当不知道当前处于什么路径时,可以用这个命令显示
$ pwd
/public/home/username/training
# 在当前目录下创建新目录 new
$ mkdir new
# -p, 在当前目录下创建多级目录
$ mkdir -p newdir1/newdir2/newdir3/newdir4
$ tree newdir1/
newdir1/
└── newdir2
└── newdir3
└── newdir4
3 directories, 0 files
# 删除普通文件
$ rm file
# 删除当前目录的所有后缀是 '.sra' 的文件
$ rm *.sra
# -r, 参数删除目录
# 删除目录 'new' 以及 'new' 下的所有子文件与子目录
$ rm -r new
# -f, 不弹出删除确认提示,删除所有 '.tmp' 文件
$ rm -f *.tmp
# 将 'file' 改名为 'file1'
$ mv file file1
# 将 'file1' 移动到 'newdir'目录内
$ mv file1 newdir/
# 复制 'file1' 文件到目录 '~/newdir' 中
$ cp file1 ~/new
# -r 复制目录
# 复制 newdir 目录到用户目录下
$ cp -r newdir/ ~
# 创建空文件 'empty.txt'
$ touch emplty.txt
# 方便批量修改文件名
# 将 'seq.fasta' 更名为 'seq.fa'
$ rename seq.fasta seq.fa seq.fasta
# 将所有fasta文件更名为fa
$ rename fasta fa *fasta
# 去掉所有fa文件前缀
$ rename Osativa_323_v7.0.id_ '' *fa
# 查找一个文件,比如 /etc路径下的hosts文件
$ find /etc/ -name hosts
# 查找当前目录及子目录后缀为gz的文件
$ find . -type f -name "*.gz"
# 查找所有的 html 网页文件
$ find . -type f -regex ".*html$"
# 将所有 fasta 文件中序列名字包含node的改成seq
$ find . -type f -regex ".*fasta$" | xargs sed 's/NODE/Seq/g'
# 查找子目录深度为4层目录的fasta文件
$ find . -type f -maxdepth 4 -name "*.fasta"
# 查找/var/log路径下权限为755的目录
$ find /var/log -type d -perm 755
# 按照用户查找用户,在tmp路径下查找属于用户 nginx 的文件
$ find /tmp -user nginx
# 用户主home路径下查找属于 root 权限,属性为644的文件
$ find . -user root -perm 644
# 打印出系统带的 python 程序的路径
$ which python
/bin/python
# 为 '~/ref/ref.fa' 在当前目录下创建软连接
$ ln -s ~/ref/ref.fa ref.fa
# 如果需要软连接与源文件文件名相同,可以如下方式简写
$ ln -s ~/ref/ref.fa .
# 删除软连接时,文件名加了/表示将源文件一并删除,不加/表示只删除当前的软连接
# 删除软连接
$ rm ref.fa
# 删除源文件
$ rm ref.fa/
# 查看data目录的磁盘使用
$ du -sh data
14G data
# home目录下运行下面的命令,查看各目录所占磁盘空间大小
$ du -h --max-depth=1
512 ./Desktop
8.1M ./githup
132K ./.ssh
45M ./perl5
512 ./Videos
u User,即文件或目录的拥有者;
g Group,即文件或目录的所属群组;
o Other,除了文件或目录拥有者或所属群组之外,其他用户皆属于这个范围;
a All,即全部的用户,包含拥有者,所属群组以及其他用户;
r 读取权限,数字代号为“4”;
w 写入权限,数字代号为“2”;
x 执行或切换权限,数字代号为“1”;
- 不具任何权限,数字代号为“0”;
s 特殊功能说明:变更文件或目录的权限;
chmod u/g/o/a +/-/= r/w/x 文件或目录
# 给文件的User加上可执行权限
$ chmod u+x file
# 将目录共享给组用户
$ chmod g+rwx dir
# 取消文件的x权限
$chmod -x file
# 取消 home 目录的共享权限
$ chmod 700 ~
# 查看 home 目录权限
$ ll -d ~
drwx------ 30 username usergroup 8192 Sep 14 10:24 /public/home/username
# 打包压缩
$ tar -czvf data.tar.gz data
# 使用pigz多线程打包压缩,加快打包压缩速度;解压同理
$ tar -cvf data.tar.gz data -I pigz
# 解压
$ tar -xf data.tar.gz
# 使用 pigz 解压
$ tar -I pigz -xf data.tar.gz
# 查看但不解压
$ tar -ztvf
# 压缩文件
$ gzip reads.fq
# -d, 解压文件
$ gzip -d reads.fq.gz
#使用方式与gzip基本一致,-p参数可以使用多线程,加速文件压缩/解压
# 压缩文件
$ pigz -p 4 reads.fq
# 解压文件
$ pigz -p 4 -d reads.fq.gz
# 多线程打包压缩
$ tar cvf - dir | pigz -p 4 >dir.tgz
# 或
$ tar -cvf data.tar.gz data -I pigz
# 使用 pigz 解压 tar.gz 压缩包
$ tar -I pigz -xf data.tar.gz
- wget 下载文件
- scp 远程文件传输
- rsync 文件传输
参考:https://wangchujiang.com/linux-command/c/wget.html
# 下载samtools
$ wget https://github.com/samtools/samtools/releases/download/1.3.1/samtools-1.3.1.tar.bz2
# -c,断点续传
$ wget -c https://github.com/samtools/samtools/releases/download/1.3.1/samtools-1.3.1.tar.bz2
# -b,后台下载
$ wget -b https://github.com/samtools/samtools/releases/download/1.3.1/samtools-1.3.1.tar.bz2
# -o,日志文件
$ wget https://github.com/samtools/samtools/releases/download/1.3.1/samtools-1.3.1.tar.bz2 -o log
# ftp下载,需要用户名和密码
$ wget --ftp-user=USERNAME --ftp-password=PASSWORD url
# ftp 匿名下载,下载目录内所有文件
$ wget -c -r ftp://download.big.ac.cn/gsa/CRA004538
# -i,下载多个文件
$ wget -i filelist.txt
$ cat filelist.txt
url1
url2
url3
url4
# 从本地传到远端
$ scp file user@ip:/dir/
# 从远端传到本地
$ scp user@ip:/dir/file .
# -r,传输目录
$ scp -r dir user@ip:/dir/
# -P,更改默认端口
$ scp -P 12345 file user@ip:/dir/
# -p,保留原文件的修改时间,访问时间和访问权限
$ scp -p file user@ip:/dir/
# 由于rsync在传输时会进行校验,因此当网络不稳定或文件数较多时,
# 可以使用rsync检验文件是否传完,以及跳过已经传输过的文件
# 同步本地目录dir1到本地目录dir2
$ rsync -avP dir1 dir2
# 同步本地目录dir1内的文件到本地目录dir2。注意dir1有无 "/" ,传输的文件有区别
$ rsync -avP dir1/ dir2
# 同步本地目录dir1 到远程目录dir2
$ rsync -avP dir1 user@ip:/dir2/
# 更改默认端口
$ rsync -avP -e 'ssh -P 12345' dir1 user@ip:/dir2/
路径特殊字符¶
| 字 符 | 含 义 |
|---|---|
| ~ | home 目录 |
| - | 代表上一次(相对于当前路径)用户所在的路径 |
| . | 当前目录 |
| .. | 上层目录 |
通配符¶
一次性操作多个文件时,命令行提供通配符(wildcards),用一种很短的文本模式(通常只有一个字符),简洁地代表一组路径。
通配符早于正则表达式出现,可以看作是原始的正则表达式。它的功能没有正则那么强大灵活,但是胜在简单和方便。
| 字 符 | 含 义 |
|---|---|
| * | 匹配0个或多个字符 |
| ** | 匹配任意级别目录(bash 4.0以上版本支持) |
| ? | 匹配任意一个字符 |
| [list] | 匹配list中的任意单一字符, 例如[abcd] 则表示一定由一个字符,可能是 a、b、c、d 中的任意一个 |
| [!list] | 匹配除list中的任意单一字符以外的字符 |
| [c1-c2] | 匹配c1-c2中的任意单一字符,如[0-9][a-z] |
| {string1, string2, ...} | 匹配string1或string2(或更多其一字符串) |
| {c1..c2} | 匹配c1-c2中的全部字符串 |
*.txt # 匹配全部后缀为.txt的文件
file?.log # 匹配file1.log, file2.log, ...
[ab].txt # 匹配 a.txt, b.txt
[a-z]*.log # 匹配a-z开头的.log文件
[^a-z]*.log # 上面的反向匹配
{a,b,c}.txt # 匹配a.txt, b.txt, c.txt
{j{p,pe}g,png} # 匹配 jpg jpeg png
~/**/*.conf # 匹配home目录下所有.conf文件
命令练习¶
Q1. 在home目录下创建自己名字命名的工作目录(如,zhangsan),进入该目录,然后再创建目录work1和work2,同时创建空文件test1.txt test2.txt。
mkdir zhangsan
cd zhangsan
mkdir work1
mkdir work2
touch test1.txt
touch test2.txt
ls -lh
Q2. 使用gzip压缩test1.txt文件,然后将压缩后的文件复制到work1目录下,最后在工作目录内目录内创建被压缩文件的软连接。
gzip test1.txt
ls -lh
cp test1.txt.gz work1
ln -s test1.txt.gz zhangsan.gz
Q3. 将当前目录下的test2.txt文件重命名,重命名后的文件为姓名首字母,如zs.txt。
mv test2.txt zs.txt
Q4. 将重命名后的文件zs.txt 移动到work2目录下,并将该文件权限改成700。
pwd
mv zs.txt work2/
chmod 700 work2/zs.txt
ls -lh work2/
Q5. 创建多层目录 1/2/3。
cd ~/zhangsan/
mkdir -p 1/2/3
Q6. 查找个人目录下所有以 gz 结尾的文件
cd ~/zhangsan/
find . -name "*gz"
Q7. 将 work1 目录 打包压缩成 work1.tar.gz,然后删除 work1 目录
tar -czvf work1.tar.gz work1
rm -rf work1
Q8. 将压缩包 work1.tar.gz 解压至 work2 中
pwd
tar -xzf work1.tar.gz -C work2
Q9. 统计工作目录下所有文件和目录的大小
cd ~/zhangsan/
du -sh *
Q10. 使用 tree 命令查看自己工作目录下的所有文件。
cd ~/zhangsan/
tree
文本操作¶
常用命令¶
- cat 打印输出文件内容, -A -n ;tac 反序输出
- less 分屏显示文件内容, -S –N
- more 类似less
- head 显示文件开头内容, -n 10; -n -10
- tail 显示文件尾部内容, -f -n 10; -n +10
- wc 文件内容计数 –l
- paste 多个文件按列合并
- sort 文件排序并输出 -k -n -r –t, 新版本的sort支持多线程排序
- uniq 显示重复行, 结合sort使用, -c –u
- diff 比较两个文件内容的异同
- tr 对输入字符进行替换 tr "ATCG" "TAGC"; tr 'A-Z' 'a-z'
- rev 逆序输出文件字符
- split 对文件进行拆分 -b –l
- cut 从文件每行提取字符 -d -f
- grep 文件内容搜索 -v -i -B -A -E -c -H
- zcat、zless、zmore、zgrep 对gzip压缩的文本做相应操作
- dos2unix windows格式文本换行成unix格式文本
- fold 把超过指定宽度的长行切成多行,默认宽度 80,常用于快速查看无换行的长序列/日志而不触发横向滚动
# 输出 'file' 文件的内容。一次输出文件全部内容,大文件请勿直接使用cat输出
$ cat file
a b 1
c d 2
f g 4
s g 3
# -n, 带行号
$ cat -n file
1 a b 1
2 c d 2
3 f g 4
4 s g 3
# -A, 打印特殊字符,比如换行符,制表键
$ cat -A file
a b 1$
c d 2$
f g 4$
s^Ig^I3$
# tac, 逆序输出文件内容,即先打印文件最后一行
$ tac file
s g 3
f g 4
c d 2
a b 1
# 分屏输出文件内容,默认只输出一屏内容
# 敲回车键,一行行显示后面的内容;
# f键,翻页显示
# 空格键:向下翻一页
# pagedown:向下翻一页
# pageup:向上翻一页
# /字符串:向下搜索字符串;注意这个斜杠也是需要输入的,不是在 「:」输入,:也和这个是一个功能
# ?字符串:向上搜索字符串
# n:重复前一个搜索(与 / 或 ?有关)
# N:反向的重复前一个搜索
# g:前进到这个资料的第一行
# G:前进到这个资料的最后一行去(注意是大写)
# q:离开 less 这个程序
$ less ref.fa
# -S, 不自动换行。less默认输出内容超过屏幕宽度会自动换行
$ less -S ref.fa
# -N, 添加行号
$ less -N ref.fa
# 输出文件前5行内容
$ head -n 5 num
1
2
3
4
5
# 输出除最后5行之外的内容
$ head -n -5 num
6
7
8
9
10
# 输出文件最后5行内容
$ tail -n 5 num
6
7
8
9
10
# 输出文件前5行之外的内容
$ tail -n +5 num
6
7
8
9
10
# 文件末尾有更新时,刷新更新内容
$ tail -f result
# 统计文件行数
$ wc -l num
10 num
$ cat file1
1
2
3
4
5
$ cat file2
a
b
c
d
e
# 按列合并两个文件,输出内容默认使用\t作为列之间的分隔符
$ paste file1 file2
1 a
2 b
3 c
4 d
5 e
$ cat file3
1
11
101
40
22
55
33
# 默认按照ASII码排序,对数字的排序与我们直观映像不同
$ sort file3
1
101
11
22
33
40
55
# -n, 按数字大小排序
$ sort -n file3
1
11
22
33
40
55
101
# -h, 按人可读的方式排序
$ sort -h file4
100
1K
1M
1G
# -k, 按指定的列排序
# 以第3列的数字排序
$ sort -nk3 file
w u 0
a b 1
c d 2
s g 3
# --parallel, 多线程
$ sort --parallel=4 file
# -T, 指定临时目录,默认为/tmp目录。对超大文件排序时,需要指定到本地,避免/tmp写满
$ sort -T ./mytem file
# -S, 指定内存。对超大文件需要限制内存使用,避免耗尽节点内存
$ sort -S 5G file
# -r, 逆序输出
$ sort -r
# 对genome.gtf文件先按染色体排序,然后再按位置排序
# sort -k1,4n genome.gtf 达不到所需要的效果,正确使用如下
$ sort -k1,1 -k4,4n genome.gtf
# 一般与sort 配合使用
# 显示重复行,并显示每个重复行的重复数
$ sort ref.fa |uniq -c
1 >chr10
99 NNNNNNNNNNNNNNNNNNNNNNNNNNNNNNNNNNNNNNNNNNNNNNNNNN
# 显示 file1和file2 2个文件内容的不同之处
$ diff file1 file2
# 碱基替换
$ echo "atctcctt"|tr "atcg" "tagc"
tagaggaa
# 大小写转换
$ echo "hello world"|tr 'a-z' 'A-Z'
HELLO WORLD
# 逆序输出
$ echo "hello world"|rev
dlrow olleh
# 碱基互补配对
$ echo "atctcctt"|tr "atcg" "tagc"|rev
aaggagat
# 将 'file' 文件拆分成10kb的小文件
$ split -b 10k file
# 将 'file' 文件拆分成每个只有10行的小文件
$ split -l 10 file
# -f 指定输出的列
# 输出文件第三列
$ cut -f2 genome.gff3|head -n5
gene
mRNA
mRNA
mRNA
mRNA
# -d 指定分隔符,默认为\t
# 输出csv文件的第三列
$ cut -d',' -f3 file.csv|head -n5
# 查找 'ref.fa' 文件中包含 "ACCC" 的行
$ grep "ACCC" ref.fa
# -v, 输出不匹配的行
# 查找 'ref.fa' 文件中不包含 "ACCC" 的行
$ grep -v "ACCC" ref.fa
# -i, 忽略大小写
# 查找 'ref.fa' 文件中包含 "ACCC" 的行,匹配时忽略大小写
$ grep -i "ACCC" ref.fa
# -c, 统计行数
# 查找 'ref.fa' 文件中包含 "ACCC" 的行的行数
$ grep -c "ACCC" ref.fa
# -e, 多模式匹配
# 查找 'ref.fa' 文件中包含 "ACCC" 或 "AGGG" 的行的行数
$ grep -e "ACCC" -e "AGGG" ref.fa
# -w, 匹配整词
# 匹配 word 而不匹配 words
$ grep -w 'word' file
# 查找 'ref.fa' 文件中包含 "ACCC" 的行,以及这行的前2行和后3行
$ grep -A2 -B3 "ACCC" ref.fa
# 打印文件 'file' 第3列
$ awk '{print $3}' file
# 提取文件 'file' 第1和3列,并用\t分隔
$ awk '{print $1"\t"$3}' file
$ awk 'print $1"\t"$3' file
# 打印文件 'file' 的倒数第二列
$ awk '{ print $(NF-1)}' file
# 将 'file' 文件中第三列相加求和
$ awk 'BEGIN{a=0}{a+=$3}END{print "Sum = ", a}' file
# 求第三列的平均值
$ awk 'BEGIN{a=0}{a+=$3}END{print "Average = ", a/NR}' file
# 求第三列的最大值
$ awk 'BEGIN {max = 0} {if ($3>max) max=$3 fi} END {print "Max=", max}' file
# 求第三列最小值
awk 'BEGIN {min = 1999999} {if ($1<min) min=$1 fi} END {print "Min=", min}' file
# 将 'genome.gff3' 文件中代表基因的行打印出来
$ awk '{if($3 == "gene" ){print $0}}' genome.gff3
$ awk '{if($3 == "gene" )print $0}' genome.gff3
# 将 'genome.gff3' 文件中代表基因、位于正链、且序列长度小于1000bp的行打印出来
$ awk '{if($3 == "gene" && $7 == "+" && ($5 -$4)<1000 ){print $0}}' genome.gff3
# 将 'file' 中的 gene 字符全部替换成 GENE
$ sed 's/gene/GENE/g' file
# 给 'file' 中每个单词加个中括号[]
$ sed 's/\w\+/[&]/g' file
# 删除 'file' 第2行
$ sed '2d' file
# 删除 'file' 中的空白行
$ sed '/^$/d' file
# 删除 'file' 中所有开头是test的行
$ sed '/^test/'d file
# 从1行开始,每隔3行插入>,并以行号为序列ID
$ sed '1~2 s/^/>\n/' example.fa|awk '{if($1 ~ />/){print $1NR}else{print $1}}' > example2.fa
# 格式化fa序列文件
fold -w 60 seq.fa > seq_60.fa
# 由于windows和linux中文本的换行符不同,windows下创建的文本在linux下直接使用可能会出错,需要进行格式转换
# windows格式文本换行成unix格式文本
$ dos2unix file
# 打印输出压缩文件
$ zcat file.gz
# 查看压缩文件
$ zless file.gz
# 搜索压缩文件中含字符串 aaaccc 的行
$ zgrep aaaccc file.gz
正则表达式¶
正则表达式(regular expression)是一些具体有特殊含义的符号,组合在一起的共同描述字符或字符串的方法。在 Linux 系统中,正则表达式通常用来对字符或字符串来进行处理,它是用于描述字符排列或匹配模式的一种语言规则。在grep/awk/sed中,经常需要使用正则表达式来处理文本。
https://man.linuxde.net/docs/shell_regex.html
| 正则表达式 | 描述 | 示例 |
|---|---|---|
| ^ | 匹配行开始 | 如:/^sed/匹配所有以sed开头的行 |
| $ | 匹配行结束 | 如:/sed$/匹配所有以sed结尾的行 |
| . | 匹配一个非换行符的任意字符 | 如:/s.d/匹配s后接一个任意字符,最后是d |
| * | 匹配0个或多个字符 | 如:/*sed/匹配所有模板是一个或多个空格后紧跟sed的行 |
| [] | 匹配一个指定范围内的字符 | 如/[ss]ed/匹配sed和Sed |
| [^] | 匹配一个不在指定范围内的字符 | 如:/[^A-RT-Z]ed/匹配不包含A-R和T-Z的一个字母开头,紧跟ed的行 |
| .. | 匹配子串,保存匹配的字符 | 如s/loveable/\1rs,loveable被替换成lovers |
| & | 保存搜索字符用来替换其他字符 | 如s/love/*&*/,love这成**love** |
| \< | 匹配单词的开始 | 如:/\<love/匹配包含以love开头的单词的行 |
| > | 匹配单词的结束 | 如/love>/匹配包含以love结尾的单词的行 |
| x{m} | 重复字符x,m次 | 如:/0{5}/匹配包含5个0的行 |
| x{m,} | 重复字符x,至少m次 | 如:/0{5,}/匹配至少有5个0的行 |
| x{m,n} | 重复字符x,至少m次,不多于n次 | 如:/0{5,10}/匹配5~10个0的行 |
# 匹配含 gl,gf的行
$ grep "g[lf]" file
# 匹配输出序列名称行
$ grep ">.*" ref.fa
# 匹配输出含 google gooogle 的行
$ grep 'go\{2,3\}gl' file
# 删除所有开头是test的行
$ sed '/^test/'d file
# 删除含 google gooogle 的行
$ sed '/go\{2,3\}gl/d' file
# 只打印染色体
awk ' {if ($1 ~ /Chr/) print $0}' genome.gff
管道¶
管道命令(pipe)的作用,是将左侧命令的标准输出转换为标准输入,提供给右侧命令作为参数,使用 | 表示。同一行命令可以使用多个管道。使用管道可以省去很多中间文件
# 大小写转换
echo "hello world"|tr 'a-z' 'A-Z'
# 列出重复行
$ sort ref.fa |uniq -c
# 统计重复行
$ grep "gene" genome.gff|wc -l
# 打印压缩文件第一列
$ zcat genome.gff.gz |awk '{print $1}'
# 找出最常用的5个命令
$ history | awk '{print $2}' | sort | uniq -u | sort -rn | head -5
# 查看CPU型号
$ cat /proc/cpuinfo |grep name|cut -d : -f 2|uniq
# bwa比对后输出排序之后的bam文件
$ bwa mem -t 8 genome.fa read1.fq.gz read2.fq.gz | samtools sort -@8 -o output_sorted.bam
重定向¶
在shell交互环境下,默认的标准输入(standard input)和输出(standard output)是stdin和stdout,即键盘和屏幕:shell中运行的程序默认从键盘读取数据,默认将数据打印到屏幕上。 如果需要更改默认的输入和输出方向,则需要使用重定向功能。
数据重定向主要分为三类:
- stdin 表示标准输入,代码为0,使用<或<<操作符 : 符号<表示以文件内容作为输入 : 符号<<表示输入时的结束符号
- stdout 表示标准输出,代码为1,使用>或>>操作符 : 符号>表示以覆盖的方式将正确的数据输出到指定文件中 : 符号>>表示以追加的方式将正确的数据输出到指定文件中
- stderr 表示标准错误输出,代码为2,使用2>或2>>操作符 : 符号2>表示以覆盖的方式将错误的数据输出到指定文件中 : 符号2>>表示以追加的方式将错误的数据输出到指定文件中
# 取前100行生成新文件
$ head -n 100 file > new
# 提取 'genome.gff3' 中gene相关的部分生成新文件
$ awk '{if($3 == "gene" ){print $0}}' genome.gff3 > gene.gff3
# 将blast结果压缩
blastn -query ./ZS97_cds_10000.fa -db ./MH63_cds -outfmt 6 |gzip > ZS97_cds_10000.gz
# 将2个文件排序再按列合并,最后输出到新文件
$ paste <(sort file1 ) <(sort file2 ) > merge
# 将2个文件解压再按列合并,最后压缩输出到新文件
paste <(gzip -dc file1.gz ) <(gzip -dc file2.gz ) | gzip > merge.gz
# 将压缩文件作为输入
blastn -query <(gzip -dc ZS97_cds_10^6.fa.gz) -db ./MH63_cds -outfmt 6 |gzip > ZS97_cds_10000.gz
# 将fq文件转成fa
zcat ERR365991_1.fastq.gz |cat -n|awk '{if(NR%4 ==2) {n=int($1/4)+1;print ">"n"\n"$2}}' |gzip > reads.fa.gz
命令练习¶
Q1. 将文件 http://hpc.ncpgr.cn/data/seq.fa 下载至自己的目录下
cd ~/zhangsan/
wget http://hpc.ncpgr.cn/data/seq.fa
Q2. 使用 cat 查看 seq.fa 的内容
cat seq.fa
Q3. 使用 head 查看 seq.fa 前5行的内容
head -n5 seq.fa
Q4. 使用 tail 查看 seq.fa 后5行的内容
tail -n5 seq.fa
Q5. 使用 wc 统计 seq.fa 有多少行
wc -l seq.fa
Q6. 统计 seq.fa 中序列条数(即“>”开头的行数)
cat seq.fa|grep ">"|wc -l
Q7. 使用 grep 和 awk 筛选出 seq.fa 中的序列名称而不要描述部分
cat seq.fa|grep ">"|awk '{print $1}'
Q8. 将 seq.fa 中小写的碱基序列 atcg 换成大写的 ATCG,序列名称中的字母不变,结果输出到新的序列文件中 seq1.fa
sed -e '/^>/!s/[atcg]/\U&/g' seq.fa > seq1.fa
Q9. 计算这段序列的反向互补配对的序列 TTTTGAGTGGTGAAAGGTGAGAGGAATAGGAGTAAAAATGTGT
echo 'TTTTGAGTGGTGAAAGGTGAGAGGAATAGGAGTAAAAATGTGT'|tr 'ATCG' 'TAGC'|rev
Q10. 使用 awk 计算 seq.fa 中所有序列行的总碱基数(不含标题行)。
awk '!/^>/ {sum+=length} END {print sum}' seq.fa
Q11. 统计 seq.fa 中碱基出现次数
# 一行超长序列拆成单字符
grep -v '^>' seq.fa | fold -w1 | sort | uniq -c
Q12. 将文件 ~/data/genome.gff.gz 拷贝至自己的目录下然后解压为 genome.gff
cd ~/zhang/
cp ~/data/genome.gff.gz .
gzip -d genome.gff.gz
Q13. 统计 genome.gff 中有多少个基因
# 统计文件第3列为 gene 的数量
awk '$3=="gene"' genome.gff |wc -l
Q14. 将 genome.gff3 文件中代表基因、位于正链、且序列长度小于2000bp的行打印出来
awk '{if($3 == "gene" && $7 == "+" && ($5 -$4)<2000 ){print $0}}' genome.gff
vim¶
Linux下常用的文本编辑器,键盘操作,功能丰富,高度定制, 无数插件, 无限创(zheng)意(teng)
三种模式: 正常模式, 插入模式, 命令模式
vim filename 打开或新建文件,并将光标置于第一行首
常用操作¶
插入模式下执行
i: 在光标前a: 在光标后o: 在下一行新开一行O: 在上一行新开一行backspace/del键: 删除前后字符
正常模式下执行
j或↓: 光标下移k或↑: 光标上移h或←: 光标左移l或→: 光标右移^: 光标移至行首$: 光标移至行尾G: 光标移至文本最后一行数字+G: 光标移至指定的行ctrl+g: 光标跳到上次的行gg: 光标移至文本第一行回车键: 光标移至下一行ctrl+f: 向下翻页ctrl+b: 向上翻页
正常模式下执行
x: 删除光标后一个字符X: 删除光标前一个字符dd: 删除当前行- 在插入模式下可以使用backspace/del键删除字符
u: 撤销操作(类似于word中的ctrl+z)ctrl+r: 撤销u的操作,即回来撤销之前(类似于word中的ctrl+y)
正常模式下执行
v: 移动光标,选择复制范围; 再按v,取消选择V: 上下移动光标,选择整行; 再按V,取消选择ctrl+v: 上下移动光标,选择文本块; 选择特定的列; 再按ctrl+v,取消选择
正常模式下执行
y: 复制,配合选择命令,可以实现复制部分文本p: 在光标后粘贴复制内容yy: 复制整行
命令模式下执行
:w保存修改:wq保存退出(也可以在正常模式下用ZZ代替):!q强制退出:set nu显示文本行号:file显示当前文本文件名:1,$s/hello/HELLO/g将整个文本中的hello替换成HELLO:!ls执行linux命令,如ls
正常模式下执行
ctrl+w s: 水平分割ctrl+w v: 垂直分割ctrl+w hjkl: 在不同窗口间移动
参考: vim自动格式化对齐代码
正常模式下执行
==: 对光标所在行进行自动格式化对齐,会根据代码情况增加或减少缩进。可以在==前面加上数字,指定要同时处理多少行。例如,4==会格式化对齐当前行、以及后面的三行gg=G: 对整个文件都重新格式化对齐
ctrl + x -> ctrl +n: 通过目前正在编辑的这个「文件的内容文件」作为关键词,补齐;ctrl + x -> ctr + f: 以当前目录内的「文件名」作为关键词,予以补齐ctrl + x -> ctrl + o: 以扩展名作为语法补充,以 vim 内置的关键词,予以补齐
基本设置¶
在命令模式下可以对vim进行个性化设置,如显示行号、高亮当前行等,常见设置如下:
| 设置 | 含义 |
|---|---|
| :set nu、:set nonu | 设置与取消行号 |
| :set hlsearch、:set nohlsearch | hlsearch 是 high light search (高亮度搜索)。设置是否将搜索到的字符串反白设置。默认为 hlsearch |
| :set autoindent、:set noautoindent | 是否自动缩排?当你按下 Enter 编辑新的一行时,光标不会在行首,而是在于上一行第一个非空格符处对齐 |
| :set backup | 是否自动存储备份文件,一般是 nobackup 的,如果设置为 backup,那么当你更改任何一个文件时,则源文件会被另存一个文件名为 filename~ 的文件。如:编辑 hosts,设置 :set backup ,那么修改 hosts 时,在同目录下就会产生 hosts~ 的文件 |
| :set ruler | 右下角的状态栏说明,是否显示或不显示该状态的显示 |
| :set shwmode | 是否要显示 ---INSERT-- 之类的提示在左下角的状态栏 |
| :set backpace=(012) | 一般来说,如果我们按下 i 进入编辑模式后,可以利用退格键(baskpace)来删除任意字符的。但是某些 distribution 则不允许如此。此时,可以通过 backpace 来设置,值为 2 时,可以删除任意值;0 或 1 时,仅可删除刚刚输入的字符,而无法删除原本就已经存在的文字 |
| :set all | 显示目前所有的环境参数设置 |
| :syntax on 、 :syntax off | 是否依据程序相关语法显示不同颜色 |
| :set bg=dark、:set bg=light | 可以显示不同颜色色调,预设是 light。如果你常常发现批注的字体深蓝色是在很不容易看,就可以设置为 dark |
| :set cursorline | 高亮显示当前行,当前行显示一条长线 |
常用设置也可写入 ~/.vimrc 中
$ cat ~/.vimrc
" 该文件的注释是使用双引号表达
set hlsearch "高亮度反白
set backspace=2 "可随时用退格键删除
set autoindent "自动缩进
set ruler "可现实最后一列的状态
set showmode "左下角那一列的状态
set nu "在每一行的最前面显示行号
set bg=dark "显示不同的底色色调
syntax on "进行语法检验,颜色显示
set cursorline "高亮显示当前行,当前行显示一条长线
# 保存后,再次打开最明显的就是自动显示行号了,可见是生效了的
插件¶
利用vim插件可以实现更多功能和酷炫的效果,见 Vim 常用插件 & 插件管理
命令练习¶
Q1. 创建文件me.txt,使用 vim 打开 me.txt,然后输入以下内容
i am zhangsan
i am learning linux
Q2. 在vim中复制第2行内容,并将其粘贴5次
Q3. 将所有的am leaning 删除,并更改为 love
Q4. 删除最后2行
进程管理¶
进程(process)即运行中的程序,同一个程序可以有多个进程在运行。
PID :进程 ID, 每个进程都会从内核获取一个唯一的 ID 值。
作为用户需要了解自己正在跑的进程,用了多少CPU资源、内存等,知道怎么杀死进程、挂起进程、在后台跑程序、长期稳定地跑程序等。
$ top
top - 09:13:17 up 129 days, 17:21, 1 user, load average: 32.10, 27.60, 25.74
Tasks: 462 total, 9 running, 453 sleeping, 0 stopped, 0 zombie
%Cpu(s): 90.0 us, 5.5 sy, 0.0 ni, 4.6 id, 0.0 wa, 0.0 hi, 0.0 si, 0.0 st
KiB Mem : 39570169+total, 15617606+free, 23756960+used, 1956012 buff/cache
KiB Swap: 4194300 total, 2389376 free, 1804924 used. 15288067+avail Mem
PID USER PR NI VIRT RES SHR S %CPU %MEM TIME+ COMMAND
55164 user1 20 0 1191524 667856 2284 S 774.8 0.2 9:26.29 hisat2-align-s
18252 user2 20 0 22.8g 19.3g 1020 R 599.7 5.1 1458:45 overlapInCore
446439 user2 20 0 22.7g 19.2g 1020 R 599.7 5.1 2487:18 overlapInCore
9442 user2 20 0 22.9g 19.3g 1020 R 599.3 5.1 1754:11 overlapInCore
13186 user2 20 0 22.7g 19.2g 1024 R 599.3 5.1 1633:27 overlapInCore
53033 user3 20 0 69.1g 67.9g 1948 R 100.0 18.0 14:23.56 fastlmmc
53415 user3 20 0 69.4g 68.4g 1956 R 99.7 18.1 12:31.07 fastlmmc
55166 user1 20 0 4756 656 472 S 21.6 0.0 0:15.62 gzip
55165 user1 20 0 4756 660 472 R 20.3 0.0 0:14.64 gzip
55162 user1 20 0 137112 4268 596 R 8.0 0.0 0:05.83 perl
55163 user1 20 0 137112 4268 596 S 8.0 0.0 0:05.70 perl
29589 root 0 -20 25.6g 6.9g 2.3g S 1.7 1.8 8821:57 mmfsd
47500 root 0 -20 40136 8384 1392 S 0.3 0.0 93:21.17 lim
55383 root 20 0 166508 2760 1676 R 0.3 0.0 0:00.02 top
1 root 20 0 54424 3120 1568 S 0.0 0.0 27:26.10 systemd
2 root 20 0 0 0 0 S 0.0 0.0 0:09.81 kthreadd
3 root 20 0 0 0 0 S 0.0 0.0 2:47.41 ksoftirqd/0
8 root rt 0 0 0 0 S 0.0 0.0 0:43.65 migration/0
9 root 20 0 0 0 0 S 0.0 0.0 0:00.00 rcu_bh
10 root 20 0 0 0 0 S 0.0 0.0 122:40.09 rcu_sched
11 root 0 -20 0 0 0 S 0.0 0.0 0:00.00 lru-add-drain
12 root rt 0 0 0 0 S 0.0 0.0 0:22.52 watchdog/0
前台任务与后台任务¶
"前台任务"(foreground job)是独占命令行窗口的任务,只有运行完了或者手动中止该任务,才能执行其他命令。如果没有特殊操作,一般用户执行的命令为前台任务。
"后台任务"(background job)通常在不打扰用户其它工作的时候默默地执行(此时可以输入其他的命令)。其特点为:
- 继承当前session(对话)的标准输出(stdout)和标准错误(stderr)。因此,后台任务的所有输出依然会同步地在命令行下显示。
- 不再继承当前session的标准输入(stdin)。你无法向这个任务输入指令了。如果它试图读取标准输入,就会暂停执行(halt)。
可以看到,"后台任务"与"前台任务"的本质区别只有一个:是否继承标准输入。所以,执行后台任务的同时,用户还可以输入其他命令。使用nohup, &, bg, diwown, screen 等命令可让命令在后台运行。
常用命令¶
- top 动态显示系统运行状况 M c
- ps 输出系统所有进程状态 ps aux
- kill 杀死进程或作业 kill –s 9 18 19 -u
- killall 杀死指定名称的进程
- pgrep 查找进程
- nohup 将程序挂起运行, 账号退出不受影响, 一般和&配合使用
- disown 使已经在运行的用户进程 不受用户退出限制
- jobs 查看任务运行状态
- bg 将作业放在后台运行, 与&类似; fg, 将作业放在前台运行
- screen 命令行终端切换软件 -s -ls -d -r; 可尝试更强大的tmux
- & 将程序放后台运行
- Ctrl+z 暂停前台运行作业; Ctrl+c, 杀死前台运行的作业
- time 统计命令运行时间
# 查看当前系统运行的进程,每几秒刷新一次
# M 按进程使用内存排序
# c 查看进程的运行参数
# u 查看指定用户的进程
# q 退出
$ top
top - 09:13:17 up 129 days, 17:21, 1 user, load average: 32.10, 27.60, 25.74
Tasks: 462 total, 9 running, 453 sleeping, 0 stopped, 0 zombie
%Cpu(s): 90.0 us, 5.5 sy, 0.0 ni, 4.6 id, 0.0 wa, 0.0 hi, 0.0 si, 0.0 st
KiB Mem : 39570169+total, 15617606+free, 23756960+used, 1956012 buff/cache
KiB Swap: 4194300 total, 2389376 free, 1804924 used. 15288067+avail Mem
PID USER PR NI VIRT RES SHR S %CPU %MEM TIME+ COMMAND
55164 user1 20 0 1191524 667856 2284 S 774.8 0.2 9:26.29 hisat2-align-s
18252 user2 20 0 22.8g 19.3g 1020 R 599.7 5.1 1458:45 overlapInCore
446439 user2 20 0 22.7g 19.2g 1020 R 599.7 5.1 2487:18 overlapInCore
9442 user2 20 0 22.9g 19.3g 1020 R 599.3 5.1 1754:11 overlapInCore
13186 user2 20 0 22.7g 19.2g 1024 R 599.3 5.1 1633:27 overlapInCore
53033 user3 20 0 69.1g 67.9g 1948 R 100.0 18.0 14:23.56 fastlmmc
53415 user3 20 0 69.4g 68.4g 1956 R 99.7 18.1 12:31.07 fastlmmc
# 输出系统所有进程
$ ps aux
# 根据cpu使用率进行排序
ps -aux --sort -pcpu | less
# 根据内存使用来升序排序
ps -aux --sort -pmem | less
# 杀死进程id(pid)为14234的进程
$ kill 14234
# 强制杀死进程,使用信号9
$ kill -s 9 14234
# 挂起进程,即让进程暂停运行,但不杀掉进程。使用信号19
$ kill -s 19 14234
# 恢复被挂起的进程,即让被暂停的进程恢复运行。使用信号18
$ kill -s 18 14234
# 杀死bwa进程
$ killall bwa
# 杀死用户username的所有进程
$ killall -u username
# 查找所有bwa进程,返回进程id
$ pgrep bwa
9442
13186
18252
446439
# 将bwa放前台运行
$ bwa mem -t 8 genome.fa read1.fq.gz read2.fq.gz | samtools sort -@8 -o output_sorted.bam
# CTRL-z挂起
$ ^Z
[1]+ Stopped bwa mem -t 8 genome.fa read1.fq.gz read2.fq.gz | samtools sort -@8 -o output_sorted.bam
# 放在后台
$ bg %1
[1]+ bwa mem -t 8 genome.fa read1.fq.gz read2.fq.gz | samtools sort -@8 -o output_sorted.bam &
# 查看后台任务
$ jobs
[1]+ Running bwa mem -t 8 genome.fa read1.fq.gz read2.fq.gz | samtools sort -@8 -o output_sorted.bam &
# 将bwa放后台运行,即使远程ssh退出也不影响bwa运行
$ nohup bwa mem -t 8 genome.fa read1.fq.gz read2.fq.gz | samtools sort -@8 -o output_sorted.bam &
# 查看后台任务状态
$ jobs
[1] Running sleep 60 &
[2] Running sleep 60 &
$ jobs
[1] Done sleep 60
[2] Done sleep 60
# 程序在前台运行了一段时间,在不杀掉程序重跑的情况下,将前台作业变为后台作业
# 不放在后台的命令
$ bwa mem -t 8 genome.fa read1.fq.gz read2.fq.gz | samtools sort -@8 -o output_sorted.bam
# CTRL-z挂起
$ ^Z
[1]+ Stopped bwa mem -t 8 genome.fa read1.fq.gz read2.fq.gz | samtools sort -@8 -o output_sorted.bam
# 放在后台
$ bg %1
[1]+ bwa mem -t 8 genome.fa read1.fq.gz read2.fq.gz | samtools sort -@8 -o output_sorted.bam &
# 查看后台任务
$ jobs
[1]+ Running bwa mem -t 8 genome.fa read1.fq.gz read2.fq.gz | samtools sort -@8 -o output_sorted.bam &
# 再使用disown命令
$ disown -h %1
# 查看进程
$ ps -aux |grep bwa
# 在Screen环境下,所有的会话都独立的运行,并拥有各自的编号、输入、输出和窗口缓存。
# 用户可以通过快捷键在不同的窗口下切换,并可以自由的重定向各个窗口的输入和输出。Screen实现了基本的文本操作,如复制粘贴等;
# 还提供了类似滚动条的功能,可以查看窗口状况的历史记录。窗口还可以被分区和命名,还可以监视后台窗口的活动。
# 会话共享 Screen可以让一个或多个用户从不同终端多次登录一个会话,并共享会话的所有特性(比如可以看到完全相同的输出)。
# 它同时提供了窗口访问权限的机制,可以对窗口进行密码保护。
# 新建名为bwa的session
$ screen -S bwa
# 列出所有的session
$ screen -ls
# 远程detach bwa的session
# 在当前这个screen窗口中时,可用ctrl+a ctrl+a 来detach当前session
$ screen -d bwa
# 回到bwa这个session
$ screen -r bwa
# screen配置
$ cat ~/.screenrc
# 显示状态栏,方便区分不同的screen session
hardstatus on
hardstatus alwayslastline
hardstatus string "%{= kw}%-w%{= kG}%{+b}[%n %S %t]%{-b}%{= kw}%+w %=[%Y-%m-%d %c]%{g}%{-}"
# 支持鼠标上下滚动
termcapinfo xterm ti@:te@
# 默认使用bash内置time命令,输出程序运行时间
$ time grep -f file.txt parttern.txt
real 3m3.760s
user 2m47.634s
sys 0m15.635s
# 也可以使用外部time程序 /usr/bin/time
# /usr/bin/time -v 输出各种资源消耗
# \time -v 也可以代替
# time -v 会报错
$ /usr/bin/time -v grep -f file.txt parttern.txt
# /usr/bin/time -f 可指定输出的资源,有多种选项可以使用
# 如 -f "%e %p %M" 可输出程序运行时间(s)、CPU使用占比、最大使用内存(kb)
$ /usr/bin/time -f "%e %P %M" grep -f file.txt parttern.txt
178.44 99% 17553368
# 可以使用alias改写time,或设置TIME环境变量设置time命令输出格式
$ alias time="$(which time) -f '%E real,\t%U user,\t%S sys,\t%K amem,\t%M mmem'"
0:16.89 real, 16.46 user, 0.38 sys, 0 amem, 458512 mmem
$ export TIME='%E real,\t%U user,\t%S sys,\t%t amem,\t%M mmem'
$ /usr/bin/time grep -f file.txt parttern.txt > xxx
0:17.26 real, 16.79 user, 0.41 sys, 0 amem, 457092 mmem
系统信息查看¶
使用Linux系统提供的工具和命令查看磁盘使用、系统负载、内存使用、系统版本等信息。
常用命令¶
- df 显示系统磁盘使用情况 -h
- free 显示系统内存使用情况 -h
- ip 显示系统IP配置
- lscpu 查看CPU配置 lscpu
- lsb_release 查看操作系统版本 -a
- uname 内核版本 -a
- lspci 查看PCI设备,CPU、GPU、网卡等
- uptime 查看系统运行时间、负载信息
- top 查看系统负载等信息
- who 查看登录用户
# 查看/public目录磁盘使用情况
$ df -h /public
Filesystem Size Used Avail Use% Mounted on
public 5.5P 4.3P 1.3P 78% /public
# 查看内存使用情况
$ free -g
total used free shared buff/cache available
Mem: 93 66 12 4 15 16
Swap: 0 0
# 查看IP地址
$ ip a
$ lscpu
Architecture: x86_64
CPU op-mode(s): 32-bit, 64-bit
Byte Order: Little Endian
CPU(s): 40
On-line CPU(s) list: 0-39
Thread(s) per core: 2
Core(s) per socket: 10
Socket(s): 2
NUMA node(s): 2
Vendor ID: GenuineIntel
CPU family: 6
Model: 85
Model name: Intel(R) Xeon(R) Gold 5115 CPU @ 2.40GHz
Stepping: 4
CPU MHz: 2400.000
BogoMIPS: 4800.00
Virtualization: VT-x
L1d cache: 32K
L1i cache: 32K
L2 cache: 1024K
L3 cache: 14080K
NUMA node0 CPU(s): 0-9,20-29
NUMA node1 CPU(s): 10-19,30-39
Flags: fpu vme de pse tsc msr pae mce cx8 apic sep mtrr pge mca cmov pat pse36 clflush dts acpi mmx fxsr sse sse2 ss ht tm pbe syscall nx pdpe1gb rdtscp lm constant_tsc art arch_perfmon pebs bts rep_good nopl xtopology nonstop_tsc aperfmperf eagerfpu pni pclmulqdq dtes64 monitor ds_cpl vmx smx est tm2 ssse3 sdbg fma cx16 xtpr pdcm pcid dca sse4_1 sse4_2 x2apic movbe popcnt tsc_deadline_timer aes xsave avx f16c rdrand lahf_lm abm 3dnowprefetch epb cat_l3 cdp_l3 intel_ppin intel_pt mba tpr_shadow vnmi flexpriority ept vpid fsgsbase tsc_adjust bmi1 hle avx2 smep bmi2 erms invpcid rtm cqm mpx rdt_a avx512f avx512dq rdseed adx smap clflushopt clwb avx512cd avx512bw avx512vl xsaveopt xsavec xgetbv1 cqm_llc cqm_occup_llc cqm_mbm_total cqm_mbm_local ibpb ibrs stibp dtherm ida arat pln pts hwp_epp pku ospke spec_ctrl intel_stibp
# 查看系统版本, 也可使用 cat /etc/redhat-release。不同linux发行版本查看方式略有不同。
$ lsb_release -a
LSB Version: :core-4.1-amd64:core-4.1-noarch:cxx-4.1-amd64:cxx-4.1-noarch:desktop-4.1-amd64:desktop-4.1-noarch:languages-4.1-amd64:languages-4.1-noarch:printing-4.1-amd64:printing-4.1-noarch
Distributor ID: CentOS
Description: CentOS Linux release 7.5.1804 (Core)
Release: 7.5.1804
Codename: Core
$ cat /etc/redhat-release
CentOS Linux release 7.5.1804 (Core)
# 查看操作系统内存版本
$ uname -a
Linux localhost 3.10.0-862.el7.x86_64 #1 SMP Fri Apr 20 16:44:24 UTC 2018 x86_64 x86_64 x86_64 GNU/Linux
# 查看系统运行时间和负载
$ uptime
11:11:42 up 69 days, 23:25, 200 usergroup, load average: 6.10, 8.08, 6.46
# 查看系统负载、内存消耗、进程运行情况
$ top
top - 09:13:17 up 129 days, 17:21, 1 user, load average: 32.10, 27.60, 25.74
Tasks: 462 total, 9 running, 453 sleeping, 0 stopped, 0 zombie
%Cpu(s): 90.0 us, 5.5 sy, 0.0 ni, 4.6 id, 0.0 wa, 0.0 hi, 0.0 si, 0.0 st
KiB Mem : 39570169+total, 15617606+free, 23756960+used, 1956012 buff/cache
KiB Swap: 4194300 total, 2389376 free, 1804924 used. 15288067+avail Mem
PID USER PR NI VIRT RES SHR S %CPU %MEM TIME+ COMMAND
55164 user1 20 0 1191524 667856 2284 S 774.8 0.2 9:26.29 hisat2-align-s
18252 user2 20 0 22.8g 19.3g 1020 R 599.7 5.1 1458:45 overlapInCore
446439 user2 20 0 22.7g 19.2g 1020 R 599.7 5.1 2487:18 overlapInCore
9442 user2 20 0 22.9g 19.3g 1020 R 599.3 5.1 1754:11 overlapInCore
13186 user2 20 0 22.7g 19.2g 1024 R 599.3 5.1 1633:27 overlapInCore
53033 user3 20 0 69.1g 67.9g 1948 R 100.0 18.0 14:23.56 fastlmmc
53415 user3 20 0 69.4g 68.4g 1956 R 99.7 18.1 12:31.07 fastlmmc
# 查看登录用户
$ who
user1 pts/0 2021-09-17 08:49 (192.168.1.31)
user2 pts/1 2021-09-16 11:51 (192.168.1.32)
user3 pts/2 2021-09-17 14:22 (192.168.1.33)
user4 pts/3 2021-09-17 18:22 (192.168.1.34)
user5 pts/5 2021-09-16 23:32 (192.168.1.35)
user6 pts/6 2021-09-17 17:38 (192.168.1.36)
命令练习¶
Q1. 列出当前节点登录的用户并去重排序后将其写入 user_list.txt 文件中
who|awk '{print $1}'|sort |uniq > user_list.txt
简单shell编程¶
shell可以理解为一种脚本语言,只需要一个能编写代码的文本编辑器和一个能解释执行的脚本解释器就可以。
shell脚本的本质是组合Linux命令, 变量, 数据结构,逻辑语句和for while if 等流程控制语句,由shell程序解析执行的脚本文本文件。
第一个shell脚本 test.sh
#!/bin/bash
echo "Hello World !"
chmod +x ./test.sh #使脚本具有执行权限
./test.sh #执行脚本
变量¶
普通变量:
# 3种定义变量的方式
# 最常见的变量定义方式
$ string="this is a string"
# 将ls /home/的输出存储到变量file中
$ file=$(ls /home/)
# 将cat file的输出存储到变量text中
$ text=`cat file`
# 使用变量,$string或${string}
echo "${string}, ${file}"
环境变量:$PWD, $HOME, $PATH ...,env 命令可以打印所有环境变量
脚本传参变量:$1 $2 ...
运算符¶
逻辑运算符
| 操作符 | 说明 | 举例 |
|---|---|---|
| && | 逻辑的 AND | [[ $a -lt 100 && $b -gt 100 ]] 返回 false |
| || | 逻辑的 OR | [[ $a -lt 100 || $b -gt 100 ]] 返回 true |
#!/bin/bash
a=10
b=20
if [[ $a -lt 100 && $b -gt 100 ]]
then
echo "返回 true"
else
echo "返回 false"
fi
if [[ $a -lt 100 || $b -gt 100 ]]
then
echo "返回 true"
else
echo "返回 false"
fi
| 操作符 | 说明 | 举例 |
|---|---|---|
| -d file | 检测文件是否是目录,如果是,则返回 true。 | [ -d $file ] 返回 false。 |
| -f file | 检测文件是否是普通文件(既不是目录,也不是设备文件),如果是,则返回 true。 | [ -f $file ] 返回 true。 |
| -r file | 检测文件是否可读,如果是,则返回 true。 | [ -r $file ] 返回 true。 |
| -w file | 检测文件是否可写,如果是,则返回 true。 | [ -w $file ] 返回 true。 |
| -x file | 检测文件是否可执行,如果是,则返回 true。 | [ -x $file ] 返回 true。 |
| -s file | 检测文件是否为空(文件大小是否大于0),不为空返回 true。 | [ -s $file ] 返回 true。 |
| -e file | 检测文件(包括目录)是否存在,如果是,则返回 true。 | [ -e $file ] 返回 true。 |
#!/bin/bash
file="/home/user/test.sh"
if [[ -f $file && -x $file ]]
then
echo "文件可执行"
fi
流程控制¶
#!/bin/bash
if [[$1 == "memory" ]]
then
free -m
else
lscpu
fi
#!/bin/bash
for i in node{1..10}
do
echo ${i}
ssh ${i} "free -g"
done
#!/bin/bash
file=/etc/hosts
while read line
do
echo ${line}
done < "$file"
调试¶
sh [-nvx] scripts.sh
选项与参数:
-n:不执行 script,仅检查语法问题
-v:执行 script 前,先将 scripts 内容输出到屏幕上
-x:将执行到的 script 内容显示到屏幕上,相当于 debug 了
综合¶
#!/bin/sh
fa=""
cat file.fa|while read line;do
if [[ ${line} =~ \> ]];then
echo ${line}
echo ${fa}|tr "ATCG "TAGC"|rev
fa=""
else
fa=${fa}${line}
fi
done
#!/bin/sh
cd $PWD
thread=5
for sample in ${HOME}/exercise/raw_data/*trim_1.fq.gz;do
index=$(basename $sample|sed 's/_trim_1\.fq\.gz//')
dir=$(dirname $sample)
if [[ -f $dir/${index}_trim_1.fq.gz && -f $dir/${index}_trim_1.fq.gz ]] ;then
#align
hisat2 -x ${HOME}/exercise/genome/reference \
-1 $dir/${index}_trim_1.fq.gz -2 $dir/${index}_trim_2.fq.gz -p ${thread} -S ${index}.sam
#sam2bam
samtools view -bs ${index}.sam > ${index}.bam
#sort bam
samtools sort -@${thread} ${index}.bam ${index}_sort.bam
#rm sam bam
rm ${index}.bam ${index}.sam
#assembly
stringtie ${index}_sort.bam -G ${HOME}/exercise/genome/reference.gtf \
-o ${index}.gtf -p ${thread}
#stat
transcript_stat.pl ${index}.gtf > ${index}_stat
fi
done
命令练习¶
Q1. 列出当前目录下所有文件和目录,并判断其是否为普通文件,是返回 filename is a file,否则返回 filename is not a file。
#!/bin/bash
for item in `ls`; do
if [[ -f "$item" ]]; then
echo "$item is a file"
else
echo "$item is not a file"
fi
done
参考资料¶
https://biohpc.cornell.edu/lab/doc/Linux_exercise_part1.pdf
https://sites.ualberta.ca/~stothard/downloads/linux_for_bioinformatics.pdf
Linux¶
# Q0:准备,假设此时的练习用户姓名为 张三,则建立练习目录 ~/zhangsan/
# 时刻谨记自己当前所在的目录,不了解时使用pwd命令查看,后面每步练习开始时都是回到自己的练习主目录中,如张三的为 ~/zhangsan/
$ mkdir ~/zhangsan/
$ cd ~/zhangsan/
$ pwd
# Q1: 在任意文件夹下面创建形如 1/2/3/4/5/6/7/8/9 格式的文件夹系列
$ cd ~/zhangsan/
$ mkdir -p 1/2/3/4/5/6/7/8/9
# Q2:在创建好的文件夹下面(1/2/3/4/5/6/7/8/9) ,里面创建文本文件 me.txt
$ cd ~/zhangsan/
$ cd 1/2/3/4/5/6/7/8/9
$ touch me.txt
# Q3:使用vim编辑器,在文本文件 me.txt 里面输入以下内容
Go to: http://hpc.ncpgr.cn/
I love bioinfomatics.
And you ?
$ cd ~/zhangsan/
$ vim me.txt
$ cat me.txt
Go to: http://hpc.ncpgr.cn/
I love bioinfomatics.
And you ?
# Q4:删除上面创建的文件夹 1/2/3/4/5/6/7/8/9 及文本文件 me.txt
$ cd ~/zhangsan/
$ ls
1
$ rm -r ./1
$ ls
# Q5:在任意文件夹下面创建 folder1~5这5个文件夹,然后每个文件夹下面继续创建 folder1~5这5个文件夹
# 方法1
$ cd ~/zhangsan/
$ for i in {1..5}; do for j in {1..5}; do mkdir -p folder_${i}/folder_${j};done;done
$ tree
# 方法2
$ mkdir -p folder_{1..5}/folder_{1..5}
# Q6:在第五题创建的每一个文件夹下面都 创建第二题文本文件 me.txt ,内容也要一样
$ cd ~/zhangsan/
$ vim me.txt
$ for i in {1..5}; do for j in {1..5}; do cp ./me.txt folder_${i}/folder_${j};done;done
# Q7: 再次删除掉前面几个步骤建立的文件夹及文件
$ cd ~/zhangsan/
$ rm -r ./fold*
# Q8: 下载 http://hpc.ncpgr.cn/data/test.bed 文件,后在里面选择含有 H3K4me3 的那一行是第几行,该文件总共有几行。
$ cd ~/zhangsan/
$ wget http://hpc.ncpgr.cn/data/test.bed
$ cat -n test.bed | grep H3K4me3
$ wc test.bed
# Q9: 下载 http://hpc.ncpgr.cn/data/rmDuplicate.zip 文件,并且解压,查看里面的文件夹结构
$ cd ~/zhangsan/
$ wget http://hpc.ncpgr.cn/data/rmDuplicate.zip
$ unzip rmDuplicate.zip
$ tree rmDuplicate
# Q10: 打开第九题解压的文件,进入 rmDuplicate/samtools/single 文件夹里面,查看后缀为 .sam 的文件,搞清楚 生物信息学里面的SAM/BAM 定义是什么。
# SAM(The Sequence Alignment / Map format)为序列比对文件的格式;
# BAM为SAM的二进制文件,主要是为了节约空间;可以用samtools工具实现sam和bam文件之间的转化。
$ cd ~/zhangsan/
$ cd rmDuplicate/samtools/single
$ ls -lh *.sam
$ head tmp.sam
$ less -SN tmp.sam
$ wc tmp.sam
# Q11: 安装 samtools 软件到成自己的练习目录内
$ cd ~/zhangsan/
$ mkdir -p ~/zhangsan/opt/samtools/
$ wget https://github.com/samtools/samtools/releases/download/1.3.1/samtools-1.3.1.tar.bz2
# 如因网络问题不能下载,使用 ctrl+c 组合键中断下载,然后从~/data/中拷贝一份
$ cp ~/data/samtools-1.3.1.tar.bz2 .
$ tar xf samtools-1.3.1.tar.bz2
$ cd samtools-1.3.1
# --prefix 参数指定安装目录
$ ./configure --prefix=${HOME}/zhangsan/opt/samtools/
$ make
$ make install
# 设置环境变量
$ export PATH=${HOME}/zhangsan/opt/samtools/bin:$PATH
# 测试是否安装成功
$ samtools --help
# 查看samtools的安装位置
$ which samtools
# Q12: 打开 后缀为BAM 的文件,找到产生该文件的命令。提示一下命令是bowtie2-align-s
# 这里的bam文件在上面下载的rmDuplicate.zip 中
$ cd ~/zhangsan/
$ cd rmDuplicate/samtools/single/
$ samtools view -h tmp.sorted.bam | grep CL
# Q13: 根据上面的命令,找到使用的参考基因组 hg38 具体有多少条染色体。
$ cd ~/zhangsan/
$ cd rmDuplicate/samtools/single/
$ samtools view -h tmp.rmdup.bam | grep SN | wc
$ samtools view -h tmp.rmdup.bam | grep SN | cut -f 2 | sort | uniq -c
# Q14: 上面的后缀为BAM 的文件的第二列,只有 0 和 16 两个数字,用 cut/sort/uniq等命令统计它们的个数。
$ cd ~/zhangsan/
$ cd rmDuplicate/samtools/single/
$ samtools view -h tmp.rmdup.bam | grep -v "^@" > tmp.txt
$ cut -f 2 tmp.txt | sort | uniq -c
# Q15: 重新打开 rmDuplicate/samtools/paired 文件夹下面的后缀为BAM 的文件,再次查看第二列,并且统计
$ cd ~/zhangsan/
$ cd rmDuplicate/samtools/single/
$ cd ../paired/
$ samtools view -h tmp.rmdup.bam | grep -v "^@" > tmp.txt
$ cut -f 2 tmp.txt | sort | uniq -c
# Q16: 下载 http://hpc.ncpgr.cn/data/sickle-results.zip 文件,并且解压,查看里面的文件夹结构, 这个文件有2.3M。
$ cd ~/zhangsan/
$ wget http://hpc.ncpgr.cn/data/sickle-results.zip
$ unzip sickle-results.zip
$ tree sickle-results
# Q17: 解压 sickle-results/single_tmp_fastqc.zip 文件,并且进入解压后的文件夹,找到 fastqc_data.txt 文件,并且搜索该文本文件以 >>开头的有多少行?
$ cd ~/zhangsan/
$ cd sickle-results/
$ unzip single_tmp_fastqc.zip
$ cd single_tmp_fastqc/
$ grep -c "^>>" fastqc_data.txt
# Q18: 下载 http://hpc.ncpgr.cn/data/hg38.tss 文件,去NCBI找到TP53/BRCA1等自己感兴趣的基因对应的 refseq数据库 ID,然后找到它们的hg38.tss 文件的哪一行。
# https://www.ncbi.nlm.nih.gov/gene/7157
# https://www.ncbi.nlm.nih.gov/gene/7157#reference-sequences
$ cd ~/zhangsan/
$ wget http://hpc.ncpgr.cn/data/hg38.tss
$ grep -n NM_000546 hg38.tss #TP53第一个refseq的ID
# Q19: 解析hg38.tss 文件,统计每条染色体的基因个数。
$ cd ~/zhangsan/
$ cut -f 2 hg38.tss | sort | uniq -c | sort -r | head -20
# Q20: 解析hg38.tss 文件,统计NM和NR开头的熟练,了解NM和NR开头的含义。
# NM 为转录产物序列,即成熟mNA转录本序列;
# NP指蛋白产物,主要是全长转录组氨基酸序列,但也有一些自由部分蛋白质的部分氨基酸的序列。
$ cd ~/zhangsan/
$ cut -f 1 hg38.tss | cut -c 1-2 | sort | uniq -c
LSF¶
参考 LSF 调度器使用
提取序列¶
将下面的代码写入作业脚本extract_seq.lsf,然后提交作业 bsub < extract_seq.lsf。
#BSUB -J extract
#BSUB -n 1
#BSUB -R span[hosts=1]
#BSUB -o %J.out
#BSUB -e %J.err
#BSUB -q test
# 生成随机数
rd=$(echo $RANDOM)
# 加载环境变量
export PATH="$HOME/data/bin/:$PATH"
# 从原始数据随机提取5%的数据
seqkit sample -s ${rd} -p 0.05 ~/data/RICasmRSYHSD3_1.fq.gz > sample_1.fq
seqkit sample -s ${rd} -p 0.05 ~/data/RICasmRSYHSD3_2.fq.gz > sample_2.fq
# 压缩fq文件,压缩后这两个文件变为了sample_1.fq.gz sample_2.fq.gz
gzip sample_1.fq sample_2.fq
比对¶
将下面的代码写入作业脚本bwa.lsf,然后提交作业 bsub < bwa.lsf。
#BSUB -J bwa
#BSUB -n 5
#BSUB -R span[hosts=1]
#BSUB -o %J.out
#BSUB -e %J.err
#BSUB -q test
# 加载环境变量
export PATH="$HOME/data/bin/:$PATH"
bwa mem -t ${LSB_DJOB_NUMPROC} ~/data/MH63RS3.fasta sample_1.fq.gz sample_2.fq.gz > sample.sam
sam转bam¶
将下面的代码写入作业脚本sam2bam.lsf,然后提交作业 bsub < sam2bam.lsf。
#BSUB -J sam2bam
#BSUB -n 2
#BSUB -R span[hosts=1]
#BSUB -o %J.out
#BSUB -e %J.err
#BSUB -q test
# 加载环境变量
export PATH="$HOME/data/bin/:$PATH"
samtools view -@${LSB_DJOB_NUMPROC} -b sample.sam > sample.bam
排序¶
将下面的代码写入作业脚本sort.lsf,然后提交作业 bsub < sort.lsf。
#BSUB -J sort
#BSUB -n 2
#BSUB -R span[hosts=1]
#BSUB -o %J.out
#BSUB -e %J.err
#BSUB -q test
# 加载环境变量
export PATH="$HOME/data/bin/:$PATH"
samtools sort -@${LSB_DJOB_NUMPROC} sample.bam -O sample_sorted.bam
# 删除中间文件
rm sample.sam sample.bam
多瑙¶
参考 多瑙调度器使用
提取序列¶
将下面的代码写入作业脚本extract_seq.dsu。
#!/bin/bash
#DSUB -n extract
#DSUB -R 'cpu=1'
#DSUB -o extract_%J.out
#DSUB -aa
#DSUB -q arm
# 生成随机数
rd=$(echo $RANDOM)
# 加载环境变量
source $HOME/data/bin/setenv.sh
# 从原始数据随机提取5%的数据
seqkit sample -s ${rd} -p 0.05 ~/data/RICasmRSYHSD3_1.fq.gz > sample_1.fq
seqkit sample -s ${rd} -p 0.05 ~/data/RICasmRSYHSD3_2.fq.gz > sample_2.fq
# 压缩fq文件,压缩后这两个文件变为了sample_1.fq.gz sample_2.fq.gz
gzip sample_1.fq sample_2.fq
提交作业
$ chmod +x extract_seq.dsu
$ dsub -s extract_seq.dsu
比对¶
将下面的代码写入作业脚本bwa.dsu。
#!/bin/bash
#DSUB -n bwa
#DSUB -R 'cpu=5'
#DSUB -o bwa_%J.out
#DSUB -aa
#DSUB -q arm
# 加载环境变量
source $HOME/data/bin/setenv.sh
bwa mem -t 5 ~/data/MH63RS3.fasta sample_1.fq.gz sample_2.fq.gz > sample.sam
提交作业
$ chmod +x bwa.dsu
$ dsub -s bwa.dsu
sam转bam¶
将下面的代码写入作业脚本sam2bam.dsu。
#!/bin/bash
#DSUB -n sam2bam
#DSUB -R 'cpu=2'
#DSUB -o sam2bam_%J.out
#DSUB -aa
#DSUB -q arm
# 加载环境变量
source $HOME/data/bin/setenv.sh
samtools view -@2 -b sample.sam > sample.bam
提交作业
$ chmod +x sam2bam.dsu
$ dsub -s sam2bam.dsu
排序¶
将下面的代码写入作业脚本sort.dsu。
#!/bin/bash
#DSUB -n sort
#DSUB -R 'cpu=2'
#DSUB -o sort_%J.out
#DSUB -aa
#DSUB -q arm
# 加载环境变量
source $HOME/data/bin/setenv.sh
samtools sort -@2 sample.bam -O sample_sorted.bam
# 删除中间文件
rm sample.sam sample.bam
$ chmod +x sort.dsu
$ dsub -s sort.dsu
- 列转换成行
#每行用逗号分隔 cat file.txt |tr "\n" ","|sed -e 's/,$/\n/' #每行用空格分隔 cat file.txt |tr "\n" " "|sed -e 's/ $/\n/' - 找到当前目录下含有字符串 rice 的文件
find . -type f|xargs -i grep -H "rice" {} -
shell参数替换
用法比较多,这里举个例子
${parameter%word}${parameter%%word}从尾开始扫描word,将匹配word正则表达式的字符过滤掉
%为最短匹配,%%为最长匹配
如下面的用法可以方便地去掉后缀提取样本id
ls raw/*_1.fastq.gz|while read i ;do id=${i%%_1.fastq.gz} bsub -J bwa_${id} -n 10 -o ${id}.out -e ${id}.err "bwa mem -t 8 ref.fa ${id}_1.fastq.gz ${id}_2.fastq.gz | samtools sort -@8 -o ${id}_srt.bam" done -
basename 取样本名
basename显示 去掉 目录成分 后的NAME. 如果 指定了SUFFIX, 就 同时 去掉 拖尾的SUFFIX。* perl解释器替换$ basename /path/to/sampleid.fastq.gz .fastq.gz sampleid#将/usr/local/bin/perl -w 替换为/usr/bin/env perl perl -p -i -e 's#/usr/local/bin/perl -w#/usr/bin/env perl#' scripts/*pl #将/usr/local/bin/perl -w 替换为/usr/bin/env perl for i in `find ./ -name "*.pl"`;do sed -i "s/bin\/perl /bin\/env perl/g" $i ;done -
shell脚本执行错误自动退出
写较长的流程shell脚本,可能会遇到中间执行错误,此时脚本仍然会往后继续执行,在脚本开头加入
set -e命令可以使脚本出错即停止运行.#!/bin/bash set -e -
不规则文件重命名
将类似
D33-1_L2_123X45.R2.fastq.gz的文件批量重命名为D33-1_2.fastq.gz。将所有文件名重命名,list文件中第一列为原字符串、第二列为重命名字符串。for i in *gz; do mv $i `echo $i |sed 's/L._...X...R//'`;done$ awk '{print "rename '' "$1" "$2" '' *.gz"}' list |sh -
grep命令加速
服务器现有版本(2.2)的grep匹配多行的pattern list的时候非常慢,如代码:
grep -vwf position_list genotype.csv > filtered.genotype如果这个pettern和文件上万行的话,总时长超过了数个小时,3.2之后的grep大概只用1s。
集群上安装了最新的版本 module load grep/3.3
少量数据测试结果如下:
$ time grep -vwf Chr01.bad_list_4000 splitChr01_1000.csv > test real 0m12.384s user 0m12.125s sys 0m0.223s $ module load grep/3.3 $ time grep -vwf Chr01.bad_list_4000 splitChr01_1000.csv > test real 0m0.015s user 0m0.006s sys 0m0.007s -
scp 后台下载数据
使用scp在不同服务器之间传数据,当数据量很大时,scp需要传很久,为防止掉线,可能想用nohup &的方法将scp放在后台运行,但实际做起来发现行不通。我们可以采用下面的方法。
nohup scp file username@ip:/home/username/dir然后输入密码开始传数据- 按下
ctrl+z - 使用jobs命令查看作业号,比如scp的作业号为1。然后bg %1,则可将scp任务放入后台,不用担心掉线
如果忘了使用nohup命令,则可以使用disown命令来补救,参见这篇文档 Linux 技巧:让进程在后台可靠运行的几种方法
这种情况也可以使用rsync传数据,可自动跳过已经传完的数据;使用screen 防止网络不稳定掉线
-
tv
csv文件展示
-
删除特殊文件
# 对于特殊字符列如<>\*开头的文件,删除加引号 $ rm "<>\*" rm: remove regular file ‘<>\\*’? y # 对于-开头的文本,删除使用- - # 使用删除加目录也可以,rm ./-B.file $ rm -- -B.file rm: remove regular file ‘-B.file’? y # 对于特殊字符!*,要增加转义字符 # linux中很多字符有着特殊的含义,在前面加上转义字符,就可以当成普通字符使用。 $ rm \!* rm: remove regular file ‘!*’? y # 按照节点号删除 # 乱码文件删除可使用这种方式 $ ls -i ./-B.file 1446218 ./-B.file $ find ./ -inum 1446218 -exec rm {} \; - 替换换行符/多行变单行的几种方法
# 用xargs为echo传入参数,默认是把换行符替换成空格【推荐】 cat file.txt | xargs echo # 用tr把换行符替换成空格【推荐】 cat file.txt | tr '\n' ' ' # 用paste替换;-s是一次处理文件的所有行,而非并行处理每一行;-d指定分割符 cat file.txt | paste -sd " " # 用sed处理;sed按行处理所以每次处理会自动添加换行符,:a在代码开始处设置标记a,代码执行结尾处通过跳转命令ta重新跳转到标号a处,重新执行代码,递归每行;N表示读入下一行【很慢】 sed ':a;N;s/\n/ /g;ta' file.txt # 用awk处理;ORS(Output Record Separator)设置输出分隔符,把换行符换成空格。head -c -1代表截取文件除了最后一个字符的字符,用于去掉文本末多的分隔符,根据情况使用 awk 'ORS=" "' file.txt |head -c -1 # 用awk处理,将RS(record separator)设置成EOF(end of file),即将文件视为一个记录;再通过gsub函数将换行符\n替换成空格 awk BEGIN{RS=EOF}'{gsub(/\n/," ");print}' file.txt -
终端记录 script 可以记录终端中所有的操作和输出,方便做分析记录。
-
程序日志
跑程序时如需要需要记录日志,可以用两种方式。
方法1:
记录单行命令的日志
$ { gatk HaplotypeCaller --native-pair-hmm-threads 64 -L chr20 -R hg38.fa -I ERR194146_sort_redup.bam -O ERR194146.vcf.gz ;} 2>&1 | tee gatk_hc.log方法2:
记录脚本的日志
#!/bin/bash logfile=gatk_hc.log exec >$logfile 2>&1 gatk HaplotypeCaller --native-pair-hmm-threads 64 -L chr20 -R hg38.fa -I ERR194146_sort_redup.bam -O ERR194146.vcf.gz
- putty 非常小巧的ssh开源登录工具,功能比较单一,一般临时应急使用。
- KiTTY 基于putty的开源ssh登录工具,相比putty功能更多,便携版下载
- xshell 功能强大的ssh老牌登录工具,正版软件为商业收费软件,一般用户可以使用免费版本,需要同时下载安装xshell和xftp。
- mobaxterm 最近两年流行起来的ssh登录工具,功能多样,有免费和收费两个版本,免费版本有一定功能限制,最主要的是最多只能保存12个账号(session)。功能演示
- tabby 是一个可高度配置的终端模拟器和 SSH 客户端,支持 Windows,macOS 和 Linux,具有非常漂亮的界面。
- terminus 支持 Windows,macOS 和 Linux。
- WindTerm 支持 Windows,macOS 和 Linux。
参考: 各种ssh工具
git bash¶
Git Bash在Windows上模拟bash环境,允许用户在Windows操作系统上使用Bash shell和大多数标准Linux命令:文件操作、文本操作、文本编辑、进程管理等。Git Bash软件体积相对较小,安装简单,可以快速获得一个基本的linux环境用于练习或简单数据处理。
下载地址:https://git-scm.com/download/win
选择 64-bit Git for Windows Setup 下载安装。
安装完成之后,在开始菜单中找到git bash打开即可,或鼠标点击桌面,然后右键->git bash,如下,可以看到有很多常用的linux命令,使用这些命令也可以处理地Windows下的文件和数据。
username@DESKTOP-XXXXXXX MINGW64 ~
$ which ls
/usr/bin/ls
$ ls /usr/bin/
'[.exe'* cp.exe* funzip.exe* grep.exe* mktemp.exe* msys-gpg-error-0.dll* msys-svn_repos-1-0.dll* psl-make-dafsa* ssh-keyscan.exe* unlink.exe*
addgnupghome* csplit.exe* gapplication.exe* groups.exe* mount.exe* msys-gssapi-3.dll* msys-svn_subr-1-0.dll* psl.exe* ssh-pageant.exe* unzip.exe*
applygnupgdefaults* cut.exe* gawk-5.0.0.exe* gsettings.exe* mpicalc.exe* msys-hcrypto-4.dll* msys-svn_swig_perl-1-0.dll* ptx.exe* ssh.exe* unzipsfx.exe*
arch.exe* cygcheck.exe* gawk.exe* gunzip* msgattrib.exe* msys-heimbase-1.dll* msys-svn_wc-1-0.dll* pwd.exe* sshd.exe* update-ca-trust*
astextplain* cygpath.exe* gdbus.exe* gzexe* msgcat.exe* msys-heimntlm-0.dll* msys-tasn1-6.dll* readlink.exe* ssp.exe* updatedb*
autopoint* cygwin-console-helper.exe* gencat.exe* gzip.exe* msgcmp.exe* msys-hogweed-6.dll* msys-ticw6.dll* realpath.exe* start* users.exe*
awk.exe* d2u.exe* getconf.exe* head.exe* msgcomm.exe* msys-hx509-5.dll* msys-unistring-2.dll* rebase.exe* stat.exe* vdir.exe*
b2sum.exe* dash.exe* getemojis* hmac256.exe* msgconv.exe* msys-iconv-2.dll* msys-uuid-1.dll* rebaseall* strace.exe* vendor_perl/
backup* date.exe* getfacl.exe* hostid.exe* msgen.exe* msys-idn2-0.dll* msys-wind-0.dll* recode-sr-latin.exe* stty.exe* vi*
base32.exe* dd.exe* getopt.exe* hostname.exe* msgexec.exe* msys-intl-8.dll* msys-z.dll* regtool.exe* sum.exe* view.exe*
base64.exe* df.exe* gettext.exe* iconv.exe* msgfilter.exe* msys-kafs-0.dll* mv.exe* reset.exe* sync.exe* vim.exe*
basename.exe* diff.exe* gettext.sh* id.exe* msgfmt.exe* msys-krb5-26.dll* nano.exe* restore* tabs.exe* vimdiff.exe*
basenc.exe* diff3.exe* gettextize* infocmp.exe* msggrep.exe* msys-ksba-8.dll* nettle-hash.exe* rm.exe* tac.exe* vimtutor*
bash.exe* dir.exe* gio-querymodules.exe* infotocap.exe* msginit.exe* msys-lz4-1.dll* nettle-lfib-stream.exe* rmdir.exe* tail.exe* watchgnupg.exe*
bashbug* dircolors.exe* git-flow* install.exe* msgmerge.exe* msys-magic-1.dll* nettle-pbkdf2.exe* rnano.exe* tar.exe* wc.exe*
bunzip2.exe* dirmngr-client.exe* git-flow-bugfix join.exe* msgunfmt.exe* msys-mpfr-6.dll* ngettext.exe* runcon.exe* tclsh.exe* which.exe*
bzcat.exe* dirmngr.exe* git-flow-config kbxutil.exe* msguniq.exe* msys-ncursesw6.dll* nice.exe* rview.exe* tclsh8.6.exe* who.exe*
bzcmp* dirname.exe* git-flow-feature kill.exe* msys-2.0.dll* msys-nettle-8.dll* nl.exe* rvim.exe* tee.exe* whoami.exe*
bzdiff* docx2txt* git-flow-hotfix ldd.exe* msys-apr-1-0.dll* msys-npth-0.dll* nohup.exe* scp.exe* test.exe* winpty-agent.exe*
bzegrep* docx2txt.pl* git-flow-init ldh.exe* msys-aprutil-1-0.dll* msys-p11-kit-0.dll* notepad* sdiff.exe* tic.exe* winpty-debugserver.exe*
bzfgrep* dos2unix.exe* git-flow-log less.exe* msys-asn1-8.dll* msys-pcre-1.dll* nproc.exe* sed.exe* tig.exe* winpty.dll*
bzgrep* du.exe* git-flow-release lessecho.exe* msys-assuan-0.dll* msys-perl5_32.dll* numfmt.exe* seq.exe* timeout.exe* winpty.exe*
bzip2.exe* dumpsexp.exe* git-flow-support lesskey.exe* msys-bz2-1.dll* msys-psl-5.dll* od.exe* setfacl.exe* toe.exe* wordpad*
bzip2recover.exe* echo.exe* git-flow-version libtcl8.6.dll* msys-cbor-0.8.dll* msys-readline8.dll* openssl.exe* setmetamode.exe* touch.exe* xargs.exe*
bzless* egrep* gitflow-common link.exe* msys-com_err-1.dll* msys-roken-18.dll* p11-kit.exe* sexp-conv.exe* tput.exe* xgettext.exe*
bzmore* env.exe* gitflow-shFlags ln.exe* msys-crypt-0.dll* msys-sasl2-3.dll* passwd.exe* sftp.exe* tr.exe* xxd.exe*
c_rehash* envsubst.exe* gkill.exe* locale.exe* msys-crypto-1.1.dll* msys-serf-1-0.dll* paste.exe* sh.exe* true.exe* yat2m.exe*
captoinfo.exe* ex.exe* glib-compile-schemas.exe* locate.exe* msys-edit-0.dll* msys-smartcols-1.dll* patch.exe* sha1sum.exe* truncate.exe* yes.exe*
cat.exe* expand.exe* gobject-query.exe* logname.exe* msys-expat-1.dll* msys-sqlite3-0.dll* pathchk.exe* sha224sum.exe* trust.exe* zcat*
chattr.exe* expr.exe* gpg-agent.exe* ls.exe* msys-ffi-7.dll* msys-ssl-1.1.dll* perl.exe* sha256sum.exe* tset.exe* zcmp*
chcon.exe* factor.exe* gpg-connect-agent.exe* lsattr.exe* msys-fido2-1.dll* msys-svn_client-1-0.dll* perl5.32.1.exe* sha384sum.exe* tsort.exe* zdiff*
chgrp.exe* false.exe* gpg-error.exe* mac2unix.exe* msys-gcc_s-seh-1.dll* msys-svn_delta-1-0.dll* pinentry-w32.exe* sha512sum.exe* tty.exe* zegrep*
chmod.exe* fgrep* gpg-wks-server.exe* md5sum.exe* msys-gcrypt-20.dll* msys-svn_diff-1-0.dll* pinentry.exe* shred.exe* tzset.exe* zfgrep*
chown.exe* fido2-assert.exe* gpg.exe* minidumper.exe* msys-gettextlib-0-19-8-1.dll* msys-svn_fs-1-0.dll* pinky.exe* shuf.exe* u2d.exe* zforce*
chroot.exe* fido2-cred.exe* gpgconf.exe* mintheme* msys-gettextsrc-0-19-8-1.dll* msys-svn_fs_fs-1-0.dll* pkcs1-conv.exe* sleep.exe* umount.exe* zgrep*
cksum.exe* fido2-token.exe* gpgparsemail.exe* mintty.exe* msys-gio-2.0-0.dll* msys-svn_fs_util-1-0.dll* pldd.exe* sort.exe* uname.exe* zipgrep*
clear.exe* file.exe* gpgscm.exe* mkdir.exe* msys-glib-2.0-0.dll* msys-svn_fs_x-1-0.dll* pluginviewer.exe* split.exe* uncompress* zipinfo.exe*
cmp.exe* find.exe* gpgsm.exe* mkfifo.exe* msys-gmodule-2.0-0.dll* msys-svn_ra-1-0.dll* pr.exe* ssh-add.exe* unexpand.exe* zless*
column.exe* findssl.sh* gpgsplit.exe* mkgroup.exe* msys-gmp-10.dll* msys-svn_ra_local-1-0.dll* printenv.exe* ssh-agent.exe* uniq.exe* zmore*
comm.exe* fmt.exe* gpgtar.exe* mknod.exe* msys-gnutls-30.dll* msys-svn_ra_serf-1-0.dll* printf.exe* ssh-copy-id* unix2dos.exe* znew*
core_perl/ fold.exe* gpgv.exe* mkpasswd.exe* msys-gobject-2.0-0.dll* msys-svn_ra_svn-1-0.dll* ps.exe* ssh-keygen.exe* unix2mac.exe*
Cygwin¶
Cygwin a large collection of GNU and Open Source tools which provide functionality similar to a Linux distribution on Windows.
cygwin是一个在windows平台上运行的linux模拟环境,相比git bash有更多的命令可以使用,还可以在改环境里面编译安装软件。
进入官网,找到对应版本进行下载 官网
- install from internet 从网络上安装
- 配置安装的目录以及使用的用户.默认即可
- 配置本地包目录,默认在C盘,可以选择其它盘
- 代理.默认即可
- use URL 使用镜像.可以选择阿里云镜像 https://mirrors.aliyu.com
- 安装linux工具跟库, view 根据分类查看目录,一般使用 category,比如可以在shell下选择
zsh安装
Copying skeleton files.
These files are for the users to personalise their cygwin experience.
They will never be overwritten nor automatically updated.
'./.bashrc' -> '/home/username//.bashrc'
'./.bash_profile' -> '/home/username//.bash_profile'
'./.inputrc' -> '/home/username//.inputrc'
'./.profile' -> '/home/username//.profile'
username@DESKTOP-XXXXXXX ~
$ ls
username@DESKTOP-XXXXXXX ~
$ pwd
/home/username
username@DESKTOP-XXXXXXX ~
$ cd C:
username@DESKTOP-XXXXXXX /cygdrive/c
$ pwd
/cygdrive/c
WSL¶
适用于Linux的Windows子系统(WSL)可让开发人员按原样运行 GNU/Linux 环境,包括大多数命令行工具、实用工具和应用程序,且不会产生传统虚拟机或双启动设置开销。
您可以:
- 在 Microsoft Store 中选择你偏好的 GNU/Linux 分发版。
- 运行常用的命令行软件工具(例如 grep、sed、awk)或其他 ELF-64 二进制文件。
- 运行 Bash shell 脚本和 GNU/Linux 命令行应用程序,包括:
- 工具:vim、emacs、tmux
- 语言:NodeJS、Javascript、Python、Ruby、C/C++、C# 与 F#、Rust、Go 等
- 服务:SSHD、MySQL、Apache、lighttpd、MongoDB、PostgreSQL。
- 使用自己的 GNU/Linux 分发包管理器安装其他软件。
- 使用类似于 Unix 的命令行 shell 调用 Windows 应用程序。
- 在 Windows 上调用 GNU/Linux 应用程序。
- 运行直接集成到 Windows 桌面的 GNU/Linux 图形应用程序
- 将 GPU 加速用于机器学习、数据科学场景等
WSL1¶
启用WSL¶
Windows搜索框搜索 启用,打开 启用或关闭 Windows 功能,勾选适用于Linux的 Windows 子系统,下载完成之后重启。
安装linux系统¶
- Windows搜索框搜索
store,打开Microsoft Store - 搜索linux,
- 从搜过结果中选择合适的版本安装
WSL2¶
WSL 2 架构在几个方面优于 WSL 1
| 功能 | WSL 1 | WSL 2 |
|---|---|---|
| Windows 和 Linux 之间的集成 | ✅ | ✅ |
| 启动时间短 | ✅ | ✅ |
| 与传统虚拟机相比,占用的资源量少 | ✅ | ✅ |
| 可以与当前版本的 VMware 和 VirtualBox 一起运行 | ✅ | ✅ |
| 托管 VM | ❌ | ✅ |
| 完整的 Linux 内核 | ❌ | ✅ |
| 完全的系统调用兼容性 | ❌ | ✅ |
| 跨 OS 文件系统的性能 | ✅ | ❌ |
windows版本要求:Windows 10 版本 2004 及更高版本(内部版本 19041 及更高版本)或 Windows 11
WSL2基于Hyper-V虚拟技术,需要提前开启虚拟化支持。
Windows搜索框搜索 启用,打开 启用或关闭 Windows 功能,勾选虚拟机平台,下载完成之后重启。
- 在
Windows 图标右键,选择Windows PowerShell(管理员)(A)启动powershell终端 - 在终端窗口中输入:wsl --install 即可自动执行。
PS C:\users\username> WSL --install 正在安装: 虚拟机平台 已安装 虚拟机平台。 正在安装:适用于 Linux 的 windows 子系统 已安装 适用于 Linux的 windows 子系统。 正在下载:WSL 内核 正在安装: WSL 内核 已安装 WSL 内核。 正在下载:GUI 应用支持 正在安装:GUI 应用支持 已安装 GUI 应用支持。 - 在完成 已安装 GUI 应用支持 后, 按住CTRL+C取消操作后重启。
- 开机后调出终端,输入执行
wsl --set-default-version 2将 WSL 默认版本调整为 WSL2。 - 在 Microsoft Store 中找到对应发行版进行安装即可;也可通过命令行安装。
-
命令行安装方法:
-
wsl -l -o可查看可安装的发行版,记录发行版名称如ubuntu-20.04PS C:\users\username> wsl -l -o 以下是可安装的有效分发的列表。 请使用“wsl --install -d <分发>”安装。 NAME FRIENDLY NAME Ubuntu Ubuntu Debian Debian GNU/Linux kali-linux Kali Linux Rolling openSUSE-42 openSUSE Leap 42 SLES-12 SUSE Linux Enterprise Server v12 Ubuntu-16.04 Ubuntu 16.04 LTS Ubuntu-18.04 Ubuntu 18.04 LTS Ubuntu-20.04 Ubuntu 20.04 LTS -
wsl --install --d ubuntu-20.04安装ubuntu20.04 - 安装完毕后,
wsl -l -v可查看安装的发行版的WSL版本。 - 在开始菜单中打开Ubuntu
-
更换安装目录¶
上述安装步骤,默认将Ubuntu安装在C盘中,C盘空间不够可能会影响后续使用,因此我们可以将其安装在更大的盘中,如D盘。
- 在D盘中创建文件夹WSL,然后在WSL文件夹中创建文件夹Ubuntu;
-
下载相关文件,如 https://aka.ms/wslubuntu2004,建议使用迅雷下载
其它linux发行版本的下载链接如下所示,参考 WSL手动安装
https://aka.ms/wslubuntu https://aka.ms/wslubuntu2204 https://aka.ms/wslubuntu2004 https://aka.ms/wslubuntu2004arm https://aka.ms/wsl-ubuntu-1804 https://aka.ms/wsl-ubuntu-1804-arm https://aka.ms/wsl-ubuntu-1604 https://aka.ms/wsl-debian-gnulinux https://aka.ms/wsl-kali-linux-new https://aka.ms/wsl-sles-12 https://aka.ms/wsl-SUSELinuxEnterpriseServer15SP2 https://aka.ms/wsl-SUSELinuxEnterpriseServer15SP3 https://aka.ms/wsl-opensuse-tumbleweed https://aka.ms/wsl-opensuseleap15-3 https://aka.ms/wsl-opensuseleap15-2 https://aka.ms/wsl-oraclelinux-8-5 https://aka.ms/wsl-oraclelinux-7-9 -
将下载的
wslubuntu2004放入新建的文件夹D:\WSL\Ubuntu\中,并重命名为wslubuntu2004.zip - 将
wslubuntu2004.zip解压,将解压出来的文件中的Ubuntu_2004.2021.825.0_x64.appx重命名为Ubuntu_2004.2021.825.0_x64.zip - 将
Ubuntu_2004.2021.825.0_x64.zip解压,双击ubuntu2004.exe即可打开Ubuntu如果出现如下报错Welcome to Ubuntu 20.04 LTS (GNU/Linux 5.10.16.3-microsoft-standard-WSL2 x86_64) * Documentation: https://help.ubuntu.com * Management: https://landscape.canonical.com * Support: https://ubuntu.com/advantage System information as of Tue Dec 6 11:25:37 CST 2022 System load: 0.0 Processes: 8 Usage of /: 0.4% of 250.98GB Users logged in: 0 Memory usage: 0% IPv4 address for eth0: 172.22.85.201 Swap usage: 0% 0 updates can be installed immediately. 0 of these updates are security updates. The list of available updates is more than a week old. To check for new updates run: sudo apt update This message is shown once once a day. To disable it please create the /root/.hushlogin file. root@DESKTOP-XXXXX:~#下载这个软件 https://wslstorestorage.blob.core.windows.net/wslblob/wsl_update_x64.msi 安装,重启系统再双击Installing, this may take a few minutes... WslRegisterDistribution failed with error: 0x800701bc Error: 0x800701bc WSL 2 ?????????????????? https://aka.ms/wsl2kernel Press any key to continue...ubuntu2004.exe打开Ubuntu
更换源¶
以Ubuntu 20.04为例,不同版本参考
# 更新证书
apt-get install --only-upgrade ca-certificates
mv /etc/apt/sources.list /etc/apt/sources.list.back
sudo sed -i "s@http://.*archive.ubuntu.com@https://mirrors.tuna.tsinghua.edu.cn@g" /etc/apt/sources.list
sudo sed -i "s@http://.*security.ubuntu.com@https://mirrors.tuna.tsinghua.edu.cn@g" /etc/apt/sources.list
sudo apt-get update
sudo apt-get upgrade
虚拟机¶
使用virtualbox或VMware Workstation Player安装linux虚拟机,可以体验完整的linux从安装到完整系统的使用。
Linux Images 中有各种Linux发行版本的virtualbox和VMware,可以直接下载使用,避免自己从头安装。
云服务器¶
前面几种方式都是在自己本地电脑上模拟Linux环境,新手用户也可以买一台云服务器,拥有一台属于自己的服务器,用于练习或者数据分析。
目前各个云服务器厂商为了做推广,新用户或者学生用户,会有比较优惠的活动,新手用户可以尝试轻量应用服务器,一般配置较低,价格较低,适合练习Linux、搭建博客等。
国内常用的云服务器厂商:
国内下载github上的软件或国外的数据可能速度会比较慢,可以尝试使用海外的云服务器。
vultr 支持Alipay支付,支持按量使用付费,使用结束后销毁服务器就不会再计费,服务器IP国内不通时也可以免费更换IP
Ended: Linux
本站总访问量 次